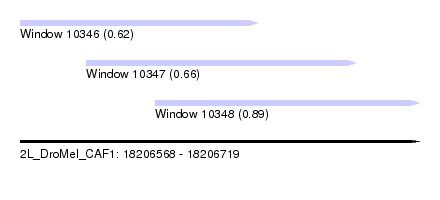
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,206,568 – 18,206,719 |
| Length | 151 |
| Max. P | 0.893374 |

| Location | 18,206,568 – 18,206,658 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 83.05 |
| Mean single sequence MFE | -38.70 |
| Consensus MFE | -26.82 |
| Energy contribution | -26.85 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.623943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18206568 90 + 22407834 CCGACACCACCGUU------CAGGCCACCCAGACCGCCCAAACCGCCGGCAC------CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....(((((((...------..((((((.......(((...(((((.(((((------......))))))))))))).))))))(((....))).))))))) ( -38.70) >DroPse_CAF1 53383 93 + 1 CCCAGGCCUC---C------UAAGCCUCCCACACCGCCGCCACUGCUGGCACCUCCGCCUCCCAGGGCUGUGGUGGCCGUGGCCGUGCUGCUGCUCGUGGUG ...((((...---.------...)))).(((((((((.(((.(((..(((......)))...)))))).)))))(((....)))(.((....)).))))).. ( -38.20) >DroSec_CAF1 49423 90 + 1 CCGGCACCACCGUU------CAGGCCACCAAGACCGCCCAAACCGCCGGCAC------CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....(((((((...------..((((((((.....((.......))((((((------......))))))))))))))((((.....))))....))))))) ( -39.50) >DroSim_CAF1 50040 90 + 1 CCGGCACCACCGUU------CAGGCCACCAAGGCCGCCCAAACCGCCGGCAC------CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....(((((((...------..((((((...(((((((........((((((------......)))))).)))))))))))))(((....))).))))))) ( -43.70) >DroEre_CAF1 49703 96 + 1 CCGGCACCACCAUUCAAAUUCAAGCCAACCGCACCGCCCAAACCACCGGCAC------CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....((((((((...................(((.(((...(((((.(((((------......))))))))))))).)))((.(.....).)))))))))) ( -34.80) >DroYak_CAF1 49574 90 + 1 CAAGCACCACCGUU------CAAGCCAGCCACACCGCCCAAACCACCGGCAC------CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....(((((((((.------..((...(((((...(((...(((((.(((((------......))))))))))))).)))))....))...)).))))))) ( -37.30) >consensus CCGGCACCACCGUU______CAAGCCACCCACACCGCCCAAACCGCCGGCAC______CACCAAGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUG ....(((((((.....................((.(((...(((((.(((((............))))))))))))).))(((.(.....).)))))))))) (-26.82 = -26.85 + 0.03)


| Location | 18,206,593 – 18,206,695 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 82.78 |
| Mean single sequence MFE | -47.47 |
| Consensus MFE | -38.52 |
| Energy contribution | -37.22 |
| Covariance contribution | -1.30 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.662775 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18206593 102 + 22407834 GACCGCCC--AAACCGCCGGCAC------CACCA---AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUGAGGUUGCAGC ........--.....((((((((------(((((---...((.((((((((....)))..))))).)))))))))))))))......(((((.(((........))).))))) ( -49.10) >DroPse_CAF1 53405 108 + 1 CACCGCCG--CCACUGCUGGCACCUCCGCCUCCC---AGGGCUGUGGUGGCCGUGGCCGUGCUGCUGCUCGUGGUGCUUGCCGCACUGUUGCCGCCACUGCUCAGGUUGCAGC .(((((.(--((.(((..(((......)))...)---))))).)))))(((.(..((.((((.((.((.......))..)).)))).))..).))).((((.......)))). ( -48.70) >DroSim_CAF1 50065 102 + 1 GGCCGCCC--AAACCGCCGGCAC------CACCA---AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGC (((.((..--.....((((((((------(((((---...((.((((((((....)))..))))).)))))))))))))))......)).)))...(((((.......))))) ( -52.32) >DroWil_CAF1 73548 107 + 1 UGCUGUUAAAACACCACCAGCAC------CACCAUUCAGAGCUGUGGUUGCUGUGGCCGUACUGCUGCUAGUGGUGCUGGCUGCACUGUUGCCACCGCUGCUUAAAUUGCAGC (((((............))))).------..............(((((.((((..(((((((..(.....)..)))).)))..))..)).))))).(((((.......))))) ( -37.80) >DroYak_CAF1 49599 102 + 1 CACCGCCC--AAACCACCGGCAC------CACCA---AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGC ....(((.--..(((((.(((((------.....---.))))))))))))).(((((((((((((.....))))))).))))))...(((((.(((........))).))))) ( -48.20) >DroPer_CAF1 53342 108 + 1 CACCGCCG--CCACUGCUGGCACCUCCGCCUCCC---AGGGCUGUGGUGGCCGUGGCCGUGCUGCUGCUCGUGGUGCUUGCCGCACUGUUGCCGCCACUGCUCAGGUUGCAGC .(((((.(--((.(((..(((......)))...)---))))).)))))(((.(..((.((((.((.((.......))..)).)))).))..).))).((((.......)))). ( -48.70) >consensus CACCGCCC__AAACCGCCGGCAC______CACCA___AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGC .((((((...........)))...................((.((((((((.(((((((((((((.....))))))).))))))......)))))))).))...)))...... (-38.52 = -37.22 + -1.30)


| Location | 18,206,619 – 18,206,719 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 90.03 |
| Mean single sequence MFE | -49.37 |
| Consensus MFE | -41.64 |
| Energy contribution | -41.23 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.893374 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18206619 100 + 22407834 -AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUGAGGUUGCAGCCGUGAUCCGCCAGGGCGUGCAGCA -...((.((((((((.(((((((((((((.....))))))).))))))......)))))))).))....(((((((((.((......)).))).)))))). ( -54.00) >DroPse_CAF1 53437 100 + 1 -AGGGCUGUGGUGGCCGUGGCCGUGCUGCUGCUCGUGGUGCUUGCCGCACUGUUGCCGCCACUGCUCAGGUUGCAGCCGUGAUCCGCCAGCGAGUGCAGCA -..(((..(((...)))..))).((((((.((((((((((.......(((.(((((.(((........))).))))).)))...))))).))))))))))) ( -47.70) >DroEre_CAF1 49760 100 + 1 -AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGCCGUGAUCCGCCAAGGCGUGCAGCA -.(.((.((((((((.(((((((((((((.....))))))).))))))......)))))))).)).)..(((((((((.((......)).))).)))))). ( -52.90) >DroWil_CAF1 73578 101 + 1 CAGAGCUGUGGUUGCUGUGGCCGUACUGCUGCUAGUGGUGCUGGCUGCACUGUUGCCACCGCUGCUUAAAUUGCAGCCAUGAUCAGCUAGUGAAUGCAGCA ....(((((..(..(((..(((((((..(.....)..)))).)))..)...((((.((..(((((.......)))))..))..)))).))..)..))))). ( -41.00) >DroYak_CAF1 49625 100 + 1 -AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGCCGUGAUCCGCCAAGGCGUGCAGCA -.(.((.((((((((.(((((((((((((.....))))))).))))))......)))))))).)).)..(((((((((.((......)).))).)))))). ( -52.90) >DroPer_CAF1 53374 100 + 1 -AGGGCUGUGGUGGCCGUGGCCGUGCUGCUGCUCGUGGUGCUUGCCGCACUGUUGCCGCCACUGCUCAGGUUGCAGCCGUGAUCCGCCAGCGAGUGCAGCA -..(((..(((...)))..))).((((((.((((((((((.......(((.(((((.(((........))).))))).)))...))))).))))))))))) ( -47.70) >consensus _AGUGCUGUGGUGGCCGUGGCCGUACUACUGCUGGUGGUGCUGGCCGCACUGUUGCCACCGCUGCUCAGGUUGCAGCCGUGAUCCGCCAGGGAGUGCAGCA ....((((((((((..(((((((((((((.....))))))).))))))........(((.(((((.......))))).)))..))))).......))))). (-41.64 = -41.23 + -0.41)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:12:17 2006