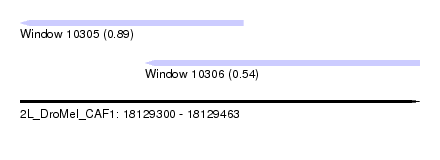
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,129,300 – 18,129,463 |
| Length | 163 |
| Max. P | 0.891070 |

| Location | 18,129,300 – 18,129,391 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 91 |
| Reading direction | reverse |
| Mean pairwise identity | 85.57 |
| Mean single sequence MFE | -46.50 |
| Consensus MFE | -38.79 |
| Energy contribution | -38.47 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.96 |
| SVM RNA-class probability | 0.891070 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
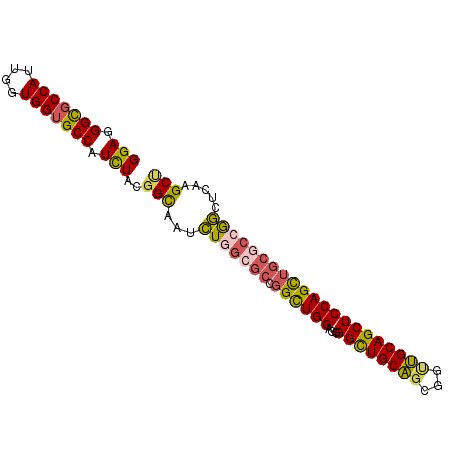
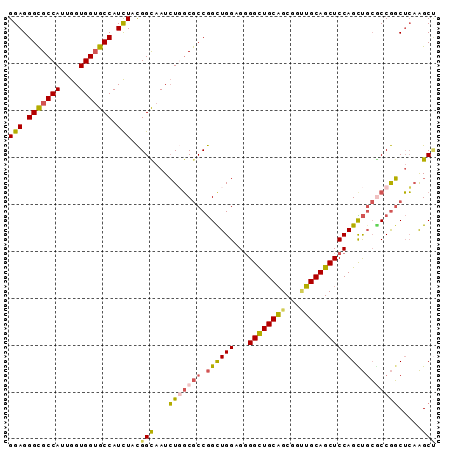
>2L_DroMel_CAF1 18129300 91 - 22407834 GGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCUCAAGCU (((.(((((((....))))))).))).(((....)))(.((((((.(.(((((((((((...))))))))))).)...)))))).)..... ( -50.80) >DroSec_CAF1 19230 91 - 1 GGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGUUGCAGCUCCAGCUGCGCCGGCUCAAGCG (((.(((((((....))))))).)))...((...(((((((.((((((...((((((((...))))))))))))))))))))).....)). ( -48.90) >DroSim_CAF1 19992 91 - 1 GGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGUGGGGCUGCAGCGGCUGCAGCUCCAGCUGCGCCGGCUCAAGCU (((.(((((((....))))))).)))..(((...(((((((.((((((...((((((((...))))))))))))))))))))).....))) ( -51.10) >DroEre_CAF1 10553 91 - 1 GGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGUUGGAGGGGUUGCAGCCGUUGCAGCUCCAGCUGCCCCGGCUCAAGCU (((.(((((((....))))))).))).((((........)))).......((.(((.(((((..(((((....)))))..)))))))).)) ( -42.60) >DroYak_CAF1 33720 91 - 1 GGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUUGCGCCGGUUGGAGGGGUUGCAGCUGUUGCAGCUCCAGUUGCACCGGCUCAAGCU (((.(((((((....))))))).)))........((((.((((((....((((((((((...))))))))))......)))))).)))).. ( -42.60) >DroPer_CAF1 21617 88 - 1 GGAGGGUGCCAUUGGUGGGGCCAUUUAUGGCAAUAUCCCACCAGUUGGAGCGGCUGCUGC---AGCAGCUCCAGUCCAGCCAUCGGCGGCC ...((.((((..(((((((((((....)))).....)))))))(((((((.((((((...---.)))))).)..))))))....)))).)) ( -43.00) >consensus GGAGGGCGCCAUUGGUGGUGCCAUCUACGGCAAUCUGGCGCCGGCUGGAGGGGCUGCAGCGGUUGCAGCUCCAGCUGCGCCGGCUCAAGCU (((.(((((((....))))))).)))..(((...(((((((.((((((...((((((((...))))))))))))))))))))).....))) (-38.79 = -38.47 + -0.33)

| Location | 18,129,351 – 18,129,463 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.07 |
| Mean single sequence MFE | -41.28 |
| Consensus MFE | -26.44 |
| Energy contribution | -24.95 |
| Covariance contribution | -1.49 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.536113 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
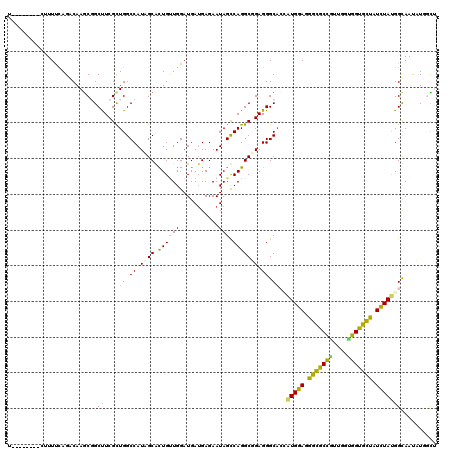
>2L_DroMel_CAF1 18129351 112 - 22407834 U--------CUUUUCAGCCAAGCUGCUUCGUUGGCCAUCGCACUGCUGGAUGAUGAGAAUAGCCAGGCGGAGGGCACGAUGGAGGGCGCCAUUGGUGGUGCCAUCUACGGUAAUCUGGCG .--------.......((((.(((((((((((((((.((((.((((((...........))).)))))))..))).))))))))(((((((....))))))).....))))....)))). ( -44.70) >DroVir_CAF1 21902 112 - 1 U--------GUUUCUAGCCAAGCGGCCUCGCUGGCCAUAGCGCUGUUGGAUGAUGAGAAUAGCCAGGCUGAGGGCACCAUGGAGGGCGCCGUCGGUGGUGCUGUUUAUGGCAAUAUAAUA (--------(((..(((((....((((.....))))...(.((((((..........))))))).)))))..))))(((((((.(((((((....))))))).))))))).......... ( -44.70) >DroGri_CAF1 22157 112 - 1 U--------UGUUUUAGGCAAGCGGCUUCGCUGGCCAUAGCGCUGUUGGAUGAUGAGAAUAGCCAGGCGGAGGGCAGCAUGGAGGGCGCCGUUGGUGGUGCUGUCUAUGGCAAUAUUGUU (--------((((((.(((.((((....)))).)))....((((..(((...((....))..))))))).)))))))((((((.(((((((....))))))).))))))........... ( -40.40) >DroWil_CAF1 45965 120 - 1 UUUUCUAUGCUUUUUAGACAAGCUGCUUCGCUGGCCAUUGCAUUGAUGGAUGAAGAGAAUAGUCAGGCUGAGGGCUCAAUGGAGGGUGCUGUGGGUGGUGUUAUUUAUGGUAAUAUUGCU ...((((.......))))((((((.(((((.(((((...((.(((((..((.......)).)))))))....))).)).))))).).))).))((..(((((((.....)))))))..)) ( -30.20) >DroMoj_CAF1 25187 109 - 1 -----------UUUCAGGCAAACGGCCUCGCUGGCCAUAGCGCUGUUGGAUGAUGAGAAUAGCCAGGCUGAGGGCAGCAUGGAGGGCGCUGUCGGCGGUGCUGUUUAUGGCAACAUCAUU -----------........(((((((.((((((((...((((((.((((...((....))..))..((((....))))...)).)))))))))))))).)))))))(((....))).... ( -40.90) >DroAna_CAF1 19380 112 - 1 C--------UUUUCCAGACAAGCGGCUUCGCUGGCCAUCGCACUGUUGGAUGAUGAGAAUAGUCAGGCGGAGGGCACUGUGGAGGGCGCCAUUGGCGGCGCCAUCUACGGUAACCUGGCU .--------....((((....((..(((((((((((((((..(....)..)))))......)))).)))))).))((((((((.((((((......)))))).))))))))...)))).. ( -46.80) >consensus U________CUUUUCAGACAAGCGGCUUCGCUGGCCAUAGCACUGUUGGAUGAUGAGAAUAGCCAGGCGGAGGGCACCAUGGAGGGCGCCGUUGGUGGUGCUAUCUAUGGCAAUAUGGCU ........................(((....((.((.((((.((((((...........))).))))))).)).))..(((((.(((((((....))))))).))))))))......... (-26.44 = -24.95 + -1.49)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:11:36 2006