
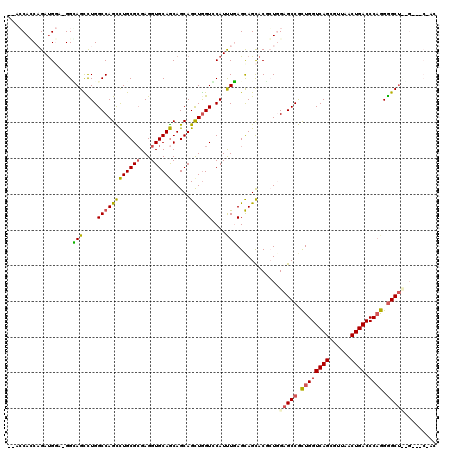
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,065,224 – 18,065,332 |
| Length | 108 |
| Max. P | 0.600730 |

| Location | 18,065,224 – 18,065,332 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.81 |
| Mean single sequence MFE | -49.70 |
| Consensus MFE | -31.44 |
| Energy contribution | -30.83 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.566123 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
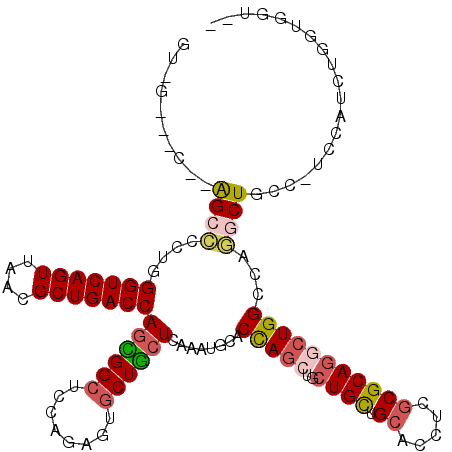
>2L_DroMel_CAF1 18065224 108 + 22407834 --ACCACCAGAUGGACGGCAGCUUGGCCAGCCUGCGGGAGGUGCAGCAGCAGCUGGUCCAUCUGAGCAGCACGCUAGAGCCGCUGGUCAGCGUUAACUGACCCAGGGGCC---------- --.....((((((((((((......))).((((.....)))).((((....)))))))))))))(((.....)))...(((.((((((((......)))).)))).))).---------- ( -48.60) >DroPse_CAF1 364843 118 + 1 --ACCACCCGAUGGAGGGGAGCCUGGCCAGCCUGCGCGAGGUGCAGCAGCAGCUGGUCCAUUUCAGCAGCACUGUGGAGCCGCUGGUCAGCGUUAACUGACCCUGGGGCUCCGUCUCGAC --....(((......)))..((..((....)).)).(((((.((((..((.(((((......))))).)).))))((((((.(.((((((......))))))..).)))))).))))).. ( -51.20) >DroGri_CAF1 363226 115 + 1 --AGCAGCAGAUG---GGUAGUUUGGCCAGUCUGCGCGAGGUGCAGCAGCAGUUGGUGCAUUUGAGCAGUACGCUGGAGCCACUGGUCAGCGUUAACUGACCCAGGAGCUUAGCACCACC --.((.((((((.---(((......))).))))))))..(((((..((((.((.(.(((......))).)))))))((((..((((((((......)))).))))..)))).)))))... ( -50.80) >DroWil_CAF1 345057 110 + 1 CACCCAUCAGAUGGAUGGCAGUCUGGCCAGUUUGCGCGAGGUGCAACAGCAGCUAGUGCAUCUGAGUAGCACUCUGGAGCCAUUGGUCAGCGUUAACUGACCCAAGGGCU---------- ...(((((.....)))))(((..(((((((..(((((.((.(((....))).)).))))).))).))..))..))).((((...((((((......))))))....))))---------- ( -41.00) >DroMoj_CAF1 411042 115 + 1 --AGCAGCAGACG---GGCAGCCUGGCCAGCCUGCGCGAGGUGCAGCAGCAGCUGGUGCAUUUGAGCAGCACGCUCGAGCCGCUGGUCAGCGUUAACUGACCCAGGGGCUUGGCAGCAAC --.((.((((.(.---(((......))).).))))((.(((((((.(((...))).)))))))..)).))..((.((((((.((((((((......)))).)))).))))))))...... ( -53.70) >DroPer_CAF1 369023 118 + 1 --ACCACCCGAUGGAGGGGAGCCUGGCCAGCCUGCGCGAGGUGCAGCAGCAGCUGGUCCAUUUCAGCAGCACUGUGGAGCCGCUGGUCAGCGUUAACUGACCCAGGGGCUCCGUCUCGAC --....(((......)))..((..((....)).)).(((((.((((..((.(((((......))))).)).))))((((((.((((((((......)))).)))).)))))).))))).. ( -52.90) >consensus __ACCACCAGAUGGA_GGCAGCCUGGCCAGCCUGCGCGAGGUGCAGCAGCAGCUGGUCCAUUUGAGCAGCACGCUGGAGCCGCUGGUCAGCGUUAACUGACCCAGGGGCU__G___C_AC ..................(((....((((((((((((...)))))).....))))))....)))............(((((.((((((((......)))).)))).)))))......... (-31.44 = -30.83 + -0.61)


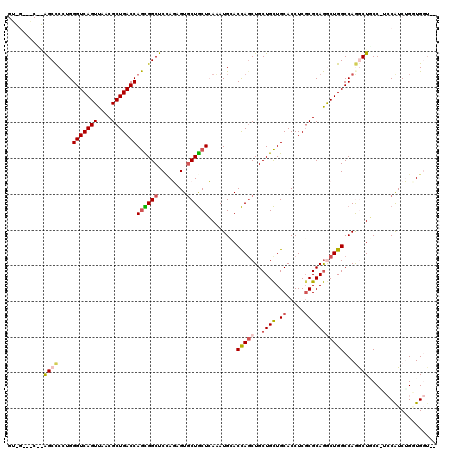
| Location | 18,065,224 – 18,065,332 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.81 |
| Mean single sequence MFE | -48.62 |
| Consensus MFE | -29.43 |
| Energy contribution | -30.10 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.600730 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18065224 108 - 22407834 ----------GGCCCCUGGGUCAGUUAACGCUGACCAGCGGCUCUAGCGUGCUGCUCAGAUGGACCAGCUGCUGCUGCACCUCCCGCAGGCUGGCCAAGCUGCCGUCCAUCUGGUGGU-- ----------((((.((((.(((((....))))))))).)))).......((..(.((((((((((((((((((((((.......))).)).)))..)))))..))))))))))..))-- ( -50.70) >DroPse_CAF1 364843 118 - 1 GUCGAGACGGAGCCCCAGGGUCAGUUAACGCUGACCAGCGGCUCCACAGUGCUGCUGAAAUGGACCAGCUGCUGCUGCACCUCGCGCAGGCUGGCCAGGCUCCCCUCCAUCGGGUGGU-- ........((((((....(((((((....)))))))((((((........))))))....(((.(((((..((((.((.....))))))))))))))))))))((.((....)).)).-- ( -54.50) >DroGri_CAF1 363226 115 - 1 GGUGGUGCUAAGCUCCUGGGUCAGUUAACGCUGACCAGUGGCUCCAGCGUACUGCUCAAAUGCACCAACUGCUGCUGCACCUCGCGCAGACUGGCCAAACUACC---CAUCUGCUGCU-- .(.(((((..(((..((((.(((((....)))))))))..))).((((((..(((......)))...)).))))..))))).)(((((((.(((.........)---))))))).)).-- ( -44.10) >DroWil_CAF1 345057 110 - 1 ----------AGCCCUUGGGUCAGUUAACGCUGACCAAUGGCUCCAGAGUGCUACUCAGAUGCACUAGCUGCUGUUGCACCUCGCGCAAACUGGCCAGACUGCCAUCCAUCUGAUGGGUG ----------.((((...(((((((....)))))))..((((........)))).((((((((.(.((.(((....))).)).).))....((((......))))...)))))).)))). ( -37.70) >DroMoj_CAF1 411042 115 - 1 GUUGCUGCCAAGCCCCUGGGUCAGUUAACGCUGACCAGCGGCUCGAGCGUGCUGCUCAAAUGCACCAGCUGCUGCUGCACCUCGCGCAGGCUGGCCAGGCUGCC---CGUCUGCUGCU-- ...((.((..((((.((((.(((((....))))))))).)))).(((.(((((((......)))..(((....))))))))))))((((((.(((......)))---.)))))).)).-- ( -50.20) >DroPer_CAF1 369023 118 - 1 GUCGAGACGGAGCCCCUGGGUCAGUUAACGCUGACCAGCGGCUCCACAGUGCUGCUGAAAUGGACCAGCUGCUGCUGCACCUCGCGCAGGCUGGCCAGGCUCCCCUCCAUCGGGUGGU-- ........((((((....(((((((....)))))))((((((........))))))....(((.(((((..((((.((.....))))))))))))))))))))((.((....)).)).-- ( -54.50) >consensus GU_G___C__AGCCCCUGGGUCAGUUAACGCUGACCAGCGGCUCCAGAGUGCUGCUCAAAUGCACCAGCUGCUGCUGCACCUCGCGCAGGCUGGCCAGGCUGCC_UCCAUCUGGUGGU__ ..........((((....(((((((....)))))))((((((........))))))........(((((..((((.((.....)))))))))))...))))................... (-29.43 = -30.10 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:38 2006