
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 18,018,400 – 18,018,550 |
| Length | 150 |
| Max. P | 0.999850 |

| Location | 18,018,400 – 18,018,512 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 94.15 |
| Mean single sequence MFE | -44.80 |
| Consensus MFE | -40.30 |
| Energy contribution | -40.06 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.90 |
| SVM decision value | 4.25 |
| SVM RNA-class probability | 0.999850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18018400 112 + 22407834 GUAUAUGAGGGGCUGUCAGGCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGCCCAUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCA ((((.....((((.((((...((((.....((((....)))).......))))..)))).))))(((((((((((.((((((....)))))).))))...)))))))))--)). ( -46.70) >DroSec_CAF1 234132 112 + 1 GUAUAUGAGGGGCUGUCAGCCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGCCCAUGCACACACUAGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCA ((((((...((((.((((...((((.....((((....)))).......))))..)))).))))..(((((((((.((((((....)))))).))))...)))))))))--)). ( -44.80) >DroSim_CAF1 233866 112 + 1 GUAUAUGAGGGGCUGUCAGCCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGCCCAUGCACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCA ((((((...((((.((((...((((.....((((....)))).......))))..)))).))))..(((((((((.((((((....)))))).))))...)))))))))--)). ( -46.90) >DroEre_CAF1 226780 113 + 1 GUAUAUGAG-GGCUGUCGUCCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGUCCAGACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUAUGUGCA (((((((.(-(((.(((((..((((.....((((....)))).......)))).))))).))))..(((((((((.((((((....)))))).))))...))))).))))))). ( -44.70) >DroYak_CAF1 243522 109 + 1 GUAUAUGAGGGGCUGUCAUCCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGUCCGUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU----- .........((((.(((((..((((.....((((....)))).......)))).))))).))))..((((((((..((((.(((.......)))))))...))))))))----- ( -40.90) >consensus GUAUAUGAGGGGCUGUCAGCCAAGGAGUCUGCCCAACUGGGCCACAAAACCUUUAUGGCUGCCCAUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU__GCA .........((((.((((...((((.....((((....)))).......))))..)))).))))..((((((((..((((.(((.......)))))))...))))))))..... (-40.30 = -40.06 + -0.24)

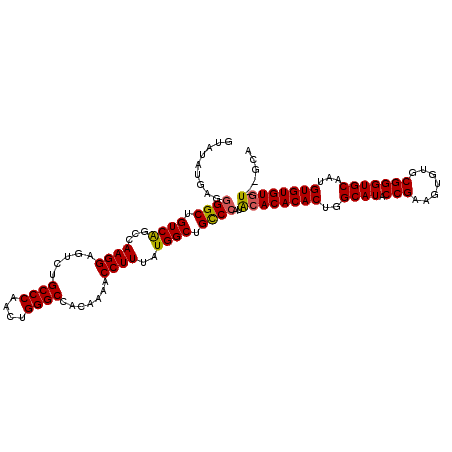
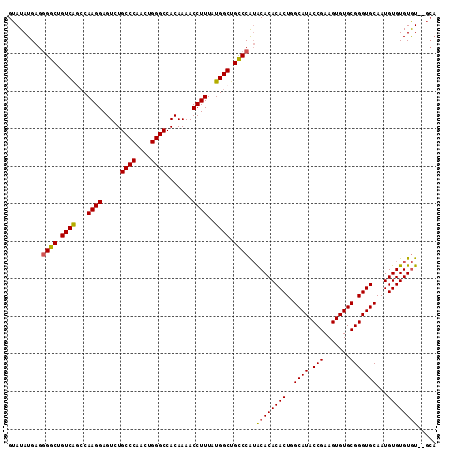
| Location | 18,018,400 – 18,018,512 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 94.15 |
| Mean single sequence MFE | -41.24 |
| Consensus MFE | -33.60 |
| Energy contribution | -34.24 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.81 |
| SVM decision value | 2.49 |
| SVM RNA-class probability | 0.994543 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18018400 112 - 22407834 UGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUAUGGGCAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGCCUGACAGCCCCUCAUAUAC ...--..((((((((((.....(((.((....)).)))))))))))))..((((.(((((((.....)))))))...((..(((((.....)))))..)).))))......... ( -46.50) >DroSec_CAF1 234132 112 - 1 UGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCUAGUGUGUGCAUGGGCAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGGCUGACAGCCCCUCAUAUAC (((--((((((.....(....)(((.((....)).)))..))))))))).(((((((((...(((..((...((((....)))).))..))))))))....))))......... ( -43.60) >DroSim_CAF1 233866 112 - 1 UGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGCAUGGGCAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGGCUGACAGCCCCUCAUAUAC (((--((((((....(((....))).((....))......))))))))).(((((((((...(((..((...((((....)))).))..))))))))....))))......... ( -43.60) >DroEre_CAF1 226780 113 - 1 UGCACAUACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUCUGGACAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGGACGACAGCC-CUCAUAUAC .......((((((((((.....(((.((....)).)))))))))))))((((...(((((((.....)))))))))))..((((.(.(((....)).).).)))-)........ ( -37.10) >DroYak_CAF1 243522 109 - 1 -----ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUACGGACAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGGAUGACAGCCCCUCAUAUAC -----..((((((((((.....(((.((....)).)))))))))))))..((...(((((((.....))))))))).(..((((.(.(((....)).).).))))..)...... ( -35.40) >consensus UGC__ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUAUGGGCAGCCAUAAAGGUUUUGUGGCCCAGUUGGGCAGACUCCUUGGCUGACAGCCCCUCAUAUAC ........(((((((((.....(((.((....)).))))))))))))...((.((((((...(((..((...((((....)))).))..)))))))))....)).......... (-33.60 = -34.24 + 0.64)



| Location | 18,018,440 – 18,018,550 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 85.38 |
| Mean single sequence MFE | -46.92 |
| Consensus MFE | -35.71 |
| Energy contribution | -38.06 |
| Covariance contribution | 2.35 |
| Combinations/Pair | 1.08 |
| Mean z-score | -4.08 |
| Structure conservation index | 0.76 |
| SVM decision value | 3.20 |
| SVM RNA-class probability | 0.998716 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18018440 110 + 22407834 GCCACAAAACCUUUAUGGCUGCCCAUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCAUGUGGCUCUGCGUGUGUAUGGGUGCUUGUGGCUCUCAA (((((((..((.....))..(((((((((((((((.((((((....)))))).)))((.((((((....--)))))).)).....))))))))))))..)))))))...... ( -50.80) >DroSec_CAF1 234172 110 + 1 GCCACAAAACCUUUAUGGCUGCCCAUGCACACACUAGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCAUGUGGCUCUGCGUGUGUAUGGGUGCUUGUGGCUCUCAA (((((((.((((.((((((.((((((((((((((..((((.(((.......))))))).....))))))--))))).)))...)).))))...))))..)))))))...... ( -51.00) >DroSim_CAF1 233906 110 + 1 GCCACAAAACCUUUAUGGCUGCCCAUGCACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU--GCAUGUGGCUCUGCGUGUGUAUGGGUGCUUGUGGCUCUCAA (((((((.((((.((((((.((((((((((((((..((((.(((.......))))))).....))))))--))))).)))...)).))))...))))..)))))))...... ( -52.10) >DroEre_CAF1 226819 112 + 1 GCCACAAAACCUUUAUGGCUGUCCAGACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUAUGUGCAUGUGGCUCCGGGUGUGUAUGGUUGCCUGUGGCUCUUAA ((((((..........(((...(((.((((((.((((....(((....(((((.(((((.....))))).))))).)))..)))))))))).)))..)))))))))...... ( -44.00) >DroYak_CAF1 243562 84 + 1 GCCACAAAACCUUUAUGGCUGUCCGUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU----------------------------GCAUGUGGCUCUUAA ((((((...((.....))......((((((((((..((((.(((.......)))))))...))))))))----------------------------)).))))))...... ( -36.70) >consensus GCCACAAAACCUUUAUGGCUGCCCAUACACACACUGGCAUACCGAAGUGUGCGGGUGCAAUGUGUGUGU__GCAUGUGGCUCUGCGUGUGUAUGGGUGCUUGUGGCUCUCAA ((((((..........(((.((((((((((((((..((((.(((.......)))))))...))))))....(((((((....))))))))))))))))))))))))...... (-35.71 = -38.06 + 2.35)

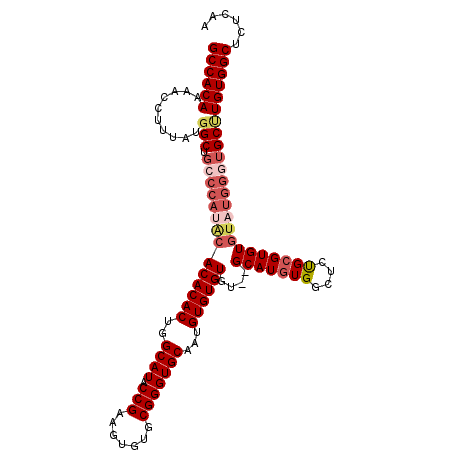
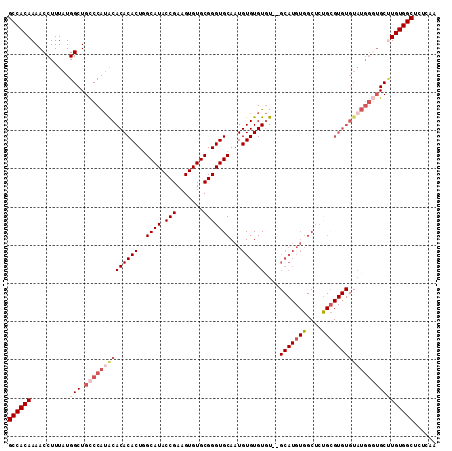
| Location | 18,018,440 – 18,018,550 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 85.38 |
| Mean single sequence MFE | -39.34 |
| Consensus MFE | -28.94 |
| Energy contribution | -30.50 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.60 |
| Structure conservation index | 0.74 |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.996397 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 18018440 110 - 22407834 UUGAGAGCCACAAGCACCCAUACACACGCAGAGCCACAUGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUAUGGGCAGCCAUAAAGGUUUUGUGGC ......(((((((...(((((((((((((.........(((--(.........))))...(((.((....)).)))..))))))))))))).((((.....))))))))))) ( -44.30) >DroSec_CAF1 234172 110 - 1 UUGAGAGCCACAAGCACCCAUACACACGCAGAGCCACAUGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCUAGUGUGUGCAUGGGCAGCCAUAAAGGUUUUGUGGC ......(((((((...................(((.(((((--((((((.....(....)(((.((....)).)))..))))))))))))))((((.....))))))))))) ( -42.80) >DroSim_CAF1 233906 110 - 1 UUGAGAGCCACAAGCACCCAUACACACGCAGAGCCACAUGC--ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGCAUGGGCAGCCAUAAAGGUUUUGUGGC ......(((((((...................(((.(((((--((((((....(((....))).((....))......))))))))))))))((((.....))))))))))) ( -42.80) >DroEre_CAF1 226819 112 - 1 UUAAGAGCCACAGGCAACCAUACACACCCGGAGCCACAUGCACAUACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUCUGGACAGCCAUAAAGGUUUUGUGGC ......(((((((((..((..........)).)))...((..((.((((((((((.....(((.((....)).))))))))))))).))..))(((.....)))..)))))) ( -36.90) >DroYak_CAF1 243562 84 - 1 UUAAGAGCCACAUGC----------------------------ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUACGGACAGCCAUAAAGGUUUUGUGGC ......((((((.((----------------------------..((((((((((.....(((.((....)).)))))))))))))..((....))......))..)))))) ( -29.90) >consensus UUGAGAGCCACAAGCACCCAUACACACGCAGAGCCACAUGC__ACACACACAUUGCACCCGCACACUUCGGUAUGCCAGUGUGUGUAUGGGCAGCCAUAAAGGUUUUGUGGC ......(((((((...................(((.((((......(((((((((.....(((.((....)).)))))))))))))))))))((((.....))))))))))) (-28.94 = -30.50 + 1.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:28 2006