
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 17,916,590 – 17,916,691 |
| Length | 101 |
| Max. P | 0.731997 |

| Location | 17,916,590 – 17,916,691 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 79.60 |
| Mean single sequence MFE | -26.11 |
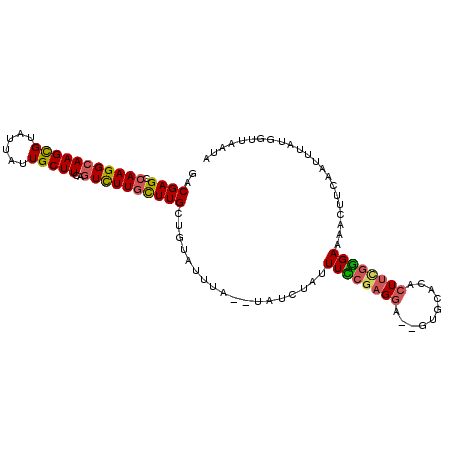
| Consensus MFE | -18.14 |
| Energy contribution | -18.20 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.731997 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
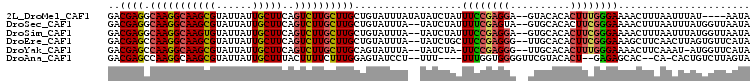
>2L_DroMel_CAF1 17916590 101 - 22407834 GACGAGGCAAGGCAAGCGUAUUAUUGCUUCAGUCUUGCUUGCUGUAUUUAUAUAUCUAUUUCCGAGGA--GUACACACUUUGGGAAAACUUUAAUUUAU----AAUA ....((((((((((((((......)))))..)))))))))..................(((((((((.--(....).))))))))).............----.... ( -24.90) >DroSec_CAF1 134290 103 - 1 GACGAGGCAAGGCAAGCGUAUUAUUGCUUCAGUCUUGCUUGCUGUAUUUA--UAUCUAUUUUCGAGUA--GUGCACACUUCGGGAAAACUUUAAUUUAUGGUUAAUA ....((((((((((((((......)))))..)))))))))((((((....--......((..(((..(--((....))))))..))..........))))))..... ( -24.15) >DroSim_CAF1 112015 103 - 1 GACGAGGCAAGGCAAGUGUAUUAUUGCUUCAGUCUUGCUUGCUGUAUUUA--UAUCUAUUUCCGAGGA--GUGCACACUUCGGGAAAACUUUAAUUUAUGGUUAAUA ....((((((((((((((......)))))..)))))))))((((((....--......(((((((((.--(....).)))))))))..........))))))..... ( -26.65) >DroEre_CAF1 123840 103 - 1 GACGAGCCAAGGCAAGCGUAUUAUUGCUUCAGUCUUGCUUGCUGUAUUUA--UAUCUGCUUCCGAGGG--UUGCACACUUCGGGAAAGCUUCAACUUAGUGUUCAUA ...(((((..((((((((....((((...))))..)))))))).......--.....((((((((((.--.......)))))))..))).........).))))... ( -26.90) >DroYak_CAF1 131668 101 - 1 GACGAGCCAAGGCAAGCGUAUUAUUGCUUCAGUCUUGCUUGCAGUAUUUA--UAUCUA-UUCCGAGGG--UUGCACACUUUGGGAAAACUUCAAAU-AUGGUUCAUA ...(((((...(((((((....((((...))))..))))))).((((((.--......-((((((((.--.......)))))))).......))))-)))))))... ( -27.74) >DroAna_CAF1 127538 96 - 1 GACGAGCCAAGGCAAGCGUAUUAUUGCUUUACUUUUCUUUGGAGUAUCCU--UUU----UUUGGUGGGGUUCGUACACU--GAGAGCAC--CA-CACUGUCUUAGUA .((((((((((((((........)))))))(((.......((.....)).--...----...)))..)))))))..(((--((((.((.--..-...))))))))). ( -26.34) >consensus GACGAGCCAAGGCAAGCGUAUUAUUGCUUCAGUCUUGCUUGCUGUAUUUA__UAUCUAUUUCCGAGGA__GUGCACACUUCGGGAAAACUUCAAUUUAUGGUUAAUA ..((((.(((((((((((......)))))..))))))))))..................((((((((..........))))))))...................... (-18.14 = -18.20 + 0.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:09:49 2006