
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
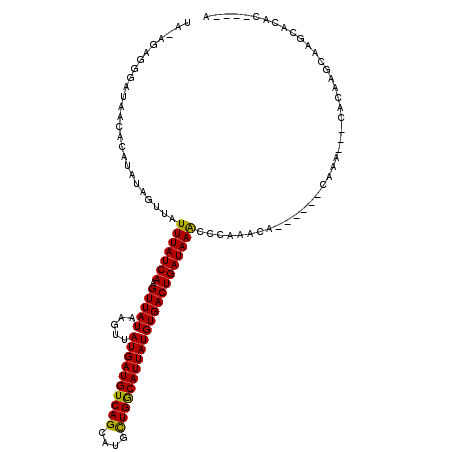
| Location | 17,319,015 – 17,319,136 |
| Length | 121 |
| Max. P | 0.999973 |

| Location | 17,319,015 – 17,319,119 |
|---|---|
| Length | 104 |
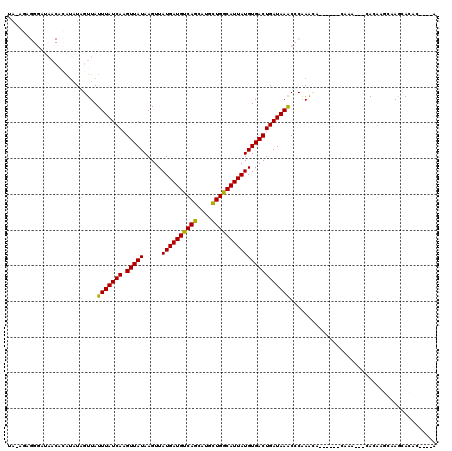
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.04 |
| Mean single sequence MFE | -26.12 |
| Consensus MFE | -19.90 |
| Energy contribution | -19.40 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.57 |
| SVM RNA-class probability | 0.964449 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17319015 104 - 22407834 UA-AGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAACCCGAACA------CAAA---CACAAGUGAGCACA------ ..-...(((.((((.......))))(((((((.(((((.....((((((((((....)))))))))))))))))))))))))(..((------(...---.....)))..)...------ ( -27.80) >DroVir_CAF1 33994 106 - 1 UA-AAA---------GGUUUAGUUAUUUAUCAUGUUAUAAGUUAUGAUGUCAGCGUGCUGGCAUUAUGUGACUGAUAAGCGCAAGAAAAUGAGCAAGGCUCACACGCAAAGACAC----A ..-...---------.((((......((((((.(((((.....((((((((((....)))))))))))))))))))))(((........(((((...)))))..)))..))))..----. ( -26.60) >DroSec_CAF1 16620 110 - 1 UA-AGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGACAUUAUGUGACUGAUAAACCCGAACA------CAAA---CACAAGUGAGCACACAUACA ..-...(((.((((.......))))(((((((.(((((.....((((((((((....)))))))))))))))))))))))))(..((------(...---.....)))..)......... ( -28.40) >DroSim_CAF1 18156 106 - 1 UA-AGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAACCCGAACA------CAAA---CACAAGUGAGCACAC----A ..-...(((.((((.......))))(((((((.(((((.....((((((((((....)))))))))))))))))))))))))(..((------(...---.....)))..)....----. ( -27.80) >DroMoj_CAF1 39938 104 - 1 UAACAA---------GACAUAGUUAUUUAUCAUGUUAUAAGUUAUGAUGUCAGCAUGUUGGCAUUAUGUGACUGAUAAGCGCAAGAAAAUGAGCAGA---CACACACAAGCACAC----A ......---------......(((..((((((.(((((.....((((((((((....)))))))))))))))))))))((.((......)).)).))---)..............----. ( -20.40) >DroAna_CAF1 26151 104 - 1 UA-AGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAACCCAAGCA------CAAA---CACAAACAAACACA------ ..-...(((.((((.......))))(((((((.(((((.....((((((((((....))))))))))))))))))))))))).....------....---..............------ ( -25.70) >consensus UA_AGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAACCCAAACA______CAAA___CACAAGCAAGCACAC____A .........................(((((((.(((((.....((((((((((....))))))))))))))))))))))......................................... (-19.90 = -19.40 + -0.50)

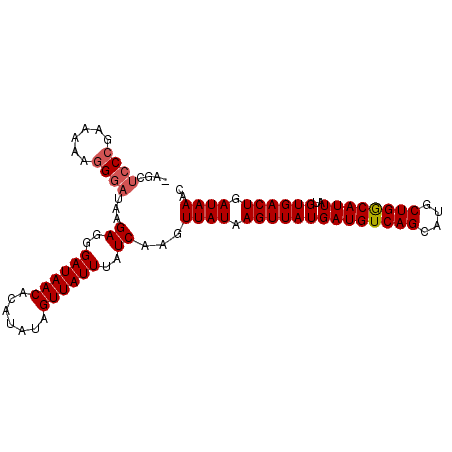
| Location | 17,319,040 – 17,319,136 |
|---|---|
| Length | 96 |
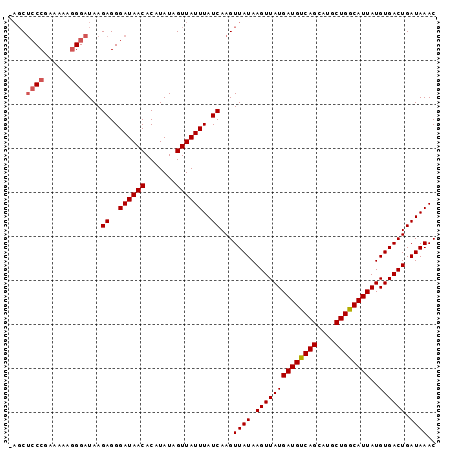
| Sequences | 6 |
| Columns | 97 |
| Reading direction | reverse |
| Mean pairwise identity | 96.89 |
| Mean single sequence MFE | -26.72 |
| Consensus MFE | -25.24 |
| Energy contribution | -25.60 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.58 |
| Structure conservation index | 0.94 |
| SVM decision value | 5.09 |
| SVM RNA-class probability | 0.999973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17319040 96 - 22407834 -AGCUCCCGAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAAC -...((((......))))...((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. ( -28.00) >DroSec_CAF1 16651 96 - 1 -AGCUCCCAAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGACAUUAUGUGACUGAUAAAC -...((((......))))...((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. ( -27.90) >DroSim_CAF1 18183 96 - 1 -AGCUCCCGAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAAC -...((((......))))...((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. ( -28.00) >DroEre_CAF1 18637 96 - 1 -AGCUACCGAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCACGCUGGCAUUAUGUGACUGAUAAAC -.....((......)).....((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. ( -23.90) >DroYak_CAF1 11586 96 - 1 -AGCUCCCGAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAAC -...((((......))))...((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. ( -28.00) >DroAna_CAF1 26176 97 - 1 GCCCGCCCGAAAAAAGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAAC .(((..((.......))......)))(((((.......)))))((((((.(((((.....((((((((((....))))))))))))))))))))).. ( -24.50) >consensus _AGCUCCCGAAAAAGGGAUAAGAGGGAUAACACAUAUAGUUAUUUAUCAAGUUAUAAGUUAUGAUGUCAGCAUGCUGGCAUUAUGUGACUGAUAAAC ....((((......))))...((..((((((.......))))))..))...((((.((((((((((((((....))))))))..)))))).)))).. (-25.24 = -25.60 + 0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:05:51 2006