
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 238,710 – 238,901 |
| Length | 191 |
| Max. P | 0.981726 |

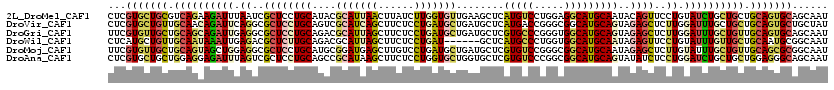
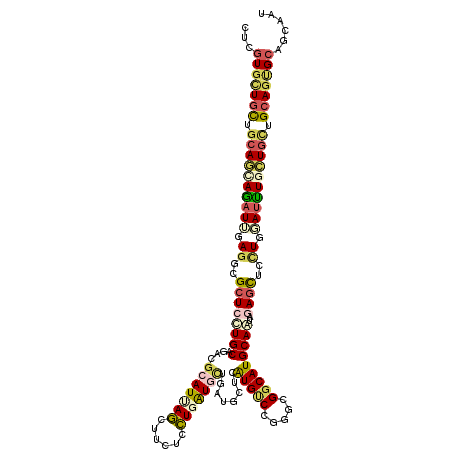
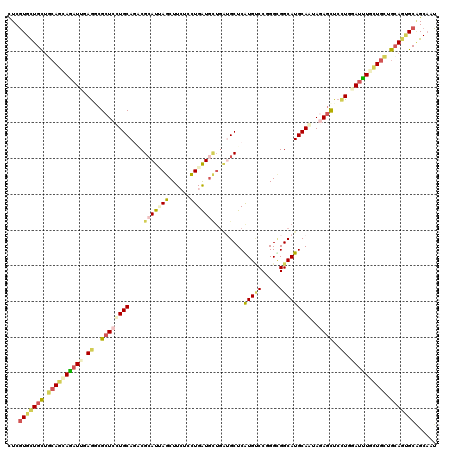
| Location | 238,710 – 238,830 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -50.57 |
| Consensus MFE | -32.60 |
| Energy contribution | -33.42 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.46 |
| Mean z-score | -3.57 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.981726 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
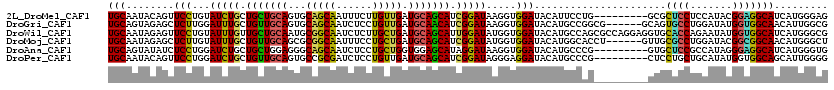
>2L_DroMel_CAF1 238710 120 + 22407834 CUCGUGCUGCGUCAGAAGAUUUAAUCGCUCCUGCAUACGCAUUAACUUAUCUUGGUGUUGAAGCUCAUGUCCUGGAGGCAUGCAAUACAGUUCCUGUAUCUGCUGCUGCAGUGCAGCAAU ...(..((((.................((((.((((..((.(((((..........))))).))..))))...))))(((.(((((((((...)))))).)))))).))))..)...... ( -35.70) >DroVir_CAF1 36072 120 + 1 CUCGUGCUGUUGCAACAGAUUCAGGCGCUCCUGCAGUCGCAUCAGCUUCUCCUGAUGCUGAUGCUCAUGACCGGGCGGCAUGCAGUAGAGCUCUUGGAUUUGCUGCUGCAGUGCUGCUAU ...(..((((.(((.((((((((((.(((((((((((((((((((......)))))))....((((......))))))).)))))..)))).)))))))))).))).))))..)...... ( -58.40) >DroGri_CAF1 37007 120 + 1 UUCGUGUUGCUGCAGCAGAUUGAGGCGCUCCUGCAGACGCAUUAGCUUCUCCUGAUGCUGAUGCUCGUGCCCGGGUGGCAUGCAGUAGAGCUCUUGGAUUUGCUGUUGCAGUGCAGCAAU ...(..((((.((((((((((.(((.((((((((.((.((((((((.((....)).))))))))))(((((.....)))))))))..)))).))).)))))))))).))))..)...... ( -60.50) >DroWil_CAF1 39060 114 + 1 CUCAUGCUGUUGCAAUAAAUUGAGACGCUCUUGCAGACGCAUUAGCUUCUCCUGAU------GCUCAUGCCCUGGUGGCAUGCAAUAGAGUUCCUGUAUUUGUUGCUGCAAUGCGGCAAU ....((((((.(((((((((..((..((((((((.((.(((((((......)))))------))))(((((.....))))))))..)))))..))..)))))))))......)))))).. ( -40.20) >DroMoj_CAF1 34567 120 + 1 UUCGUGUUGCUGCAGUAGCUGGAGGCGCUCCUGCAUGCGGAUGAGCUUGUCCUGAUGCUGAUGCUCGUGUCCGGGCGGCAUGCAAUAGAGCUCUUGUAUUUGCUGUUGCAGCGCGGCAAU .(((((((((.(((((((.((.(((.((((.(((((((((((......)))).........((((((....)))))))))))))...)))).))).)).))))))).))))))))).... ( -55.20) >DroAna_CAF1 36748 120 + 1 CUCGUGCUGCUGGAGGAGAUUUAGUCGCUCCUGCAGCCGCAUAAGCUUCUCCUGGUGCUGGUGCUCGUGUCCCGGCGGCAUGCAGUAUAUCUCCUGGAUCUGCUGCUGGAGGGCAGCAAU ...((((.((((.(((((.........))))).)))).))))..(((.((((((.((((((..(....)..)))))).)).((((((.(((.....))).)))))).)))))))...... ( -53.40) >consensus CUCGUGCUGCUGCAGCAGAUUGAGGCGCUCCUGCAGACGCAUUAGCUUCUCCUGAUGCUGAUGCUCAUGUCCGGGCGGCAUGCAAUAGAGCUCCUGGAUUUGCUGCUGCAGUGCAGCAAU ...(((((((.((((((((((.((..((((((((....(((((((......)))))))........(((((.....)))))))))..))))..)).)))))))))).)))))))...... (-32.60 = -33.42 + 0.81)
| Location | 238,750 – 238,870 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.50 |
| Mean single sequence MFE | -42.10 |
| Consensus MFE | -27.57 |
| Energy contribution | -26.93 |
| Covariance contribution | -0.63 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.547531 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 238750 120 + 22407834 AUUAACUUAUCUUGGUGUUGAAGCUCAUGUCCUGGAGGCAUGCAAUACAGUUCCUGUAUCUGCUGCUGCAGUGCAGCAAUUUCUUGUUGAUGCAGCAUCGGAUAAGGUGGAUACAUUCCU ..............((((((.....((((((.....))))))))))))....(((.((((((.(((((((...((((((....)))))).))))))).))))))))).(((.....))). ( -39.70) >DroVir_CAF1 36112 120 + 1 AUCAGCUUCUCCUGAUGCUGAUGCUCAUGACCGGGCGGCAUGCAGUAGAGCUCUUGGAUUUGCUGCUGCAGUGCUGCUAUUUCCUGCUGAUGCAGCAUCGGAUACGGCGGAUACAUGCCA ((((((.((....)).))))))((((......))))((((((((((((..(.....).....))))))).((((((((...((((((((...)))))..)))...))))).)))))))). ( -47.70) >DroGri_CAF1 37047 120 + 1 AUUAGCUUCUCCUGAUGCUGAUGCUCGUGCCCGGGUGGCAUGCAGUAGAGCUCUUGGAUUUGCUGUUGCAGUGCAGCAAUCUCCUGUUGAUGCAACAUCGGAUAAGGUGGAUACAUGCCG ((((((.((....)).))))))(((((....)))))((((((..((...((.(((..(((((.(((((((...(((((......))))).))))))).))))))))))..)).)))))). ( -40.20) >DroWil_CAF1 39100 114 + 1 AUUAGCUUCUCCUGAU------GCUCAUGCCCUGGUGGCAUGCAAUAGAGUUCCUGUAUUUGUUGCUGCAAUGCGGCAAUCUCUUGCUGAUGCAGCAUUGGAUAUGGUGGAUACAUGCCA ....((.....(((.(------((..(((((.....)))))))).))).((((((((((((..(((((((...((((((....)))))).)))))))..)))))))).))))....)).. ( -38.30) >DroMoj_CAF1 34607 120 + 1 AUGAGCUUGUCCUGAUGCUGAUGCUCGUGUCCGGGCGGCAUGCAAUAGAGCUCUUGUAUUUGCUGUUGCAGCGCGGCAAUUUCCUGCUGAUGCAGCAUCGGAUAUGGUGGAUACAUGGCA ....((((((((.(((((((.((((((....))))))((.((((((((.((....)).....)))))))))).(((((......)))))...))))))))))))..(((....)))))). ( -43.90) >DroAna_CAF1 36788 120 + 1 AUAAGCUUCUCCUGGUGCUGGUGCUCGUGUCCCGGCGGCAUGCAGUAUAUCUCCUGGAUCUGCUGCUGGAGGGCAGCAAUCUCCUGCUGGUGGAGCAUAGGAUAAGGUGGAUACAUGCCC ....(((......)))(((((..(....)..)))))((((((.....((((((((..((((((((((....))))))...((((.......))))....)))).))).))))))))))). ( -42.80) >consensus AUUAGCUUCUCCUGAUGCUGAUGCUCAUGUCCGGGCGGCAUGCAAUAGAGCUCCUGGAUUUGCUGCUGCAGUGCAGCAAUCUCCUGCUGAUGCAGCAUCGGAUAAGGUGGAUACAUGCCA ....(((...(.(((.((....))))).)....)))((((((...((...(..((..(((((.(((((((...(((((......))))).))))))).)))))..))..).)))))))). (-27.57 = -26.93 + -0.63)

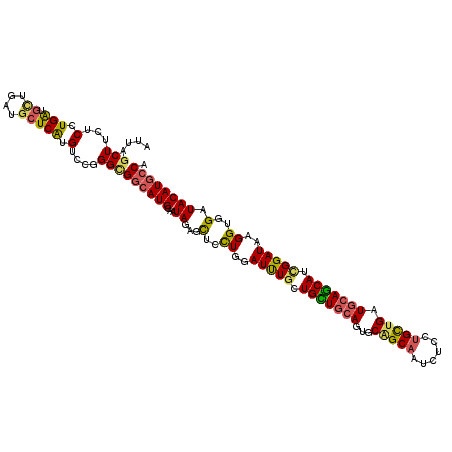
| Location | 238,790 – 238,901 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.35 |
| Mean single sequence MFE | -42.22 |
| Consensus MFE | -27.36 |
| Energy contribution | -26.25 |
| Covariance contribution | -1.11 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901902 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
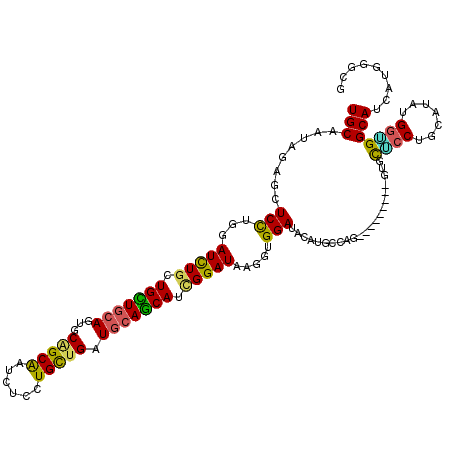
>2L_DroMel_CAF1 238790 111 + 22407834 UGCAAUACAGUUCCUGUAUCUGCUGCUGCAGUGCAGCAAUUUCUUGUUGAUGCAGCAUCGGAUAAGGUGGAUACAUUCCUG---------GCGCUCCUCCAUACGGAGGCAUCAUGGGAG .........(((((..((((((.(((((((...((((((....)))))).))))))).))))))....))).))..(((((---------.....(((((....))))).....))))). ( -42.00) >DroGri_CAF1 37087 114 + 1 UGCAGUAGAGCUCUUGGAUUUGCUGUUGCAGUGCAGCAAUCUCCUGUUGAUGCAACAUCGGAUAAGGUGGAUACAUGCCGGCG------GCAGUGCCUGGAUAUGGUGGCAACAUUGGCG ....(((..((.(((..(((((.(((((((...(((((......))))).))))))).))))))))))...)))..(((((((------....)))).......((((....))))))). ( -35.80) >DroWil_CAF1 39134 120 + 1 UGCAAUAGAGUUCCUGUAUUUGUUGCUGCAAUGCGGCAAUCUCUUGCUGAUGCAGCAUUGGAUAUGGUGGAUACAUGCCAGCGCCAGGAGGUGCACCAGAAUAUGGUGGCAUCAUGGGCG .((....(.((((((((((((..(((((((...((((((....)))))).)))))))..)))))))).)))).)(((((.(((((....)))))((((.....))))))))).....)). ( -48.60) >DroMoj_CAF1 34647 114 + 1 UGCAAUAGAGCUCUUGUAUUUGCUGUUGCAGCGCGGCAAUUUCCUGCUGAUGCAGCAUCGGAUAUGGUGGAUACAUGGCACCU------GUUGCGCCUGGAUACGGCGGCAACAUGGGCU .((......(((..((((((..((((((((...(((((......))))).))))))).((....)))..)))))).))).(((------((((((((.......))).)))))).)))). ( -43.70) >DroAna_CAF1 36828 111 + 1 UGCAGUAUAUCUCCUGGAUCUGCUGCUGGAGGGCAGCAAUCUCCUGCUGGUGGAGCAUAGGAUAAGGUGGAUACAUGCCCG---------GUGCUCCGCCAUAGGGAGGCAUCAUGGGUG ............(((((((.(((((((....))))))).((((((..(((((((((((.((.....(((....)))..)).---------)))))))))))..)))))).))).)))).. ( -44.60) >DroPer_CAF1 37859 111 + 1 UGCAAUACAGUUCCUGGAUCUGCUGUUGCAGUGCCGCGAUCUCCUGUUGAUGCAGCAUCGGAUAGGGAGGAUACAUGCCCG---------CUCCUGCUGCAUAUGGUGGCAGCAUUGGGG ((((((((((((....)).))).)))))))...((.((((((((.....((((((((..(((..(((.(....)...))).---------.))))))))))).....)).)).)))).)) ( -38.60) >consensus UGCAAUAGAGCUCCUGGAUCUGCUGCUGCAGUGCAGCAAUCUCCUGCUGAUGCAGCAUCGGAUAAGGUGGAUACAUGCCAG_________GUGCUCCUGCAUAUGGUGGCAUCAUGGGCG (((........(((...(((((.(((((((...(((((......))))).))))))).))))).....)))......................((((.......)))))))......... (-27.36 = -26.25 + -1.11)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:24:45 2006