
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 17,279,832 – 17,279,941 |
| Length | 109 |
| Max. P | 0.542738 |

| Location | 17,279,832 – 17,279,941 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 79.22 |
| Mean single sequence MFE | -30.38 |
| Consensus MFE | -18.68 |
| Energy contribution | -19.02 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.542738 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17279832 109 - 22407834 GUAUCAAUCUG-AGCGUUGUACAUGUAUGCG-UGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCAGCAGCAGCUUCGGGCU .....((((((-((((.(((((........)-)))).))))))))))....(((........(((((.((((......))))...))))).((.(((....))).))))). ( -34.70) >DroSec_CAF1 117930 109 - 1 GUAUCCAUCUG-AGCGUUGUACAUGUAUGCG-UGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCAGCAGCAGCUUCGGGCU ...((((((((-((((.(((((........)-)))).)))))))))....(((.(((.((..(((((.((((......))))...)))))..))))).))).....))).. ( -34.40) >DroEre_CAF1 117559 110 - 1 GCAUACUUUCGAAGCGUUGUAUAUGUGUGCG-UGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCAGCAGCAGCUUCGGGCU .......(((((((((((((...((((((((-.((((.(........).)))))))......(((((.((((......))))...))))))))))))))).)))))))).. ( -35.20) >DroWil_CAF1 141383 90 - 1 ---------------GAUGUACGUGUAUAUGCUGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCU-CAGCAAACCC----- ---------------((((((.((......))))))))((((.(....((((.(((((((.......((((.....))))...))))))).))))-...)))))..----- ( -21.30) >DroYak_CAF1 120185 110 - 1 GUAUCCUUCUGAAGCGCUGUAUAUGUGUGCG-UGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCAGCAGCAGCUUCGGGCU .......(((((((((((((...((((((((-.((((.(........).)))))))......(((((.((((......))))...))))))))))))))).)))))))).. ( -38.20) >DroAna_CAF1 186172 88 - 1 GUAAACU----------CGUAUGUGUUUUAGCAGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCGUGUCAUUUAUAAGCAGCACAGCG------------- .......----------(((.((((((..(((.((((.(........).)))).)))......((((.((((......))))...)))))))))))))------------- ( -18.50) >consensus GUAUCCUUCUG_AGCGUUGUACAUGUAUGCG_UGCAUCGUUUAGAUUUAGUGCCGCUUGUUACGCUUUGAUGUCAUGUCAUUUAUAAGCGACGCAGCAGCAGCUUCGGGCU .......((((.((((((((..(((((.....)))))............(((.(((((((.......((((.....))))...))))))).))).))))).))).)))).. (-18.68 = -19.02 + 0.34)

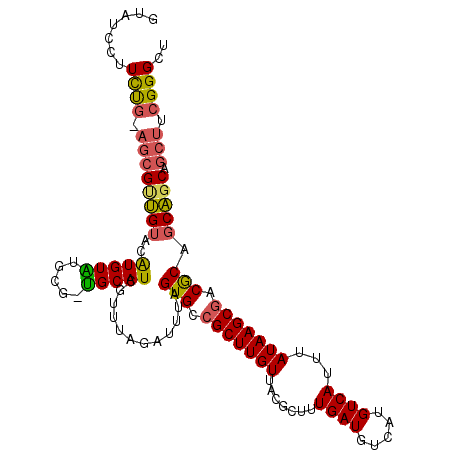
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:04:51 2006