
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 17,153,413 – 17,153,649 |
| Length | 236 |
| Max. P | 0.999484 |

| Location | 17,153,413 – 17,153,529 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.11 |
| Mean single sequence MFE | -36.68 |
| Consensus MFE | -33.88 |
| Energy contribution | -33.64 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.81 |
| SVM RNA-class probability | 0.978297 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17153413 116 + 22407834 UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACACCGCACACACCCACACACAUGCAUA----UACAUAUGCGUCUGAGGAGUAGUGUCUGUGCUGGU .(..(((((....((...(((((.......)))))...))((((((((..((......))......((...((.(((((((----....))))))).)).)).)))))))))))))..). ( -34.90) >DroSec_CAF1 114289 116 + 1 UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACACCGCACACACCCACACACAUGCAUA----UACAUAUGCGUCUGAGGAGUAGUGUCUGUGCUAGU .((((((((....((...(((((.......)))))...))((((((((..((......))......((...((.(((((((----....))))))).)).)).)))))))))))))))). ( -35.10) >DroSim_CAF1 126488 116 + 1 UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACACCGCACACACCCACACACAUGCAUA----UACAUAUGCGUCUGAGGAGUAGUGUCAGUGCUAGU .(((((((.....((...(((((.......)))))...))((((((((..((......))......((...((.(((((((----....))))))).)).)).)))))))).))))))). ( -35.00) >DroEre_CAF1 111642 116 + 1 UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACAGCGCACACACCCACACACAUGCAUA----UACAUAUGCGUCUGAGGAGUAGUGUCUGUGCUGGU .(..(((((...(((...(((((.......)))))...)))((((((.......))))))((((((((...((.(((((((----....))))))).)).)).)).)))).)))))..). ( -38.50) >DroYak_CAF1 114311 120 + 1 UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACACCGCACUCACCCACACACAUGCAUAUGUAUACAUAUGCGUCUGAGGAGUAGUGUCUGUGCUAGU .((((((((....((...(((((.......)))))...))((((((((..((......))......((...((.(((((((((....))))))))).)).)).)))))))))))))))). ( -39.90) >consensus UUUGGCGCAAUUUGCAUGUGAAUGUGAUAAAUUCAAUGGCGGCGCUGCACGUUACACCGCACACACCCACACACAUGCAUA____UACAUAUGCGUCUGAGGAGUAGUGUCUGUGCUAGU .(((((((.....((...(((((.......)))))...))((((((((..((......))......((...((.(((((((........))))))).)).)).)))))))).))))))). (-33.88 = -33.64 + -0.24)

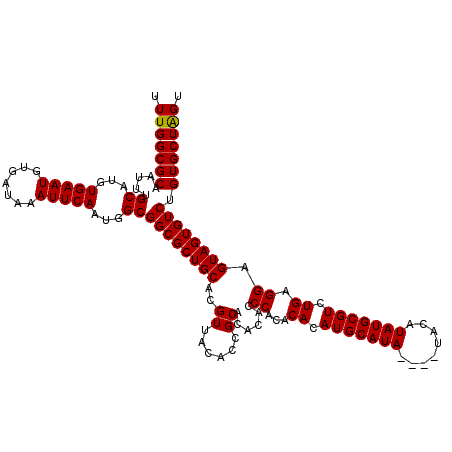
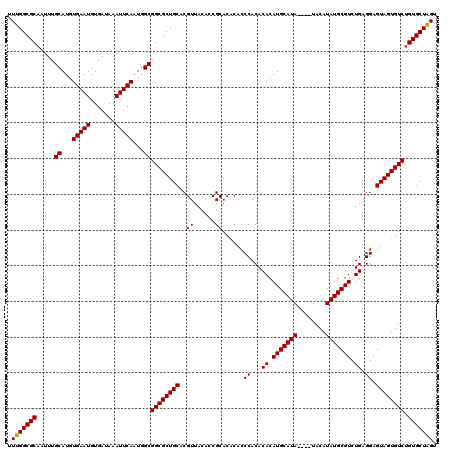
| Location | 17,153,413 – 17,153,529 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.11 |
| Mean single sequence MFE | -37.48 |
| Consensus MFE | -35.16 |
| Energy contribution | -35.56 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.64 |
| SVM RNA-class probability | 0.999484 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17153413 116 - 22407834 ACCAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....(((((.((.((((((....)).((((((((.----...)))))))))))))).)))))(((((((..((((((...))...(((((.......)))))..))))..)))))))... ( -37.20) >DroSec_CAF1 114289 116 - 1 ACUAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....(((((.((.((((((....)).((((((((.----...)))))))))))))).)))))(((((((..((((((...))...(((((.......)))))..))))..)))))))... ( -37.20) >DroSim_CAF1 126488 116 - 1 ACUAGCACUGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....(((((((.........))))..(((((....----))))).((((((((((((((.(((((((........)))))))............))))))))))))))...)))...... ( -37.02) >DroEre_CAF1 111642 116 - 1 ACCAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGCUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....((.(((................(((((....----))))).((((((((((((((.(((((((.....)))))))...............))))))))))))))..))).)).... ( -36.09) >DroYak_CAF1 114311 120 - 1 ACUAGCACAGACACUACUCCUCAGACGCAUAUGUAUACAUAUGCAUGUGUGUGGGUGAGUGCGGUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....((((..((((((((((((..((((((((((((....))))))))))))))).))))..)))))...)))).((((.((...(((((.......)))))...)).....)))).... ( -39.90) >consensus ACUAGCACAGACACUACUCCUCAGACGCAUAUGUA____UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCCGCCAUUGAAUUUAUCACAUUCACAUGCAAAUUGCGCCAAA ....((((..(((((.((....))(((((((((((......))))))))))).)))))))))(((((((..((((((...))...(((((.......)))))..))))..)))))))... (-35.16 = -35.56 + 0.40)

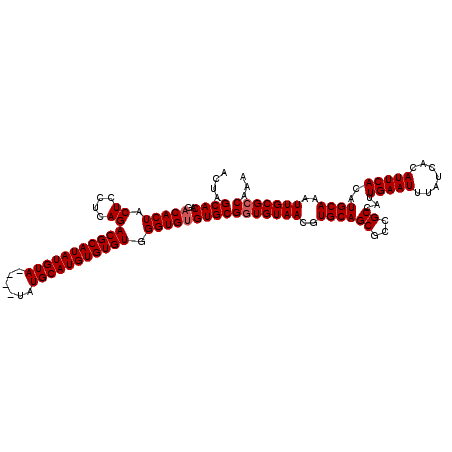

| Location | 17,153,453 – 17,153,569 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.77 |
| Mean single sequence MFE | -39.16 |
| Consensus MFE | -36.46 |
| Energy contribution | -36.46 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.994798 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17153453 116 - 22407834 UCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCACACCAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCC ..(((((((......))))))).........((((((.(((((.(((((.((.((((((....)).((((((((.----...)))))))))))))).))))))))))..)))).)).... ( -38.90) >DroSec_CAF1 114329 116 - 1 UCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCACACUAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCC ..(((((((......))))))).........((((((.(((((.(((((.((.((((((....)).((((((((.----...)))))))))))))).))))))))))..)))).)).... ( -36.70) >DroSim_CAF1 126528 116 - 1 UCUUUCUGCGGGAAAGCGGGAAAAACAAAUACUCACGCACACUAGCACUGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCC ..(((((((......)))))))..............((((....((((((.(((.((.(((...((((((((((.----...))))))))))))).)))))))))))...))))...... ( -41.50) >DroEre_CAF1 111682 116 - 1 UCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCACACCAGCACAGACACUACUCCUCAGACGCAUAUGUA----UAUGCAUGUGUGUGGGUGUGUGCGCUGUAACGUGCAGCGCC .....(((((((...(((.((...........)).)))...))..)))))((((.((((.....((((((((((.----...))))))))))))))))))(((((((.....))))))). ( -39.40) >DroYak_CAF1 114351 120 - 1 UCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCACACUAGCACAGACACUACUCCUCAGACGCAUAUGUAUACAUAUGCAUGUGUGUGGGUGAGUGCGGUGUAACGUGCAGCGCC ..(((((((......)))))))........((((((.((((((((........)))..........(((((((....)))))))....))))).))))))(((.(((.....))).))). ( -39.30) >consensus UCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCACACUAGCACAGACACUACUCCUCAGACGCAUAUGUA____UAUGCAUGUGUGUGGGUGUGUGCGGUGUAACGUGCAGCGCC ..(((((((......))))))).........((((((.(((((.((((..(((((.((....))(((((((((((......))))))))))).))))))))))))))..)))).)).... (-36.46 = -36.46 + 0.00)

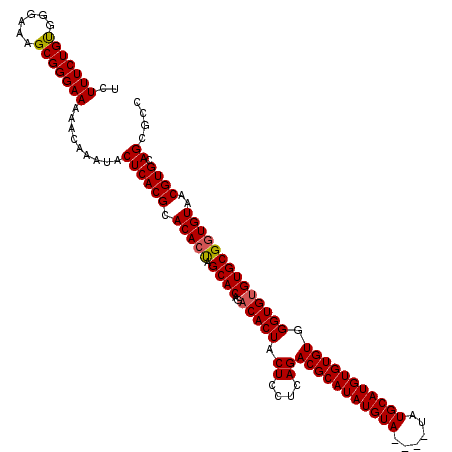
| Location | 17,153,529 – 17,153,649 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.25 |
| Mean single sequence MFE | -32.20 |
| Consensus MFE | -29.40 |
| Energy contribution | -29.64 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.967002 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 17153529 120 - 22407834 UGUGGGUGGGGUCACGCCCACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUCCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCAC .((((((((((.....)))..............(((((......(((((((((....(((.....)))..)))))))))(((((.....))))))))))..........))))))).... ( -31.90) >DroSec_CAF1 114405 120 - 1 UGUGGGUGGGGUCACGCCCACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUCCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCAC .((((((((((.....)))..............(((((......(((((((((....(((.....)))..)))))))))(((((.....))))))))))..........))))))).... ( -31.90) >DroSim_CAF1 126604 120 - 1 UGUGGGUGGGGUCACGCCCACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUCCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGCGGGAAAGCGGGAAAAACAAAUACUCACGCAC .(((((((......)))))))..............((........((((((((....(((.....)))..))))))))(((.(((((((......))))))).)))..........)).. ( -33.30) >DroEre_CAF1 111758 120 - 1 UGUGGGUGGGGUUGCGCCAACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUGCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCAC .(((.(((((((((...))))............(((((.((((((((((.(((((....).)))).))).)))))))..(((((.....))))))))))............))))).))) ( -32.80) >DroYak_CAF1 114431 120 - 1 UGGGGGUGGGUUUCCGCCCACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUCCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCAC (((((((((((....)))))))...........(((((......(((((((((....(((.....)))..)))))))))(((((.....))))))))))............))))..... ( -31.10) >consensus UGUGGGUGGGGUCACGCCCACUUAUUUAAUAUACCGCUACACUUUUCAUUUUUCCAAACGUCAAACGUGAAAAAGUGAGUUCUUUCUGUGGGAAAGCGGGAAAAACAAAUACUCACGCAC .(((((((......)))))))..............((........((((((((....(((.....)))..))))))))(((.(((((((......))))))).)))..........)).. (-29.40 = -29.64 + 0.24)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:03:56 2006