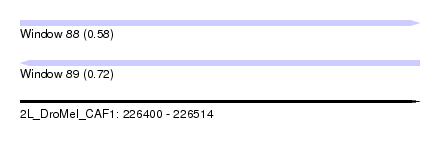
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 226,400 – 226,514 |
| Length | 114 |
| Max. P | 0.719787 |

| Location | 226,400 – 226,514 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.93 |
| Mean single sequence MFE | -48.52 |
| Consensus MFE | -23.65 |
| Energy contribution | -25.32 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.575406 |
| Prediction | RNA |
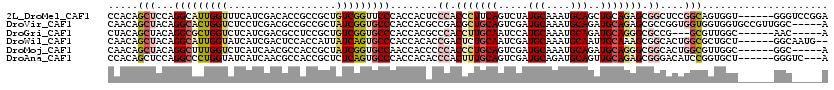
Download alignment: ClustalW | MAF
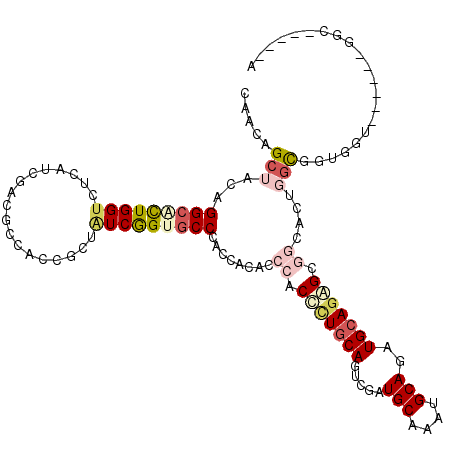
>2L_DroMel_CAF1 226400 114 + 22407834 UCCGGACCC------ACCACUGCCGGAGCCGCUCUGCAGCUGCAUUUGCAUAGACUGAAGGGUGGGAGUGGUGGGAACCGACAGCGGCGGUGUCGAUGAAACCAAUGCCUGGAGCUGUGG ..(((.(((------((((((.(((....)(((((.(((((((....))..)).))).))))))).)))))))))..)))(((((((((((.........)))...)))....))))).. ( -46.60) >DroVir_CAF1 23139 115 + 1 U-----GCCAACGGCACCACCACCACCGGCGCUCUGCAUCUGCAUUUGCAUCGACUGCAGCGUCGGCGUGGUGGGCACCGAUAGCGGCGGCGUCGAGGAGACCAGUGCCUGUAGCUGUUG .-----..(((((((.(((((((..((((((((..((((((((....)))..)).))))))))))).)))))))((((((....))).((((..(.(....))..))))))).))))))) ( -54.30) >DroGri_CAF1 22290 106 + 1 U-----GUU------GCCAACGC---CGGCGCCCUGCAUCUGCAUUUGCAUGGAUUGCAAGGUGGGCGUGGUGGGCACCGACAGCGGAGGCGUCGAUGAGACCAGCGCCUGUAGCUGUAG .-----(.(------(((..(((---((.((((((((....)))((((((.....))))))..)))))))))))))).).(((((..((((((.(.......).))))))...))))).. ( -46.70) >DroWil_CAF1 25450 112 + 1 --CAUUGCC------AGCAGCGCCAGUGCCGCUUUGCAAUUGCAUUUGCAUCGAUUGCAGAGUCGGUGUGGUGGGCACUGAUAAUGGUGGAGUCGAUGAUACCAAUGCCUGUAGCUGUUG --......(------((((((((((.(((((((((((((((((....)))...))))))))).)))))))))(((((((......))(((.(((...))).))).)))))...))))))) ( -43.40) >DroMoj_CAF1 20396 109 + 1 U-----GCC------GCCAACGCCAGUGCCGCCCUGCAUCUGCAUUUGCAUCGACUGCAGGGUGGGGGUGGUUGGCACCGAUAGCGGUGGCGUUGAUGAGACCAAAGCCUGUAGCUGUUG .-----...------((((..((((.(.(((((((((((((((....)))..)).)))))))))).).))))))))..(((((((...(((.(((.......))).)))....))))))) ( -52.10) >DroAna_CAF1 25138 111 + 1 U---GACCC------AGCACCGGAUGUCCCGCUCUGCAACUGCAUCUGCAUCGACUGCAAAGUGGGUGUGGUGGGCACUGAGAGCGGUGGCGUUGAUGAUACCAGGGCCUGGAGCUGUGG .---..(((------(((....(((((((((((((.((..(((..(..((((.(((....))).))))..)...))).))))))))).)))))).......((((...)))).)))).)) ( -48.00) >consensus U_____GCC______ACCACCGCCAGUGCCGCUCUGCAUCUGCAUUUGCAUCGACUGCAGGGUGGGCGUGGUGGGCACCGAUAGCGGUGGCGUCGAUGAGACCAAUGCCUGUAGCUGUUG ....................(((((...((((((((((..(((....))).....))))))))))...))))).......(((((...(((((.(.......).)))))....))))).. (-23.65 = -25.32 + 1.67)

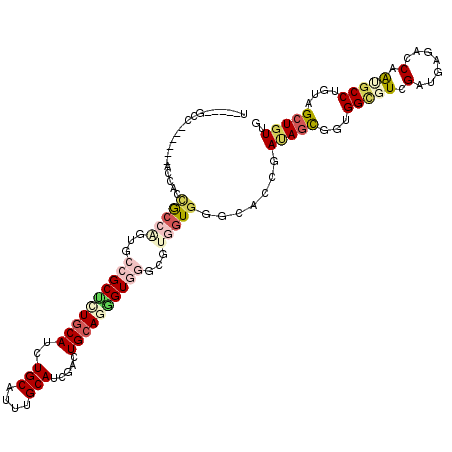
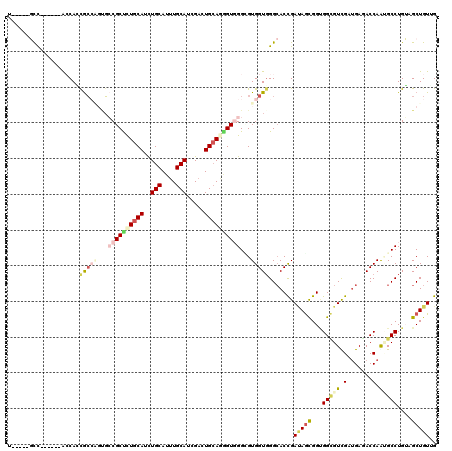
| Location | 226,400 – 226,514 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.93 |
| Mean single sequence MFE | -43.01 |
| Consensus MFE | -17.92 |
| Energy contribution | -19.17 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.42 |
| SVM decision value | 0.40 |
| SVM RNA-class probability | 0.719787 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 226400 114 - 22407834 CCACAGCUCCAGGCAUUGGUUUCAUCGACACCGCCGCUGUCGGUUCCCACCACUCCCACCCUUCAGUCUAUGCAAAUGCAGCUGCAGAGCGGCUCCGGCAGUGGU------GGGUCCGGA ..(((((....(((...(((.((...)).)))))))))))(((..(((((((((.((.............(((....)))(((((...)))))...)).))))))------))).))).. ( -44.50) >DroVir_CAF1 23139 115 - 1 CAACAGCUACAGGCACUGGUCUCCUCGACGCCGCCGCUAUCGGUGCCCACCACGCCGACGCUGCAGUCGAUGCAAAUGCAGAUGCAGAGCGCCGGUGGUGGUGGUGCCGUUGGC-----A .....((((..(((....(((.....)))(((((((((((((((((.(.....((((((......))))..))...(((....)))).))))))))))))))))))))..))))-----. ( -54.70) >DroGri_CAF1 22290 106 - 1 CUACAGCUACAGGCGCUGGUCUCAUCGACGCCUCCGCUGUCGGUGCCCACCACGCCCACCUUGCAAUCCAUGCAAAUGCAGAUGCAGGGCGCCG---GCGUUGGC------AAC-----A .....((((...((((((((..(((((((((....)).)))))))........((((...(((((.....))))).(((....)))))))))))---))))))))------...-----. ( -42.10) >DroWil_CAF1 25450 112 - 1 CAACAGCUACAGGCAUUGGUAUCAUCGACUCCACCAUUAUCAGUGCCCACCACACCGACUCUGCAAUCGAUGCAAAUGCAAUUGCAAAGCGGCACUGGCGCUGCU------GGCAAUG-- ((.((((....((((((((((................))))))))))..(((..(((.((.((((((...(((....))))))))).)))))...))).)))).)------)......-- ( -35.09) >DroMoj_CAF1 20396 109 - 1 CAACAGCUACAGGCUUUGGUCUCAUCAACGCCACCGCUAUCGGUGCCAACCACCCCCACCCUGCAGUCGAUGCAAAUGCAGAUGCAGGGCGGCACUGGCGUUGGC------GGC-----A .....((...((((....))))......(((((.(((((..((((.....)))).((.(((((((.....(((....)))..))))))).))...))))).))))------)))-----. ( -43.80) >DroAna_CAF1 25138 111 - 1 CCACAGCUCCAGGCCCUGGUAUCAUCAACGCCACCGCUCUCAGUGCCCACCACACCCACUUUGCAGUCGAUGCAGAUGCAGUUGCAGAGCGGGACAUCCGGUGCU------GGGUC---A ((.((((.((.(((..((((...))))..))).((((((((((((.....)))......((((((.....))))))......)).))))))).......)).)))------)))..---. ( -37.90) >consensus CAACAGCUACAGGCACUGGUCUCAUCGACGCCACCGCUAUCGGUGCCCACCACACCCACCCUGCAGUCGAUGCAAAUGCAGAUGCAGAGCGGCACUGGCGGUGGU______GGC_____A .....(((...(((((((((..................)))))))))........((.(((((((.....(((....)))..))))))).))....)))..................... (-17.92 = -19.17 + 1.25)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:24:37 2006