
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 16,468,367 – 16,468,593 |
| Length | 226 |
| Max. P | 0.958594 |

| Location | 16,468,367 – 16,468,460 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 81.24 |
| Mean single sequence MFE | -48.53 |
| Consensus MFE | -31.03 |
| Energy contribution | -32.78 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468367 93 - 22407834 CCAGAC---------GGUGGGUCUGUCCCAUCUGGGUGUGGUGCCUCCACACCAGCGGGGCUCCCCCUCAUCGGCGGAGGCAG---CGGCAGCCGCGCUUCAGCA ...((.---------((.(((...(((((..((((.(((((.....))))))))).))))).)))))))....((.(((((.(---(((...))))))))).)). ( -45.80) >DroSec_CAF1 93190 93 - 1 CCAGAC---------GGUGGGUCUGUCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGAUCCCCCUCAUCGGCGGAGGCAG---CGGCAGCCGCGCUUCAGCA .....(---------((((((...(((((..((((.(((((.....)))))))))..)))))....)))))))((.(((((.(---(((...))))))))).)). ( -47.10) >DroSim_CAF1 90652 93 - 1 CCAGAC---------GGUGGGUCUGUCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGAUCCCCCUCAUCGGCGGAGGCAG---CGGCAGCCGCGCUUCAGCA .....(---------((((((...(((((..((((.(((((.....)))))))))..)))))....)))))))((.(((((.(---(((...))))))))).)). ( -47.10) >DroEre_CAF1 90653 93 - 1 CCAAAC---------GGUGGGUCUCUCCCACCUGGGUGUGGUGCCACCACACCAGCGGGGCUCCCCCACAUCGGCGGAGGCGG---CCGCCGCCGCCCUUCAGCA ......---------((((((.....))))))..(((((((.....))))))).((((((...)))).....(((((.(((..---..))).))))).....)). ( -50.30) >DroYak_CAF1 95212 93 - 1 CCAAAC---------GGUGGGUCUGUCCCACUUGGGCGUGGUGCCCCCACACCAGCGGGGCUCCCCCUCAUCGGCGGAGGCGG---CCGCAGCCGCGCUUCAGCA .....(---------((((((...(((((.((.((..((((.....)))).)))).))))).....)))))))((((((((((---(....))))).)))).)). ( -43.30) >DroPer_CAF1 65558 96 - 1 UCAGGCAGCAGCGGCGGCAGCCGUGCACCACCUCGGUGUGGUGCCGC------AACGCGGCUCCCCUU---CUGCGGAGGCAGCCGCUGCCGCCGCCCUGCAACA ....((((..(((((((((((((.(((((((......))))))))).------.....((((((((..---....)).)).))))))))))))))).)))).... ( -57.60) >consensus CCAGAC_________GGUGGGUCUGUCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGCUCCCCCUCAUCGGCGGAGGCAG___CGGCAGCCGCGCUUCAGCA ...............((((((.....))))))..(((((((.....))))))).((((((...))))......((((..((.......))..))))......)). (-31.03 = -32.78 + 1.75)

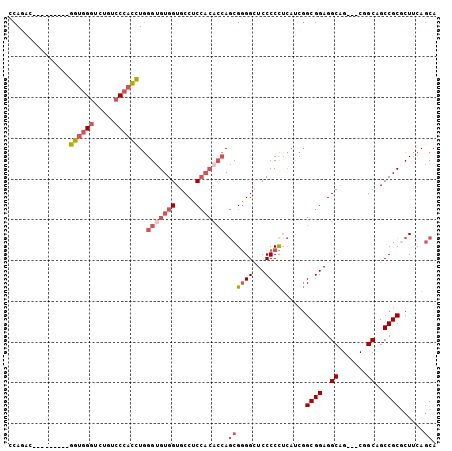
| Location | 16,468,398 – 16,468,489 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 78.18 |
| Mean single sequence MFE | -40.75 |
| Consensus MFE | -26.20 |
| Energy contribution | -27.48 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.530701 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468398 91 + 22407834 AUGAGGGGGAGCCCCGCUGGUGUGGAGGCACCACACCCAGAUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGA---GGCGGC------AUGCUGGUGGAG ....(((....)))(((..(((((..((((((((..((((((((..---........))-))))))..)))))))..---..)..)------))))..)))... ( -43.70) >DroGri_CAF1 72443 93 + 1 ---UGGGUGAGCCGUGCUGGCCGACAACAACAACGCCCAGAUGAUGCGCCGG-------ACCCGGAUGCUGAUGCGAGGAGGCGGCUGCCGCAUGUUGAUGCA- ---..(((.((((((.((((.((..........)).)))).....((((((.-------...))).)))............)))))))))((((....)))).- ( -31.20) >DroSec_CAF1 93221 91 + 1 AUGAGGGGGAUCCCCGCUGGUGUGGAGGCACCACACCCAGGUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGA---GGCGGC------AUGCUGGUGGAG ....((((...))))((..(((((..(((((((((((..((((((.---....))))))-....))).)))))))..---..)..)------))))..)).... ( -42.30) >DroSim_CAF1 90683 91 + 1 AUGAGGGGGAUCCCCGCUGGUGUGGAGGCACCACACCCAGGUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGA---GGCGGC------AUGCUGGUGGAG ....((((...))))((..(((((..(((((((((((..((((((.---....))))))-....))).)))))))..---..)..)------))))..)).... ( -42.30) >DroEre_CAF1 90684 91 + 1 AUGUGGGGGAGCCCCGCUGGUGUGGUGGCACCACACCCAGGUGGGA---GAGACCCACC-GUUUGGUGGUGGUGCGA---GGCGGC------GUGCUGGUGGAG ....(((....)))(((..(..((.((((((((((((..((((((.---....))))))-....))).)))))))..---..)).)------)..)..)))... ( -45.00) >DroYak_CAF1 95243 91 + 1 AUGAGGGGGAGCCCCGCUGGUGUGGGGGCACCACGCCCAAGUGGGA---CAGACCCACC-GUUUGGUGGUGGUGCGA---GGCGGC------GUGCUGGUGGAG ((.((.(.(..((((((....))))))(((((((..(((((((((.---....))))).-..))))..)))))))..---.....)------.).)).)).... ( -40.00) >consensus AUGAGGGGGAGCCCCGCUGGUGUGGAGGCACCACACCCAGGUGGGA___CAGACCCACC_GUCUGGUGGUGGUGCGA___GGCGGC______AUGCUGGUGGAG .....((....))((((((((......(((((((..(((((((((........)))))....))))..)))))))(......)...........)))))))).. (-26.20 = -27.48 + 1.28)



| Location | 16,468,398 – 16,468,489 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 78.18 |
| Mean single sequence MFE | -34.95 |
| Consensus MFE | -23.04 |
| Energy contribution | -24.71 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.49 |
| SVM RNA-class probability | 0.958594 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468398 91 - 22407834 CUCCACCAGCAU------GCCGCC---UCGCACCACCACCAGAC-GGUGGGUCUG---UCCCAUCUGGGUGUGGUGCCUCCACACCAGCGGGGCUCCCCCUCAU ............------...(((---((((.............-((((((....---.))))))..(((((((.....))))))).))))))).......... ( -36.70) >DroGri_CAF1 72443 93 - 1 -UGCAUCAACAUGCGGCAGCCGCCUCCUCGCAUCAGCAUCCGGGU-------CCGGCGCAUCAUCUGGGCGUUGUUGUUGUCGGCCAGCACGGCUCACCCA--- -.((((....))))((.(((((....((.((..(((((.(.(.((-------((((........)))))).).).)))))...)).))..)))))...)).--- ( -28.70) >DroSec_CAF1 93221 91 - 1 CUCCACCAGCAU------GCCGCC---UCGCACCACCACCAGAC-GGUGGGUCUG---UCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGAUCCCCCUCAU ............------....((---((((.............-((((((....---.))))))..(((((((.....))))))).))))))........... ( -35.70) >DroSim_CAF1 90683 91 - 1 CUCCACCAGCAU------GCCGCC---UCGCACCACCACCAGAC-GGUGGGUCUG---UCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGAUCCCCCUCAU ............------....((---((((.............-((((((....---.))))))..(((((((.....))))))).))))))........... ( -35.70) >DroEre_CAF1 90684 91 - 1 CUCCACCAGCAC------GCCGCC---UCGCACCACCACCAAAC-GGUGGGUCUC---UCCCACCUGGGUGUGGUGCCACCACACCAGCGGGGCUCCCCCACAU ............------...(((---((((.............-((((((....---.))))))..(((((((.....))))))).))))))).......... ( -39.70) >DroYak_CAF1 95243 91 - 1 CUCCACCAGCAC------GCCGCC---UCGCACCACCACCAAAC-GGUGGGUCUG---UCCCACUUGGGCGUGGUGCCCCCACACCAGCGGGGCUCCCCCUCAU ............------...(((---((((....(((((....-)))))....(---(((.....)))).(((((......)))))))))))).......... ( -33.20) >consensus CUCCACCAGCAU______GCCGCC___UCGCACCACCACCAGAC_GGUGGGUCUG___UCCCACCUGGGUGUGGUGCCUCCACACCAGCGGGGCUCCCCCUCAU ..................((.........((((((.((((.....((((((........))))))..))))))))))..........))(((.....))).... (-23.04 = -24.71 + 1.67)


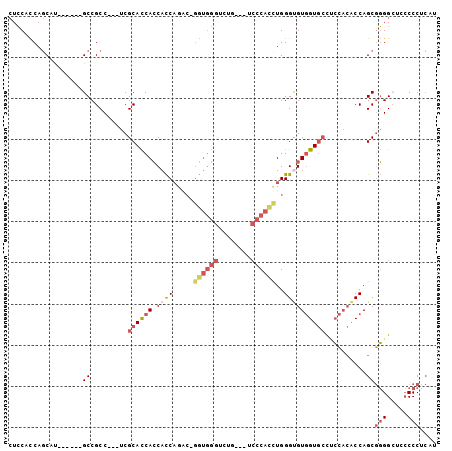
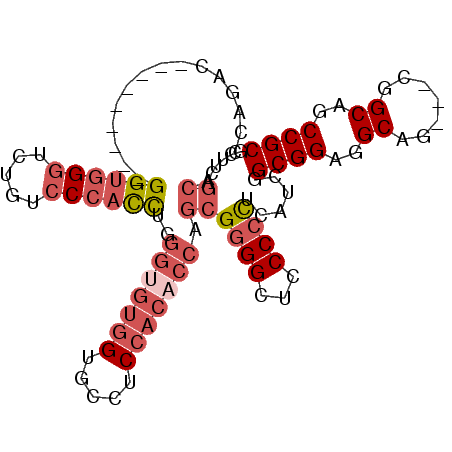
| Location | 16,468,435 – 16,468,529 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 83.15 |
| Mean single sequence MFE | -48.82 |
| Consensus MFE | -32.95 |
| Energy contribution | -34.95 |
| Covariance contribution | 2.00 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.67 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509291 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468435 94 + 22407834 CAGAUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGAGGCGGC------AUGCUGGUGGAGCUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCC (((((((..---........))-)))))(..(((((((..(((.((------((((..(.....)..)))))).))))).)))))..).(((((....))))). ( -51.50) >DroSec_CAF1 93258 94 + 1 CAGGUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGAGGCGGC------AUGCUGGUGGAGCUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCC ..((((((.---....))))))-.....(..(((((((..(((.((------((((..(.....)..)))))).))))).)))))..).(((((....))))). ( -53.20) >DroSim_CAF1 90720 94 + 1 CAGGUGGGA---CAGACCCACC-GUCUGGUGGUGGUGCGAGGCGGC------AUGCUGGUGGAGCUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCC ..((((((.---....))))))-.....(..(((((((..(((.((------((((..(.....)..)))))).))))).)))))..).(((((....))))). ( -53.20) >DroEre_CAF1 90721 94 + 1 CAGGUGGGA---GAGACCCACC-GUUUGGUGGUGGUGCGAGGCGGC------GUGCUGGUGGAGCUGGUGUGCGGCAGCUGCCGCUGCAGCCGCUGCAGCGGCC ..((((((.---....))))))-.....(..(((((((...((.((------(..(..(.....)..)..))).)).)).)))))..).(((((....))))). ( -51.40) >DroYak_CAF1 95280 94 + 1 CAAGUGGGA---CAGACCCACC-GUUUGGUGGUGGUGCGAGGCGGC------GUGCUGGUGGAGCUGGUGUGCGGCUGCUGCCGCUGCAGCCGCUGCAGCGGCC ...(((((.---....))))).-.....(..(((((((..(((.((------(..(..(.....)..)..))).))))).)))))..).(((((....))))). ( -46.50) >DroMoj_CAF1 63953 97 + 1 CAGAUGAUGUGCCUGAUG----GGCCUGAUGAUGGUGCGAAGCGGCUGCAGCAUGUUGAUGCA---AAUGAGCAGCGGCUGCGGCAGCGGCGGCAGUAGCGGCC .........((((...((----.(((.......))).))..((.(((((.(((.((((.(((.---.....))).))))))).))))).))))))......... ( -37.10) >consensus CAGGUGGGA___CAGACCCACC_GUCUGGUGGUGGUGCGAGGCGGC______AUGCUGGUGGAGCUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCC ..((((((........)))))).....((((((.(..((.(.((((........)))).)........))..).))))))((((((((((...)))))))))). (-32.95 = -34.95 + 2.00)


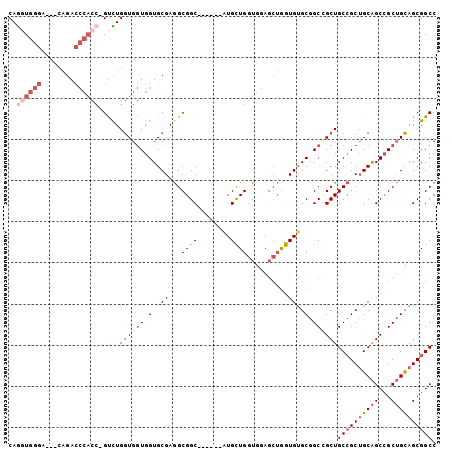
| Location | 16,468,435 – 16,468,529 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 83.15 |
| Mean single sequence MFE | -38.26 |
| Consensus MFE | -29.89 |
| Energy contribution | -31.20 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.598246 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468435 94 - 22407834 GGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAGCUCCACCAGCAU------GCCGCCUCGCACCACCACCAGAC-GGUGGGUCUG---UCCCAUCUG .((((((((((...)))))))))).(((((.((...............))..------)))))...(((...(((((....-)))))...))---)........ ( -39.26) >DroSec_CAF1 93258 94 - 1 GGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAGCUCCACCAGCAU------GCCGCCUCGCACCACCACCAGAC-GGUGGGUCUG---UCCCACCUG .((((((((((...)))))))))).(((((.(((.....(((.....))).)------)).)))..)).............-((((((....---.)))))).. ( -41.50) >DroSim_CAF1 90720 94 - 1 GGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAGCUCCACCAGCAU------GCCGCCUCGCACCACCACCAGAC-GGUGGGUCUG---UCCCACCUG .((((((((((...)))))))))).(((((.(((.....(((.....))).)------)).)))..)).............-((((((....---.)))))).. ( -41.50) >DroEre_CAF1 90721 94 - 1 GGCCGCUGCAGCGGCUGCAGCGGCAGCUGCCGCACACCAGCUCCACCAGCAC------GCCGCCUCGCACCACCACCAAAC-GGUGGGUCUC---UCCCACCUG (((.(((((.((((((((....)))))))).))).....(((.....)))..------)).))).................-((((((....---.)))))).. ( -40.20) >DroYak_CAF1 95280 94 - 1 GGCCGCUGCAGCGGCUGCAGCGGCAGCAGCCGCACACCAGCUCCACCAGCAC------GCCGCCUCGCACCACCACCAAAC-GGUGGGUCUG---UCCCACUUG (((.((....((((((((.......))))))))......(((.....)))..------)).)))..(((...(((((....-)))))...))---)........ ( -37.00) >DroMoj_CAF1 63953 97 - 1 GGCCGCUACUGCCGCCGCUGCCGCAGCCGCUGCUCAUU---UGCAUCAACAUGCUGCAGCCGCUUCGCACCAUCAUCAGGCC----CAUCAGGCACAUCAUCUG (((.((....)).)))(.(((((((((.(((((.....---.((((....)))).))))).)))..)).((.......))..----.....)))))........ ( -30.10) >consensus GGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAGCUCCACCAGCAU______GCCGCCUCGCACCACCACCAGAC_GGUGGGUCUG___UCCCACCUG ((((((....))))))((.(((((.((....))......(((.....)))........)))))...))..............((((((........)))))).. (-29.89 = -31.20 + 1.31)

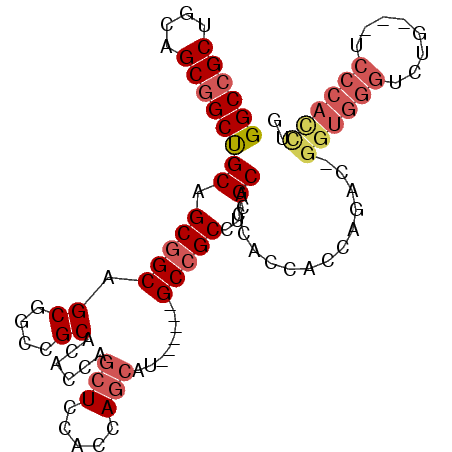
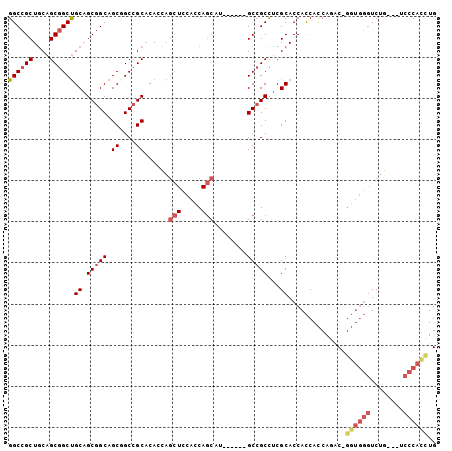
| Location | 16,468,460 – 16,468,569 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.70 |
| Mean single sequence MFE | -53.00 |
| Consensus MFE | -31.82 |
| Energy contribution | -30.72 |
| Covariance contribution | -1.11 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.708317 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468460 109 + 22407834 UGG--UGGUGCGAGGCGGC------AUGCUGGUGGAGCUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCCGCUGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCA .((--(((..((.(((.((------((((..(.....)..)))))).)))((.((((((((((...)))))))))).)).((((..((((........))))))))))..))))).. ( -56.50) >DroVir_CAF1 79715 112 + 1 UGA--UGGUGUGAGGCGACUGCAGCAUGUUGAUGCA---AAUGAGCAGCAGCUGCGGCAGCGGCGGCGGUAGCGGCCGCCGAACCCGUGGAUAAAUUAUCACGCUCCGUGUCACUCA .((--(((..((..(((.(((((((.(((((...(.---...)..))))))))))))..(((((((((((....)))))))...)))).............)))..))..))).)). ( -47.80) >DroEre_CAF1 90746 109 + 1 UGG--UGGUGCGAGGCGGC------GUGCUGGUGGAGCUGGUGUGCGGCAGCUGCCGCUGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCA .((--(((..((.((((((------(.((((.((.(((.(.((((((((....))))).))).).))).)).))))))))..)))(((((........)))))...))..))))).. ( -55.20) >DroWil_CAF1 127817 114 + 1 UGGGAUGAUGCGAUGCCGCCGCUGCAUGCUGAUGGAG---AUGAGCUGCAGCUGCAGCAGCAGCGGCUGCGGCUGCGGCCGAGCCCGUAGAUAGAUUAUCACGUUCGGUGUCACUCA .(((.(((((((.(((.((((((((.(((((.((.((---.....)).))....))))))))))))).)))..))).(((((((..((.((((...)))))))))))))))))))). ( -52.00) >DroYak_CAF1 95305 109 + 1 UGG--UGGUGCGAGGCGGC------GUGCUGGUGGAGCUGGUGUGCGGCUGCUGCCGCUGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCA .((--(((..((..(((((------(..(..(.....)..)..)))((((((.((((((((((...)))))))))).))..))))................)))..))..))))).. ( -55.70) >DroMoj_CAF1 63978 112 + 1 UGA--UGGUGCGAAGCGGCUGCAGCAUGUUGAUGCA---AAUGAGCAGCGGCUGCGGCAGCGGCGGCAGUAGCGGCCGCCGAACCCGUGGAUAGAUUAUCACGCUCCGUGUCACUCA .((--(((..((.((((((((((((.((((.((...---.)).))))...))))))))..(((((((.......)))))))....................)))).))..))).)). ( -50.80) >consensus UGG__UGGUGCGAGGCGGC______AUGCUGAUGGAG__GAUGAGCGGCAGCUGCCGCAGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCA .....(((..((..(((((........))(((((..........(((((.(((((.((.......)).))))).)))))((....)).........))))))))..))..))).... (-31.82 = -30.72 + -1.11)



| Location | 16,468,460 – 16,468,569 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 80.70 |
| Mean single sequence MFE | -45.54 |
| Consensus MFE | -33.46 |
| Energy contribution | -32.38 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468460 109 - 22407834 UGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCAGCGGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAGCUCCACCAGCAU------GCCGCCUCGCACCA--CCA ...((((..(((...(((.......(((....)))...(((((((((((.((.......)).))))))))))).)))..(((.....)))..------.)))..))))....--... ( -46.20) >DroVir_CAF1 79715 112 - 1 UGAGUGACACGGAGCGUGAUAAUUUAUCCACGGGUUCGGCGGCCGCUACCGCCGCCGCUGCCGCAGCUGCUGCUCAUU---UGCAUCAACAUGCUGCAGUCGCCUCACACCA--UCA ((((((((......((((..........))))(((.(((((((.......)))))))..)))(((((((.(((.....---.))).))....))))).)))).)))).....--... ( -42.00) >DroEre_CAF1 90746 109 - 1 UGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCGGCGGCCGCUGCAGCGGCUGCAGCGGCAGCUGCCGCACACCAGCUCCACCAGCAC------GCCGCCUCGCACCA--CCA ...((((..(((..((((.....(((.....(((((.(((((((((....)))))))).(((((....)))))...).)))))...))))))------))))..))))....--... ( -46.10) >DroWil_CAF1 127817 114 - 1 UGAGUGACACCGAACGUGAUAAUCUAUCUACGGGCUCGGCCGCAGCCGCAGCCGCUGCUGCUGCAGCUGCAGCUCAU---CUCCAUCAGCAUGCAGCGGCGGCAUCGCAUCAUCCCA (((((((.......(((((((...)))).)))((((((((....)))).))))((((((((((((((((........---......)))).)))))))))))).)))).)))..... ( -49.84) >DroYak_CAF1 95305 109 - 1 UGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCGGCGGCCGCUGCAGCGGCUGCAGCGGCAGCAGCCGCACACCAGCUCCACCAGCAC------GCCGCCUCGCACCA--CCA ...((((..(((..((((.....(((.....(((((.(((((((((....)))))))).(((((....)))))...).)))))...))))))------))))..))))....--... ( -45.80) >DroMoj_CAF1 63978 112 - 1 UGAGUGACACGGAGCGUGAUAAUCUAUCCACGGGUUCGGCGGCCGCUACUGCCGCCGCUGCCGCAGCCGCUGCUCAUU---UGCAUCAACAUGCUGCAGCCGCUUCGCACCA--UCA ((.(((...(((((((................(((((((((((.((.......)).))))))).))))(((((.....---.((((....)))).)))))))))))))))))--... ( -43.30) >consensus UGAGUGACACGGAACGUGAUAAUCUAUCCACGGGCUCGGCGGCCGCUGCAGCCGCUGCAGCGGCAGCUGCCGCACACC__CUCCACCAGCAU______GCCGCCUCGCACCA__CCA ...((((..(((..(((((((...)))).))).(((..(((((.((....)).))))).(((((....)))))..............))).........)))..))))......... (-33.46 = -32.38 + -1.08)

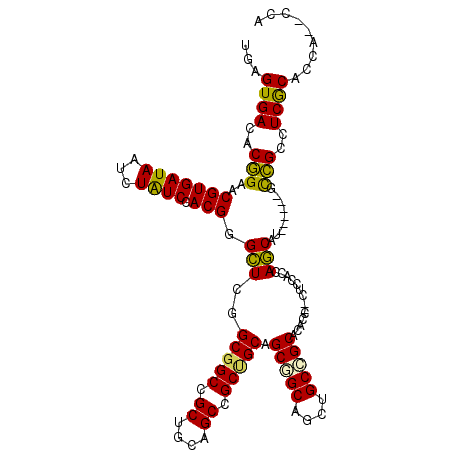

| Location | 16,468,489 – 16,468,593 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -41.80 |
| Consensus MFE | -25.93 |
| Energy contribution | -26.60 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916423 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468489 104 + 22407834 CUGGUGUGCGGCCGCUGCCGCUGCAGCCGCUGCAGCGGCCGCUGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCAAAUCUGU----CAGAAAAGACACGAAUU ..(((..(((((((((((.((.......)).)))))))))))...)))((((((((((((..((((...)))).......)))))))----)........)))).... ( -45.80) >DroVir_CAF1 79749 100 + 1 --AAUGAGCAGCAGCUGCGGCAGCGGCGGCGGUAGCGGCCGCCGAACCCGUGGAUAAAUUAUCACGCUCCGUGUCACUCAAGUCUGG----CAAAAGACAUACAAA-- --..((((..((....((((.((((((((((((....)))))))...))((((........)))))))))))))..)))).((((..----....)))).......-- ( -34.90) >DroEre_CAF1 90775 104 + 1 CUGGUGUGCGGCAGCUGCCGCUGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCAAAUCUGU----CAGGGGAGACACGAAUU ...((((.((((....((((((((((...)))))))))).))))..(((...((((((((..((((...)))).......)))))))----).)))...))))..... ( -46.80) >DroWil_CAF1 127854 101 + 1 ---AUGAGCUGCAGCUGCAGCAGCAGCGGCUGCGGCUGCGGCCGAGCCCGUAGAUAGAUUAUCACGUUCGGUGUCACUCAAGUCUGAGAGGCAGAAGAGA----AAUA ---....((((((((((((((.......))))))))))))))(((((..((.((((...))))))))))).((((.((((....)))).)))).......----.... ( -42.40) >DroYak_CAF1 95334 104 + 1 CUGGUGUGCGGCUGCUGCCGCUGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCAAAUCUGU----CAGAAAAGACACGAAUU ...((((((((((((.((((((((((...)))))))))).))..)).)))).((((((((..((((...)))).......)))))))----).......))))..... ( -43.80) >DroMoj_CAF1 64012 100 + 1 --AAUGAGCAGCGGCUGCGGCAGCGGCGGCAGUAGCGGCCGCCGAACCCGUGGAUAGAUUAUCACGCUCCGUGUCACUCAAGUCUGU----UGAAAGGCACACAAA-- --...((((.....(..(((...(((((((.......)))))))...)))..)...(.....)..)))).(((((..((((.....)----)))..))))).....-- ( -37.10) >consensus __GAUGAGCGGCAGCUGCCGCAGCAGCCGCUGCAGCGGCCGCCGAGCCCGUGGACAGAUUAUCACGUUCCGUGUCACUCAAAUCUGU____CAGAAGAGACACGAAUU ....((.(((((.(((((.((.......)).))))).)))))))....(((((........)))))....(((((.((....((((.....)))))).)))))..... (-25.93 = -26.60 + 0.67)


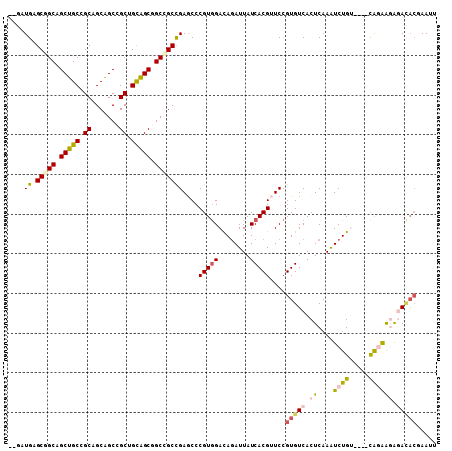
| Location | 16,468,489 – 16,468,593 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -39.18 |
| Consensus MFE | -27.17 |
| Energy contribution | -27.89 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.793729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16468489 104 - 22407834 AAUUCGUGUCUUUUCUG----ACAGAUUUGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCAGCGGCCGCUGCAGCGGCUGCAGCGGCAGCGGCCGCACACCAG ..((((((((..(((..----........))).))))))))..(((.......(((....)))...(((((((((((.((.......)).))))))))))).)))... ( -46.50) >DroVir_CAF1 79749 100 - 1 --UUUGUAUGUCUUUUG----CCAGACUUGAGUGACACGGAGCGUGAUAAUUUAUCCACGGGUUCGGCGGCCGCUACCGCCGCCGCUGCCGCAGCUGCUGCUCAUU-- --.......((((....----..)))).(((((..((.(..(((((..........))).(((.(((((((.......)))))))..)))))..)))..)))))..-- ( -31.80) >DroEre_CAF1 90775 104 - 1 AAUUCGUGUCUCCCCUG----ACAGAUUUGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCGGCGGCCGCUGCAGCGGCUGCAGCGGCAGCUGCCGCACACCAG .....((((..(((..(----(((((((.......((((...))))..))))))))...)))....((((((((....)))))))).(((((....)))))))))... ( -44.40) >DroWil_CAF1 127854 101 - 1 UAUU----UCUCUUCUGCCUCUCAGACUUGAGUGACACCGAACGUGAUAAUCUAUCUACGGGCUCGGCCGCAGCCGCAGCCGCUGCUGCUGCAGCUGCAGCUCAU--- ....----.(((.((((.....))))...)))...(((.....))).............(((((((((.(((((.((((...)))).))))).)))).)))))..--- ( -33.90) >DroYak_CAF1 95334 104 - 1 AAUUCGUGUCUUUUCUG----ACAGAUUUGAGUGACACGGAACGUGAUAAUCUGUCCACGGGCUCGGCGGCCGCUGCAGCGGCUGCAGCGGCAGCAGCCGCACACCAG ..((((((((..(((..----........))).))))))))..(((.......(((....))).((((.((((((((((...))))))))))....)))))))..... ( -43.80) >DroMoj_CAF1 64012 100 - 1 --UUUGUGUGCCUUUCA----ACAGACUUGAGUGACACGGAGCGUGAUAAUCUAUCCACGGGUUCGGCGGCCGCUACUGCCGCCGCUGCCGCAGCCGCUGCUCAUU-- --..(((((.(..((((----(.....))))).))))))(((((((..........))).(((((((((((.((.......)).))))))).))))...))))...-- ( -34.70) >consensus AAUUCGUGUCUCUUCUG____ACAGACUUGAGUGACACGGAACGUGAUAAUCUAUCCACGGGCUCGGCGGCCGCUGCAGCCGCUGCAGCGGCAGCUGCCGCACACC__ ..((((((((.(((...............))).)))))))).(((((((...)))).)))......(((((.((....)).))))).(((((....)))))....... (-27.17 = -27.89 + 0.72)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:53:35 2006