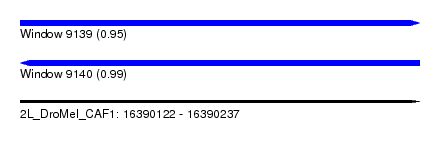
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 16,390,122 – 16,390,237 |
| Length | 115 |
| Max. P | 0.986916 |

| Location | 16,390,122 – 16,390,237 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.93 |
| Mean single sequence MFE | -30.96 |
| Consensus MFE | -24.88 |
| Energy contribution | -25.48 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.946891 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
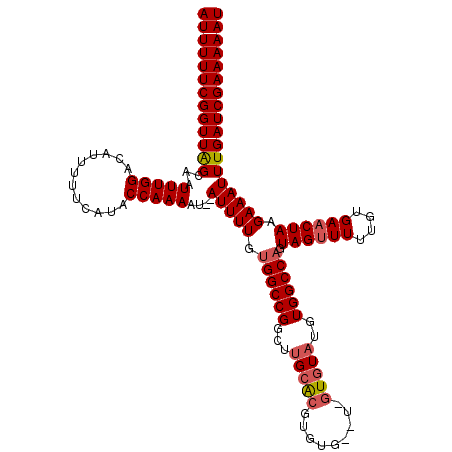
>2L_DroMel_CAF1 16390122 115 + 22407834 AUUUUUCGGUUAGCAAUUUGGACAUUUUUCAUACCAAAAU--AUUUUGUGGCCGGUUUGCACGUGUG--U-GUAUAUGUGGCCAGUAGUUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU (((((((((((((...(((((............)))))..--(((((.((((((((.(((((....)--)-))).)).)))))).((((((....)))))).)))))))))))))))))) ( -29.90) >DroSec_CAF1 14711 117 + 1 AUUUUUCGGUUAGCAAUUUGGACAUUUUUCAUACCAAAAU--AUUUUGUGGCCGGCUUGCGCGUGUGUGU-GUGUAUGUGGCCAGUAGUUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU (((((((((((((...(((((............)))))..--(((((.((((((.(.((((((......)-))))).))))))).((((((....)))))).)))))))))))))))))) ( -31.40) >DroSim_CAF1 11718 115 + 1 AUUUUUCGGUUAGCAAUUUGGACAUUUUUCAUACCAAAAU--AUUUUGUGGCCGGCUUGCACGUGUG--U-GUGUAUGUGGCCAGUAGUUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU (((((((((((((...(((((............)))))..--(((((.((((((.(.((((((....--)-))))).))))))).((((((....)))))).)))))))))))))))))) ( -31.60) >DroEre_CAF1 11506 115 + 1 AUUUUUCGGUUGGCAAUUUGGACAUUUUUCAUACCAAAAUAUAUUUUGUGGCCGGCUGGCACGUGAG-----UGUAUGUGGCCAGUAGCUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU ((((((((((..(...(((((.(((......((((((((....)))))((((((....((((....)-----)))...)))))))))......)))..)))))....)..)))))))))) ( -31.80) >DroYak_CAF1 15029 116 + 1 AUUUUUCGGUUGGCAAUUUGGACAUUUUUCAUACCAAAAU--AUUUUGUGGCCGGGGUGCACGUGAG--UGGUGUAUGUGGCCCGUAGUUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU ((((((((((..(...(((((............)))))..--((((((.(((((.(.(((((.....--..)))))).)))))).((((((....)))))).))))))..)))))))))) ( -30.10) >consensus AUUUUUCGGUUAGCAAUUUGGACAUUUUUCAUACCAAAAU__AUUUUGUGGCCGGCUUGCACGUGUG__U_GUGUAUGUGGCCAGUAGUUUUUGUGAACUAAGAAAUUUGAUCGAAAAAU (((((((((((((...(((((............)))))....(((((.((((((...(((((.........)))))..)))))).((((((....)))))).)))))))))))))))))) (-24.88 = -25.48 + 0.60)

| Location | 16,390,122 – 16,390,237 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.93 |
| Mean single sequence MFE | -20.36 |
| Consensus MFE | -17.10 |
| Energy contribution | -16.74 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.986916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
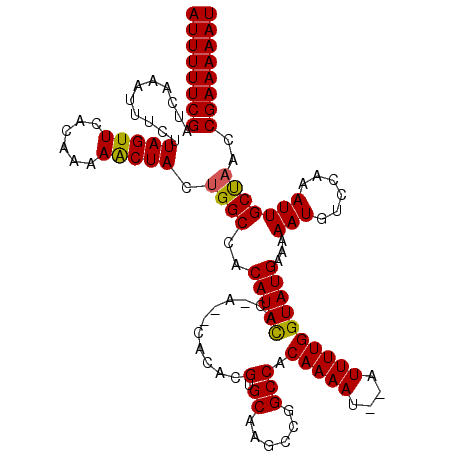
>2L_DroMel_CAF1 16390122 115 - 22407834 AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAACUACUGGCCACAUAUAC-A--CACACGUGCAAACCGGCCACAAAAU--AUUUUGGUAUGAAAAAUGUCCAAAUUGCUAACCGAAAAAU ((((((((...........(((((......))))).(((((.......(-(--(....)))......)))))......--..((((((((.....))).)))))........)))))))) ( -20.12) >DroSec_CAF1 14711 117 - 1 AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAACUACUGGCCACAUACAC-ACACACACGCGCAAGCCGGCCACAAAAU--AUUUUGGUAUGAAAAAUGUCCAAAUUGCUAACCGAAAAAU ((((((((...........(((((......))))).(((((........-........((....)).)))))......--..((((((((.....))).)))))........)))))))) ( -20.13) >DroSim_CAF1 11718 115 - 1 AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAACUACUGGCCACAUACAC-A--CACACGUGCAAGCCGGCCACAAAAU--AUUUUGGUAUGAAAAAUGUCCAAAUUGCUAACCGAAAAAU ((((((((...........(((((......))))).(((((.....(((-.--.....)))......)))))......--..((((((((.....))).)))))........)))))))) ( -20.50) >DroEre_CAF1 11506 115 - 1 AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAGCUACUGGCCACAUACA-----CUCACGUGCCAGCCGGCCACAAAAUAUAUUUUGGUAUGAAAAAUGUCCAAAUUGCCAACCGAAAAAU ((((((((...........(((((......))))).((((..(((((.-----.....(.(((....)))).(((((....))))))))))...(((......)))))))..)))))))) ( -22.00) >DroYak_CAF1 15029 116 - 1 AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAACUACGGGCCACAUACACCA--CUCACGUGCACCCCGGCCACAAAAU--AUUUUGGUAUGAAAAAUGUCCAAAUUGCCAACCGAAAAAU ((((((((.........(.(((((......))))).)((((........((--(....)))......)))).......--..((((((((.....))).)))))........)))))))) ( -19.04) >consensus AUUUUUCGAUCAAAUUUCUUAGUUCACAAAAACUACUGGCCACAUACAC_A__CACACGUGCAAGCCGGCCACAAAAU__AUUUUGGUAUGAAAAAUGUCCAAAUUGCUAACCGAAAAAU ((((((((...........(((((......))))).((((..(((((...........(.((......))).(((((....))))))))))...(((......)))))))..)))))))) (-17.10 = -16.74 + -0.36)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:18 2006