
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 16,335,482 – 16,335,749 |
| Length | 267 |
| Max. P | 0.999589 |

| Location | 16,335,482 – 16,335,592 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 68.19 |
| Mean single sequence MFE | -44.94 |
| Consensus MFE | -27.39 |
| Energy contribution | -29.66 |
| Covariance contribution | 2.27 |
| Combinations/Pair | 1.26 |
| Mean z-score | -3.03 |
| Structure conservation index | 0.61 |
| SVM decision value | 3.76 |
| SVM RNA-class probability | 0.999589 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335482 110 + 22407834 AGUCCACCGCCCGGAUCUGGCCGACCACUAGAUCCCGGACCGGGAUAUCCUCUGGGUCCCGGACAAGAAUAUCUUCUGGGUCCCUGACCAGGAUAUCCUCUGGGUCCCGG ......(((...((((((((.......)))))))).(((((((((((((((..(((.((((((..((.....)))))))).))).....))))))))))...)))))))) ( -51.60) >DroSec_CAF1 9395 101 + 1 ---------CCCGGUCCGGGAUAACCUCUGGGUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGACAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGG ---------.((((.((((((.........((((...)))).(((((((((...(((((.((((((((....)))...))))).))))))))))))))))))))..)))) ( -53.70) >DroSim_CAF1 10021 101 + 1 ---------CCCGGUCCGGGAUAACCUCUGGGUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGACAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGG ---------.((((.((((((.........((((...)))).(((((((((...(((((.((((((((....)))...))))).))))))))))))))))))))..)))) ( -53.70) >DroEre_CAF1 9889 110 + 1 UGUCCACCACCCGGACUUGGCAGCCCUCUAGAUCCCGGACUUGGCAGCCCUCUAGAUCCCGGAGCGGGAUAUCCUCUAGAUCCCGGAGCGGGAUAUCCUCUAGAUCCCGG ..........((((....(((.(((.(((.......)))...))).))).(((((((((((...((((((.........))))))...))))).....))))))..)))) ( -36.30) >DroYak_CAF1 11021 86 + 1 GGUCCACCGGCCGAA------------------------UCUGGAAGUCCUCUAGAUCCCGGAGCGGAAUAUCCUCUAGAUCCCGGGCCUGAAUAUCCCCUGGGUCCAGG (((((.((((....(------------------------((((((.....))))))).))))((.((.....)).)).......))))).........((((....)))) ( -29.40) >consensus _GUCCACC_CCCGGACCGGGAUAACCUCUAGAUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGAUAUCCUCUAGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGG ..........((((...(((..............)))((((((((((((((...(((((.((((((((......))).))))).)))))))))))))))...)))))))) (-27.39 = -29.66 + 2.27)

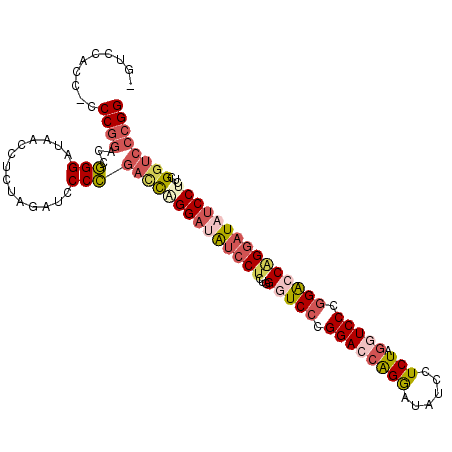
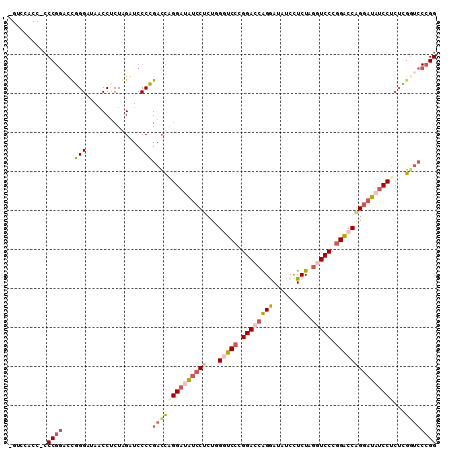
| Location | 16,335,482 – 16,335,592 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 68.19 |
| Mean single sequence MFE | -48.24 |
| Consensus MFE | -29.44 |
| Energy contribution | -32.16 |
| Covariance contribution | 2.72 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.61 |
| SVM decision value | 3.17 |
| SVM RNA-class probability | 0.998655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335482 110 - 22407834 CCGGGACCCAGAGGAUAUCCUGGUCAGGGACCCAGAAGAUAUUCUUGUCCGGGACCCAGAGGAUAUCCCGGUCCGGGAUCUAGUGGUCGGCCAGAUCCGGGCGGUGGACU ((.(((((..(.(((((((((((((..((((..((((....)))).))))..))))...))))))))))))))).)).((((.((.((((......)))).)).)))).. ( -51.60) >DroSec_CAF1 9395 101 - 1 CCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUGUCCUGGUCCGGGACCCAGAGGAUAUCCUGGUCGGGGACCCAGAGGUUAUCCCGGACCGGG--------- ((((((((...(((((((((((((((.(((((...(((....)))))))).)))))...))))))))))))))(((((((....))))..)))...)))).--------- ( -58.30) >DroSim_CAF1 10021 101 - 1 CCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUGUCCUGGUCCGGGACCCAGAGGAUAUCCUGGUCGGGGACCCAGAGGUUAUCCCGGACCGGG--------- ((((((((...(((((((((((((((.(((((...(((....)))))))).)))))...))))))))))))))(((((((....))))..)))...)))).--------- ( -58.30) >DroEre_CAF1 9889 110 - 1 CCGGGAUCUAGAGGAUAUCCCGCUCCGGGAUCUAGAGGAUAUCCCGCUCCGGGAUCUAGAGGGCUGCCAAGUCCGGGAUCUAGAGGGCUGCCAAGUCCGGGUGGUGGACA ..((((((((((.....(((((...))))))))))).....))))(((((.((((((...(((((....))))).)))))).).))))......(((((.....))))). ( -43.90) >DroYak_CAF1 11021 86 - 1 CCUGGACCCAGGGGAUAUUCAGGCCCGGGAUCUAGAGGAUAUUCCGCUCCGGGAUCUAGAGGACUUCCAGA------------------------UUCGGCCGGUGGACC .((((((((.(((..........)))))).))))).......((((((((((.((((...((....)))))------------------------)))))..)))))).. ( -29.10) >consensus CCGGGACCCAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCCAGAGGAUAUCCAGGUCCGGGACCCAGAGGUCAGCCAGGUCCGGG_GGUGGAC_ ((((((((....((((((((((((((.(((((..((.....))..))))).)))))...))))))))).))))...((((....))))........)))).......... (-29.44 = -32.16 + 2.72)

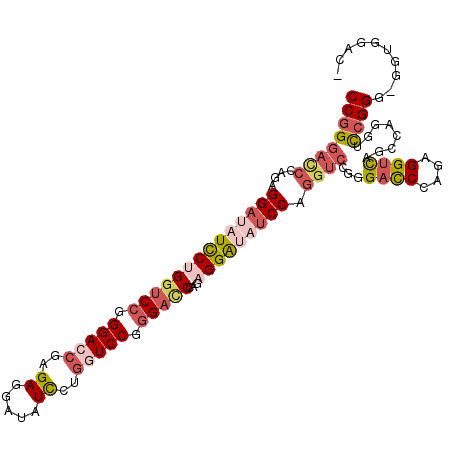
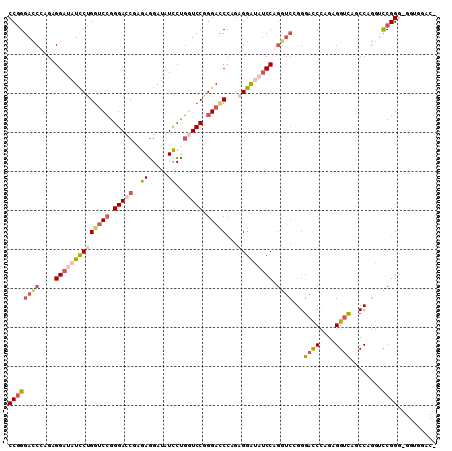
| Location | 16,335,512 – 16,335,629 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.17 |
| Mean single sequence MFE | -59.42 |
| Consensus MFE | -45.14 |
| Energy contribution | -47.74 |
| Covariance contribution | 2.60 |
| Combinations/Pair | 1.20 |
| Mean z-score | -6.00 |
| Structure conservation index | 0.76 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995440 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335512 117 + 22407834 GAUCCCGGACCGGGAUAUCCUCUGGGUCCCGGACAAGAAUAUCUUCUGGGUCCCUGACCAGGAUAUCCUCUGGGUCCCGGACCGGGAUAUCCCCUGGGUCCACGACCUGGAUAUCCU (.(((.(((((((((((((((..(((.((((((..((.....)))))))).))).....))))))))))...))))).))).)(((((((((...(((((...)))))))))))))) ( -60.80) >DroSec_CAF1 9416 117 + 1 GGUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGACAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUGGGUCCCUGACCUGGAUAUCCU (((((..((((((((((((((...(((((.((((((((....)))...))))).)))))))))))))))...))))..)))))(((((((((...(((((...)))))))))))))) ( -68.40) >DroSim_CAF1 10042 117 + 1 GGUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGACAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCUCCGGGUCCCUGACCUGGAUAUCCU (((((..((((((((((((((...(((((.((((((((....)))...))))).)))))))))))))))...))))..)))))(((((((((...(((((...)))))))))))))) ( -68.40) >DroEre_CAF1 9919 117 + 1 GAUCCCGGACUUGGCAGCCCUCUAGAUCCCGGAGCGGGAUAUCCUCUAGAUCCCGGAGCGGGAUAUCCUCUAGAUCCCGGACCUGGAUAUCCCCUAGGUCCCGGACCUGGAUAUCCC ..(((.(((((.((....))...)).))).)))..((((((((((((((((((((...))))).....))))))(((.((((((((.......)))))))).)))...))))))))) ( -52.40) >DroYak_CAF1 11036 108 + 1 ---------UCUGGAAGUCCUCUAGAUCCCGGAGCGGAAUAUCCUCUAGAUCCCGGGCCUGAAUAUCCCCUGGGUCCAGGACCUGGGUAUCCCCUGGCUCCUGGACCUGGAUAUCCC ---------((((((.....)))))).(((((((.((.....)).)).....))))).....((((((...((((((((((((.(((....))).)).))))))))))))))))... ( -47.10) >consensus GAUCCCCGACCAGGAUAUCCUCUGGGUCCCGGACCAGGAUAUCCUCUAGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCCCUGGGUCCCGGACCUGGAUAUCCU ............((((((((...(((((..(((((((((((((((...(((((.(((((.(((.....))).))))).)))))))))))))))...)))))..))))))))))))). (-45.14 = -47.74 + 2.60)


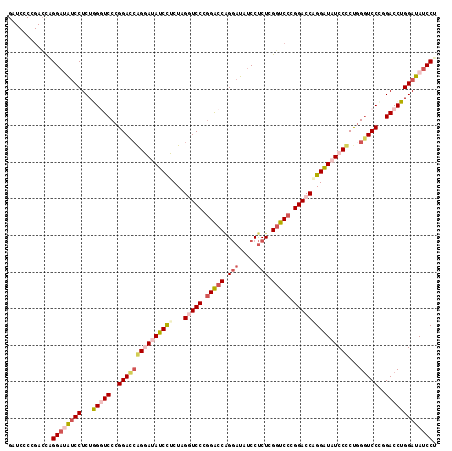
| Location | 16,335,512 – 16,335,629 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 80.17 |
| Mean single sequence MFE | -62.42 |
| Consensus MFE | -51.06 |
| Energy contribution | -51.18 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.28 |
| Mean z-score | -5.38 |
| Structure conservation index | 0.82 |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.998214 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335512 117 - 22407834 AGGAUAUCCAGGUCGUGGACCCAGGGGAUAUCCCGGUCCGGGACCCAGAGGAUAUCCUGGUCAGGGACCCAGAAGAUAUUCUUGUCCGGGACCCAGAGGAUAUCCCGGUCCGGGAUC .((....)).((((.((((((...(((((((((((((((.((((...(((((((((((((........))))..))))))))))))).)))))..).))))))))))))))).)))) ( -61.50) >DroSec_CAF1 9416 117 - 1 AGGAUAUCCAGGUCAGGGACCCAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUGUCCUGGUCCGGGACCCAGAGGAUAUCCUGGUCGGGGACC (((((((((.((((..(((((...(((((((((((((((.((((((.((.....)).)))))).)))))...)))))))))))))))..))))....)))))))))((((...)))) ( -68.90) >DroSim_CAF1 10042 117 - 1 AGGAUAUCCAGGUCAGGGACCCGGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUGUCCUGGUCCGGGACCCAGAGGAUAUCCUGGUCGGGGACC (((((((((...((.(((.((((((((((((((((((((.((((((.((.....)).)))))).)))))...)))))))))...)))))).))).)))))))))))((((...)))) ( -69.10) >DroEre_CAF1 9919 117 - 1 GGGAUAUCCAGGUCCGGGACCUAGGGGAUAUCCAGGUCCGGGAUCUAGAGGAUAUCCCGCUCCGGGAUCUAGAGGAUAUCCCGCUCCGGGAUCUAGAGGGCUGCCAAGUCCGGGAUC (((((((((((((((.((((((.(((....))))))))).))))))...)))))))))..(((....((((((.....(((((...)))))))))))(((((....))))).))).. ( -61.70) >DroYak_CAF1 11036 108 - 1 GGGAUAUCCAGGUCCAGGAGCCAGGGGAUACCCAGGUCCUGGACCCAGGGGAUAUUCAGGCCCGGGAUCUAGAGGAUAUUCCGCUCCGGGAUCUAGAGGACUUCCAGA--------- .((((((((.(((((((((.((.(((....))).)))))))))))....))))))))((((((((.....((.((.....)).))))))).)))...((....))...--------- ( -50.90) >consensus AGGAUAUCCAGGUCAGGGACCCAGGGGAUAUCCAGGUCCGGGACCCAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCCAGAGGAUAUCCAGGUCCGGGACC .((((((((.((((..(((((...(((((((((((((((.(((((..((.....))..))))).))))))...))))))))))))))..))))....))))))))............ (-51.06 = -51.18 + 0.12)


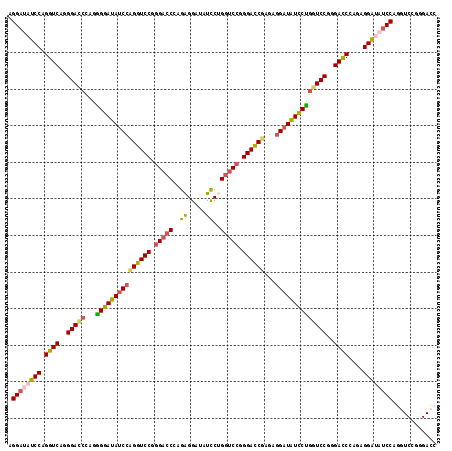
| Location | 16,335,552 – 16,335,669 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 85.56 |
| Mean single sequence MFE | -67.38 |
| Consensus MFE | -59.02 |
| Energy contribution | -61.02 |
| Covariance contribution | 2.00 |
| Combinations/Pair | 1.16 |
| Mean z-score | -7.31 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972514 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335552 117 + 22407834 AUCUUCUGGGUCCCUGACCAGGAUAUCCUCUGGGUCCCGGACCGGGAUAUCCCCUGGGUCCACGACCUGGAUAUCCUCUGGGUCCGGGUCCUGGAUAUCCUCUGGGUCCGGGACCUG .......(((((((.(((((((((((((...(((.(((((((((((((((((...(((((...))))))))))))))...)))))))).))))))))))))...)))).))))))). ( -74.30) >DroSec_CAF1 9456 117 + 1 AUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCUCUGGGUCCCUGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUG ...(((((((.((((((...((((((((....((((((((((((((((((((...(((((...))))))))))))))...))))))))))).)))))))))))))).)))))))... ( -78.30) >DroSim_CAF1 10082 117 + 1 AUCCUCUCGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCUCCGGGUCCCUGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUG ...(((((((.((((((...((((((((....((((((((((((((((((((...(((((...))))))))))))))...))))))))))).)))))))))))))).)))))))... ( -78.30) >DroEre_CAF1 9959 117 + 1 AUCCUCUAGAUCCCGGAGCGGGAUAUCCUCUAGAUCCCGGACCUGGAUAUCCCCUAGGUCCCGGACCUGGAUAUCCCCUGGGUCCCGGACCUGAAUAUCUCCUGUAUCCCGGACCUG ......(((.(((.(((((((((.((..((.((.(((.((((((((((((((...(((((...)))))))))))))...)))))).))).))))..)).)))))).))).))).))) ( -50.30) >DroYak_CAF1 11067 117 + 1 AUCCUCUAGAUCCCGGGCCUGAAUAUCCCCUGGGUCCAGGACCUGGGUAUCCCCUGGCUCCUGGACCUGGAUAUCCCCUGGGCCCUGAACCUGGAUAUCCCCUCGGUCCCGGACCUG ............(((((.((((.........((((((((((((.(((....))).)).))))))))))((((((((...((........)).))))))))..)))).)))))..... ( -55.70) >consensus AUCCUCUAGGUCCCGGACCAGGAUAUCCUCUCGGUCCCGGACCAGGAUAUCCCCUGGGUCCCGGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGACCUG ........(.((((((((((((((((((....((((((((((((((((((((...(((((...))))))))))))))...))))))))))).)))))))))...))))))))).).. (-59.02 = -61.02 + 2.00)


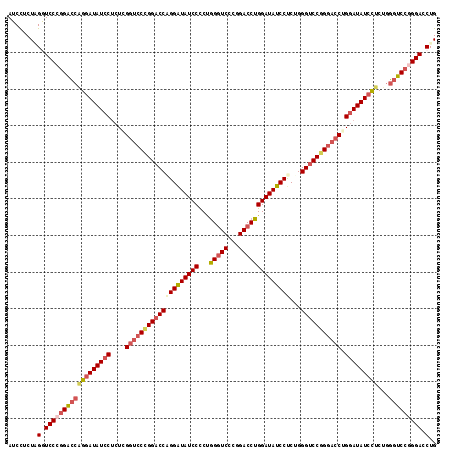
| Location | 16,335,552 – 16,335,669 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 85.56 |
| Mean single sequence MFE | -69.58 |
| Consensus MFE | -61.82 |
| Energy contribution | -63.26 |
| Covariance contribution | 1.44 |
| Combinations/Pair | 1.19 |
| Mean z-score | -7.12 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.86 |
| SVM RNA-class probability | 0.980268 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335552 117 - 22407834 CAGGUCCCGGACCCAGAGGAUAUCCAGGACCCGGACCCAGAGGAUAUCCAGGUCGUGGACCCAGGGGAUAUCCCGGUCCGGGACCCAGAGGAUAUCCUGGUCAGGGACCCAGAAGAU ..((((((.((((...(((((((((.((.((((((((..(.((((((((.((((...))))....))))))))))))))))).))....))))))))))))).))))))........ ( -67.30) >DroSec_CAF1 9456 117 - 1 CAGCUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCAGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAU ....(((((((((...(((((((((.(((((((((((...(((((((((.((((...))))....))))))))))))))))))))....))))))))))))))))))((....)).. ( -77.60) >DroSim_CAF1 10082 117 - 1 CAGCUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCGGAGGAUAUCCUGGUCCGGGACCGAGAGGAUAUCCUGGUCCGGGACCGAGAGGAU ....(((((((((...(((((((((.(((((((((((...(((((((((.((((...))))....))))))))))))))))))))....))))))))))))))))))((....)).. ( -77.60) >DroEre_CAF1 9959 117 - 1 CAGGUCCGGGAUACAGGAGAUAUUCAGGUCCGGGACCCAGGGGAUAUCCAGGUCCGGGACCUAGGGGAUAUCCAGGUCCGGGAUCUAGAGGAUAUCCCGCUCCGGGAUCUAGAGGAU .((((((.(((....((.(((((((((((((.(((((....(((((((((((((...)))))...)))))))).))))).))))))...)))))))))..))).))))))....... ( -60.10) >DroYak_CAF1 11067 117 - 1 CAGGUCCGGGACCGAGGGGAUAUCCAGGUUCAGGGCCCAGGGGAUAUCCAGGUCCAGGAGCCAGGGGAUACCCAGGUCCUGGACCCAGGGGAUAUUCAGGCCCGGGAUCUAGAGGAU .((((((.(((((..(((....))).))))).(((((....((((((((.(((((((((.((.(((....))).)))))))))))....)))))))).))))).))))))....... ( -65.30) >consensus CAGGUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCAGGGGAUAUCCAGGUCCGGGACCCAGAGGAUAUCCUGGUCCGGGACCGAGAGGAU ..(((((((((((...(((((((((.(((((((((((....((((((((.((((...))))....)))))))).)))))))))))....))))))))))))))))))))........ (-61.82 = -63.26 + 1.44)


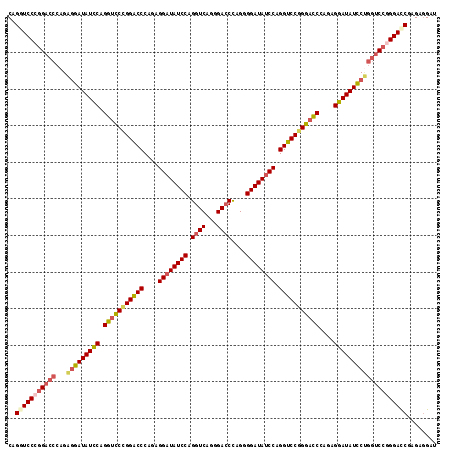
| Location | 16,335,592 – 16,335,709 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 85.30 |
| Mean single sequence MFE | -55.16 |
| Consensus MFE | -39.22 |
| Energy contribution | -40.70 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.21 |
| Mean z-score | -4.34 |
| Structure conservation index | 0.71 |
| SVM decision value | 2.86 |
| SVM RNA-class probability | 0.997448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335592 117 + 22407834 ACCGGGAUAUCCCCUGGGUCCACGACCUGGAUAUCCUCUGGGUCCGGGUCCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCAGGAACUGAAUAUUGUUGGCCUGG .(((((......(((((..(((.((...((((((((...((((((.((.((..((.....))..)).)).)))))))))))))))))))..)))))(((........)))..))))) ( -60.60) >DroSec_CAF1 9496 117 + 1 ACCAGGAUAUCCUCUGGGUCCCUGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUGGAUAUCCCCUGGGUCCCGGAACUGAGUAUUCUUGGCCUGG ...(((((((((...(((((((.((((..((.....))..)))).))))))))))))))))(..(((((((((.(..(.......)..).))))))).(..((....))..)))..) ( -63.20) >DroSim_CAF1 10122 117 + 1 ACCAGGAUAUCCUCCGGGUCCCUGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUGGAUAUCCCCUGGGUCCCGGAACUGAGUAUUGUUGGCCUGG .(((((......((((((((((.((((..((.....))..)))).))))))))))....(((.((.(((((((.(..(.......)..).))))))).))))).........))))) ( -62.60) >DroEre_CAF1 9999 117 + 1 ACCUGGAUAUCCCCUAGGUCCCGGACCUGGAUAUCCCCUGGGUCCCGGACCUGAAUAUCUCCUGUAUCCCGGACCUGGAUAUCCCCUGGAUCCCGCACUUGAAUAUCGUUGGCCUUG .((.((((((((..(((((((.(((((..(.......)..))))).)))))))......(((........)))...))))))))...)).....((...((.....))...)).... ( -42.40) >DroYak_CAF1 11107 117 + 1 ACCUGGGUAUCCCCUGGCUCCUGGACCUGGAUAUCCCCUGGGCCCUGAACCUGGAUAUCCCCUCGGUCCCGGACCUGGAUAUCCCCUUGGUCCCAAAUCUGAAUAUCGGUGGCCUUG ....((((((((...((.(((.(((((.((((((((...((........)).))))))))....))))).))))).))))))))....((((((.............)).))))... ( -47.02) >consensus ACCAGGAUAUCCCCUGGGUCCCGGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCCCUGGGUCCCGGAACUGAAUAUUGUUGGCCUGG .((.((((((((...(((((...)))))))))))))((((((((((((....((((((((((((....))))....)))))))))))))).)))))).............))..... (-39.22 = -40.70 + 1.48)

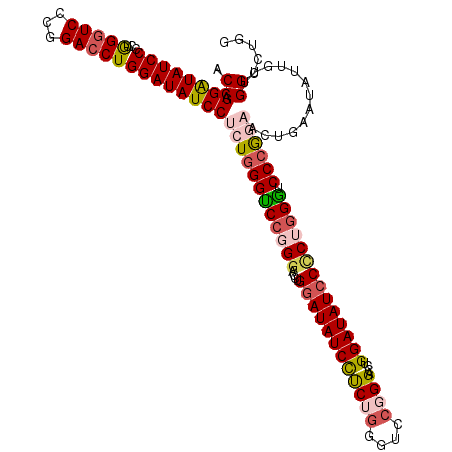
| Location | 16,335,592 – 16,335,709 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 85.30 |
| Mean single sequence MFE | -49.92 |
| Consensus MFE | -37.98 |
| Energy contribution | -37.90 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.27 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.76 |
| SVM decision value | 2.43 |
| SVM RNA-class probability | 0.993904 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335592 117 - 22407834 CCAGGCCAACAAUAUUCAGUUCCUGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGACCCGGACCCAGAGGAUAUCCAGGUCGUGGACCCAGGGGAUAUCCCGGU (((((..(((........))))))))..((.(.((((((((.(((((..((((....((((((((.((........))...)))))))).))))..)))))....))))))))))). ( -48.10) >DroSec_CAF1 9496 117 - 1 CCAGGCCAAGAAUACUCAGUUCCGGGACCCAGGGGAUAUCCAGCUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCAGAGGAUAUCCUGGU ((((..........(((.(.(((((((.(..(((....))).).))))))).)..)))(((((((.((((((.((((..((.....))..)))).))))))....))))))))))). ( -50.10) >DroSim_CAF1 10122 117 - 1 CCAGGCCAACAAUACUCAGUUCCGGGACCCAGGGGAUAUCCAGCUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCGGAGGAUAUCCUGGU (((((((.((........))((((((.(((..((((........)))).((((....((((((((.((((...))))....)))))))).)))).))).))))))))....))))). ( -50.20) >DroEre_CAF1 9999 117 - 1 CAAGGCCAACGAUAUUCAAGUGCGGGAUCCAGGGGAUAUCCAGGUCCGGGAUACAGGAGAUAUUCAGGUCCGGGACCCAGGGGAUAUCCAGGUCCGGGACCUAGGGGAUAUCCAGGU ....(((...(((((((..((.(.(((((....((((((((.(((((.((((...(((....)))..)))).)))))....)))))))).))))).).)).....)))))))..))) ( -49.30) >DroYak_CAF1 11107 117 - 1 CAAGGCCACCGAUAUUCAGAUUUGGGACCAAGGGGAUAUCCAGGUCCGGGACCGAGGGGAUAUCCAGGUUCAGGGCCCAGGGGAUAUCCAGGUCCAGGAGCCAGGGGAUACCCAGGU ...(((..(((.....).......(((((....((((((((.(((((.(((((..(((....))).))))).)))))....)))))))).))))).)).))).(((....))).... ( -51.90) >consensus CCAGGCCAACAAUAUUCAGUUCCGGGACCCAGGGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCCCGGACCCAGAGGAUAUCCAGGUCAGGGACCCAGGGGAUAUCCAGGU ....(((.............(((..((((....((((((((.(((((.(((((..((.....))..))))).)))))....)))))))).))))..))).....(((....)))))) (-37.98 = -37.90 + -0.08)


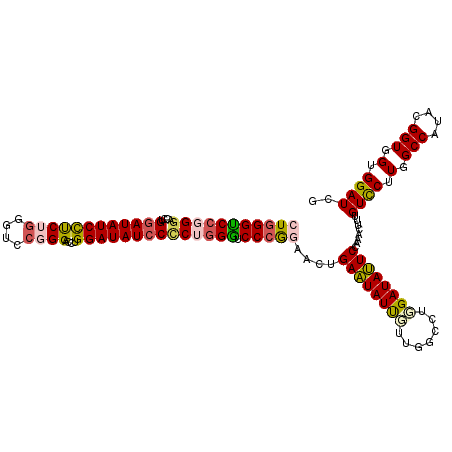
| Location | 16,335,629 – 16,335,749 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.58 |
| Mean single sequence MFE | -46.99 |
| Consensus MFE | -36.00 |
| Energy contribution | -36.56 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.740722 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16335629 120 + 22407834 CUGGGUCCGGGUCCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCAGGAACUGAAUAUUGUUGGCCUGGAUAUUCCCAACUUUGUCCUUGGCCAUACGGUGGUGGAUCG ((((((((((((((((((...((....))))))))))))))))((((...))))))))((.((...(((((((((((.(((((..........)))))..))))).))))))..)).)). ( -53.00) >DroSec_CAF1 9533 120 + 1 CUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUGGAUAUCCCCUGGGUCCCGGAACUGAGUAUUCUUGGCCUGGAUACUCCAAACUUUGUCCUUGGCCAUACGGUGGUGGAUCG ..((.((((((((((((((((((((((....))))....))))))))...)))))))))).))((((((((.......))))))))........((((.(.(((....))).).)))).. ( -52.20) >DroSim_CAF1 10159 120 + 1 CUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGAGCUGGAUAUCCCCUGGGUCCCGGAACUGAGUAUUGUUGGCCUGGAUACUCCAAACUUUGUCCUUGGCCAUACGGUGGUGGAUCG ((.((((((((((((((((((((((((....))))....))))))))...))))))))).))).))(((((((((((.(((((..........)))))..))))).))))))........ ( -54.50) >DroEre_CAF1 10036 120 + 1 CUGGGUCCCGGACCUGAAUAUCUCCUGUAUCCCGGACCUGGAUAUCCCCUGGAUCCCGCACUUGAAUAUCGUUGGCCUUGAUAUUCCGCACUACGUUCUUGGCCGUACGGUGGUGGAUCG (..(((((.(((...((.....)).....))).)))))..)..........(((((.((.......(((((((((((..((...............))..))))).))))))))))))). ( -35.37) >DroYak_CAF1 11144 120 + 1 CUGGGCCCUGAACCUGGAUAUCCCCUCGGUCCCGGACCUGGAUAUCCCCUUGGUCCCAAAUCUGAAUAUCGGUGGCCUUGAUAUUCCGCACUAUGUCCUUGGCCGUACGGUGGUGGAUCG .((((.((.......((((((((....((((...)))).))))))))....)).)))).((((...(((((..((((..(((((........)))))...))))...)))))..)))).. ( -39.90) >consensus CUGGGUCCGGGACCUGGAUAUCCUCUGGGUCCGGGACCUGGAUAUCCCCUGGGUCCCGGAACUGAAUAUUGUUGGCCUGGAUAUUCCAAACUUUGUCCUUGGCCAUACGGUGGUGGAUCG (((((((((((....((((((((((((....))))....)))))))))))))).)))))....((((((((.......))))))))........((((.(.(((....))).).)))).. (-36.00 = -36.56 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:51:23 2006