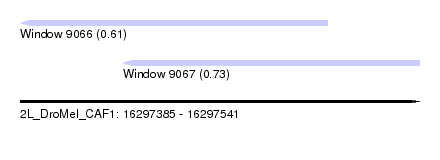
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 16,297,385 – 16,297,541 |
| Length | 156 |
| Max. P | 0.731346 |

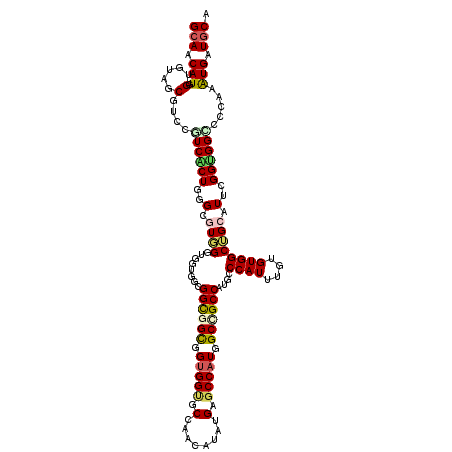
| Location | 16,297,385 – 16,297,505 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.00 |
| Mean single sequence MFE | -55.28 |
| Consensus MFE | -29.27 |
| Energy contribution | -29.42 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.609219 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16297385 120 - 22407834 GCAGCAUAGUGUAACGUCCUUCACUUGGCGUUGGCGGUGGAGGCGGCGGCGGUGCCAACAUGUGAGCCAUUGCAGCCAUUCCCAUUUGUGUGGCGGGAUUCGGUGGGCCGAGAUGAUGCA (((.(((.......((((...(((.(((...((((.(..(.(((((((....))))..(....).))).)..).))))...)))...))).))))...(((((....)))))))).))). ( -40.90) >DroVir_CAF1 12627 120 - 1 GCAACAUGGUGUAGCGACCGUCGCUGGGCGUGGGUGGUGGCGGCGGUGGUGGUGCCAGCAUAUGUGCCAUUGCCGCCAUGCCCAUUUGGGUGGCUGCAUUAGGCGGCGCCAAAUGGUGCA ((....(((((((((.((((((....)))(((((((.(((((((((((((.(((....)))....))))))))))))))))))))...))).))))))))).)).(((((....))))). ( -67.00) >DroGri_CAF1 12125 120 - 1 GCAACAUGGUGUAGCGGCCGUCACUGGGUGUAGGGGGUGGCGGUGGUGGUGGAGCCAACAUAUGUGCCAUGGCUGCCAUUCCCAUUUGUGUGGCAGCAUUUGGUGGCGCCAAAUGAUGCA (((.(((.(.....)(((.(((((..(((((.((((((((((((.((((((.(.........).)))))).))))))))))))...((.....)))))))..))))))))..))).))). ( -55.40) >DroMoj_CAF1 12301 120 - 1 GCAACAUUGUGUAACGCCCAUCGCUGGGUGUGGGUGGAGGCGGUGGUGGUGGUGCCAACAUAUGCGCCAUGGCCGCCAUGCCCAUUUGGGUGGCUGCAUUCGGUGGCGCCAAAUGAUGCA (((.((((((((.(((((((....)))))))(((((.((.(((((((.(((((((........))))))).))))))..((((....))))).)).)))))....))))..)))).))). ( -59.20) >DroAna_CAF1 11482 120 - 1 GCAGCAUCGUAUAGCGUCCUUCGCUAGGGGUUGGCGGAGGAGGUGGCGGUGGCGCCAACAUAUGAGCCAUUGCCGCCAUGCCCAUUUGUGUGGCGGGAUUGGGAGGACCCAGGUGGUGCA (((.((((.....((.(((((((((((...))))))))))).))(((((((((..((.....)).)))))))))(((((((......))))))).....((((....)))))))).))). ( -58.80) >DroPer_CAF1 11828 120 - 1 GGAGCAUUGAGUAGCGUCCCUCACUGGGCGUGGGUGGUGGCGGCGGCGGUGGUGCUAACAUGUGAGCCAUGGCGGCCAUUCCCAUUUGUGUGGCUGCAUUUGGUGGACCCAGAUGAUGCA ...(((((..(((((...((.....))(((..(((((..(.(((.((.(((((((......))..))))).)).))).)..)))))..))).)))))((((((.....))))))))))). ( -50.40) >consensus GCAACAUGGUGUAGCGUCCGUCACUGGGCGUGGGUGGUGGCGGCGGCGGUGGUGCCAACAUAUGAGCCAUGGCCGCCAUGCCCAUUUGUGUGGCUGCAUUCGGUGGCCCCAAAUGAUGCA (((.(((.(.....)....((((((..(.((((........((((((.(((((.(........).))))).))))))....((((....)))))))).)..)))))).....))).))). (-29.27 = -29.42 + 0.14)

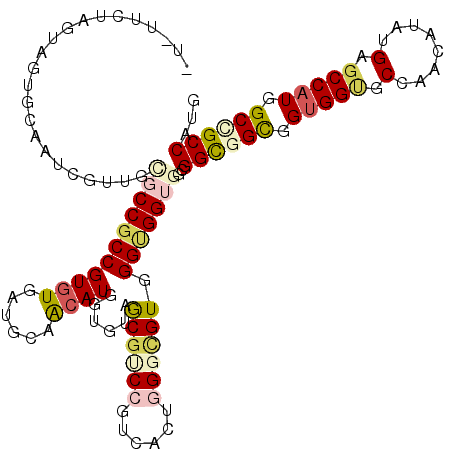
| Location | 16,297,425 – 16,297,541 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.39 |
| Mean single sequence MFE | -47.42 |
| Consensus MFE | -31.52 |
| Energy contribution | -32.33 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.731346 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 16297425 116 - 22407834 UA-CUCUAAUAAUGUAGUCUUUGUGCCGCCGUGUGGUGCAGCAUAGUGUAACGUCCUUCACUUGGCGUUGGCGGUGGAGGCGGCGGCGGUGCCAACAUGUGAGCCAUUGCAGCCAUU ..-.............((((((..((((((((((......)))).....((((((........))))))))))))))))))(((.(((((((((.....)).).)))))).)))... ( -38.00) >DroVir_CAF1 12667 115 - 1 -U-UUCUAGUAGUGCAAACGUUGCGCCGCCGUGUGAUGCAACAUGGUGUAGCGACCGUCGCUGGGCGUGGGUGGUGGCGGCGGUGGUGGUGCCAGCAUAUGUGCCAUUGCCGCCAUG -.-..........((..((.(..(((((((((((......))))))).(((((.....)))))))))..)))....))(((((..((((..(........)..))))..)))))... ( -54.70) >DroGri_CAF1 12165 115 - 1 -U-UUCUAGUAGUGUAACCGUUGCGCCGCCGUGUGGUGCAACAUGGUGUAGCGGCCGUCACUGGGUGUAGGGGGUGGCGGUGGUGGUGGAGCCAACAUAUGUGCCAUGGCUGCCAUU -(-..(((.(((((..((.(((((..((((((((......))))))))..))))).)))))))....)))..)((((((((.((((((.(.........).)))))).)))))))). ( -47.00) >DroMoj_CAF1 12341 115 - 1 -U-UUUUAGUAGUGCAGUCGCUGUGCCGCCGUAUGGUGCAACAUUGUGUAACGCCCAUCGCUGGGUGUGGGUGGAGGCGGUGGUGGUGGUGCCAACAUAUGCGCCAUGGCCGCCAUG -.-................(((...(((((.((..(((...)))..))..(((((((....))))))).))))).)))((((((.(((((((........))))))).))))))... ( -50.20) >DroAna_CAF1 11522 116 - 1 UA-GUUUAGUAAUGUAAUCUCUGAGCCGCCGUGUGAUGCAGCAUCGUAUAGCGUCCUUCGCUAGGGGUUGGCGGAGGAGGUGGCGGUGGCGCCAACAUAUGAGCCAUUGCCGCCAUG ..-((((((...........))))))(((..(((((((...)))))))..)))(((((((((((...)))))))))))((((((((((((..((.....)).))))))))))))... ( -50.10) >DroPer_CAF1 11868 117 - 1 UCCAUUUAGUAGUGCAACCUUUGGGCCGCCGUGUGAUGGAGCAUUGAGUAGCGUCCCUCACUGGGCGUGGGUGGUGGCGGCGGCGGUGGUGCUAACAUGUGAGCCAUGGCGGCCAUU .......................(((((((((((((.(((((........)).))).))))..(((..(.(((.(((((.((....)).))))).))).)..))))))))))))... ( -44.50) >consensus _U_UUCUAGUAGUGCAAUCGUUGCGCCGCCGUGUGAUGCAACAUGGUGUAGCGUCCGUCACUGGGCGUGGGUGGUGGCGGCGGCGGUGGUGCCAACAUAUGAGCCAUGGCCGCCAUG .......................(((((((((((......))))......((((((......)))))).)))))))..((((((.(((((.(........).))))).))))))... (-31.52 = -32.33 + 0.81)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:51:06 2006