| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 16,043,949 – 16,044,069 |
| Length | 120 |
| Max. P | 0.998448 |

| Location | 16,043,949 – 16,044,069 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.56 |
| Mean single sequence MFE | -54.55 |
| Consensus MFE | -44.85 |
| Energy contribution | -45.16 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.99 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.942571 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
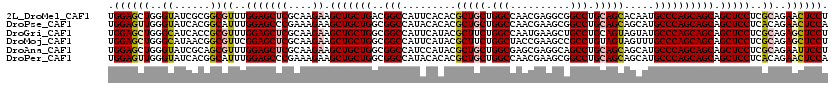
>2L_DroMel_CAF1 16043949 120 + 22407834 UGGAGCUGGGUAUCGCGGCGUUUGGAGCUCGCAAGAAGCUGCUGACGGCCAUUCACACGCUGCUGGCCAACGAGGCGGCCUGCAGCACAAUGCCCAGCAGCAGCUCCUCGCAGAACUCCU .((((((((((((...((((((.(.(((((....).)))).).))).)))........(((((.((((........)))).)))))...))))))))).((........)).....))). ( -54.90) >DroPse_CAF1 252898 120 + 1 UGGAGUUGGGUAUCACGGCAUUUGGAGCCCGAAAGAAGCUGCUGGCGGCCAUACACACGCUGCUGGCCAACGAAGCGGCCUGCAGCAGCAUGCCCAGCAGCAGCUCCUCACAGAACUCCA .((((((..((......))....(((((.(....)..((((((((((......)....(((((.((((........)))).))))).....).)))))))).)))))......)))))). ( -52.40) >DroGri_CAF1 207339 120 + 1 UGGAGCUGGGCAUCACCGCGUUUGGAGCUCGCAAGAAGCUGCUGGCGGCCAUUCAUACGCUUCUGGCCAAUGAAGCUGCCUGCAGUAGUAUGCCCAGCAGCAGCUCCUCGCAGAGCUCCU .((((((((......))(((...(((((((....)).((((((((((((((............))))).(((..((((....))))..)))).)))))))).))))).)))..)))))). ( -58.80) >DroMoj_CAF1 228635 120 + 1 UGGAGCUGGGCAUAACGGCGUUCGGAGCUCGCAAGAAGCUGCUGGCGGCCAUUCAUACGCUUCUGGCUACCGAAGCCGCCUGUAGUAGUUUGCCCAGCAGCAGCUCCUCGCAGAGCUCCU .(((((((((((.((((((.((((((((((....).))))((..(.(((.........))).)..))..))))))))((.....)).)))))))))))....((((......))))))). ( -52.20) >DroAna_CAF1 153261 120 + 1 UGGAGCUGGGUAUCGCAGCGUUUGGAGCUCGCAAGAAGCUGCUGGCGGCCAUCCAUACGCUGCUGGCGAGCGAGGCAGCCUGCAGCAGCAUGCCCAGCAGCAGCUCCUCGCAGAAUUCCU .(((((((.(...).))))((..(((((((....)).(((((((((((....))....(((((((.((.((......)).)))))))))..).)))))))).)))))..)).....))). ( -56.60) >DroPer_CAF1 282297 120 + 1 UGGAGUUGGGUAUCACGGCAUUUGGAGCCCGAAAGAAGCUGCUGGCGGCCAUACACACGCUGCUGGCCAACGAAGCGGCCUGCAGCAGCAUGCCCAGCAGCAGCUCCUCACAGAACUCCA .((((((..((......))....(((((.(....)..((((((((((......)....(((((.((((........)))).))))).....).)))))))).)))))......)))))). ( -52.40) >consensus UGGAGCUGGGUAUCACGGCGUUUGGAGCUCGCAAGAAGCUGCUGGCGGCCAUUCACACGCUGCUGGCCAACGAAGCGGCCUGCAGCAGCAUGCCCAGCAGCAGCUCCUCGCAGAACUCCU .((((((..((......))((..(((((((....)).(((((((..(((.........(((((.(((..........))).))))).....)))))))))).)))))..))..)))))). (-44.85 = -45.16 + 0.31)

| Location | 16,043,949 – 16,044,069 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.56 |
| Mean single sequence MFE | -61.50 |
| Consensus MFE | -53.89 |
| Energy contribution | -54.50 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.10 |
| Mean z-score | -4.26 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.10 |
| SVM RNA-class probability | 0.998448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
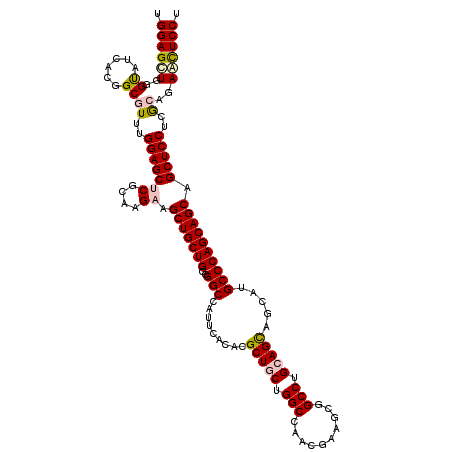
>2L_DroMel_CAF1 16043949 120 - 22407834 AGGAGUUCUGCGAGGAGCUGCUGCUGGGCAUUGUGCUGCAGGCCGCCUCGUUGGCCAGCAGCGUGUGAAUGGCCGUCAGCAGCUUCUUGCGAGCUCCAAACGCCGCGAUACCCAGCUCCA .(((((((.((((((((((((((.(((.((((.((((((.(((((......))))).))))))....)))).))).))))))))))))))))))))).......((........)).... ( -66.20) >DroPse_CAF1 252898 120 - 1 UGGAGUUCUGUGAGGAGCUGCUGCUGGGCAUGCUGCUGCAGGCCGCUUCGUUGGCCAGCAGCGUGUGUAUGGCCGCCAGCAGCUUCUUUCGGGCUCCAAAUGCCGUGAUACCCAACUCCA .((((((..((((((((((((((.(((.(((((((((((.(((((......))))).))))))...))))).))).))))))))))).((((((.......))).))))))..)))))). ( -63.30) >DroGri_CAF1 207339 120 - 1 AGGAGCUCUGCGAGGAGCUGCUGCUGGGCAUACUACUGCAGGCAGCUUCAUUGGCCAGAAGCGUAUGAAUGGCCGCCAGCAGCUUCUUGCGAGCUCCAAACGCGGUGAUGCCCAGCUCCA .(((((((.((((((((((((((.(((.(((.((((((....))(((((........)))))))).).))).))).)))))))))))))))))))))....((((......)).)).... ( -59.30) >DroMoj_CAF1 228635 120 - 1 AGGAGCUCUGCGAGGAGCUGCUGCUGGGCAAACUACUACAGGCGGCUUCGGUAGCCAGAAGCGUAUGAAUGGCCGCCAGCAGCUUCUUGCGAGCUCCGAACGCCGUUAUGCCCAGCUCCA .(((((((.(((((((((((((..(((.....))).....((((((((((...((.....))...)))..)))))))))))))))))))))))))))....................... ( -56.40) >DroAna_CAF1 153261 120 - 1 AGGAAUUCUGCGAGGAGCUGCUGCUGGGCAUGCUGCUGCAGGCUGCCUCGCUCGCCAGCAGCGUAUGGAUGGCCGCCAGCAGCUUCUUGCGAGCUCCAAACGCUGCGAUACCCAGCUCCA .(((.(((.((((((((((((((.(((.(((.(((((((.(((.((...))..))).))))))...).))).))).))))))))))))))))).)))....((((.(....))))).... ( -60.50) >DroPer_CAF1 282297 120 - 1 UGGAGUUCUGUGAGGAGCUGCUGCUGGGCAUGCUGCUGCAGGCCGCUUCGUUGGCCAGCAGCGUGUGUAUGGCCGCCAGCAGCUUCUUUCGGGCUCCAAAUGCCGUGAUACCCAACUCCA .((((((..((((((((((((((.(((.(((((((((((.(((((......))))).))))))...))))).))).))))))))))).((((((.......))).))))))..)))))). ( -63.30) >consensus AGGAGUUCUGCGAGGAGCUGCUGCUGGGCAUGCUGCUGCAGGCCGCUUCGUUGGCCAGCAGCGUAUGAAUGGCCGCCAGCAGCUUCUUGCGAGCUCCAAACGCCGUGAUACCCAGCUCCA .(((((((.((((((((((((((.(((.(((.(((((((.(((..........))).))))))...).))).))).))))))))))))))))))))).......((........)).... (-53.89 = -54.50 + 0.61)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:49:27 2006