
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 1,756,798 – 1,756,933 |
| Length | 135 |
| Max. P | 0.975714 |

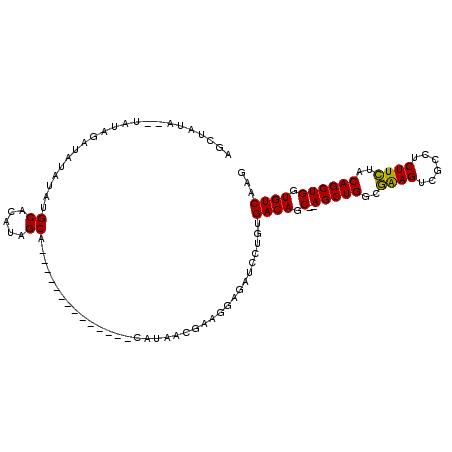
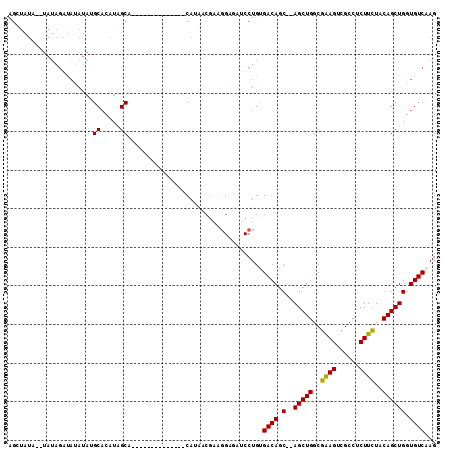
| Location | 1,756,798 – 1,756,893 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 78.25 |
| Mean single sequence MFE | -31.83 |
| Consensus MFE | -17.05 |
| Energy contribution | -16.55 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.975714 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1756798 95 + 22407834 AGCUAUA--UAUAGAUAUAUAUGCACAUAGCA--------------CAUAACGAAGGAUCUCCUGUGACAGCACAGCUGGCGAAGUCGCCUCUUCUCCAGCUGGUGUCAAG .((((((--((((....)))))....))))).--------------........(((....))).(((((.(.(((((((.((((......)))).))))))))))))).. ( -31.70) >DroSec_CAF1 13914 93 + 1 AGCUAUA--UAUAGAUAUAUAAGCACAUAGCA--------------CAUAACGAAGGAUCUCCUGCGACAGC--AGCUGGCGAAGUCGCCUCUUCUCCAGCUGGUGUCAAG .((((((--((....))))).)))........--------------......(.(((....))).)((((.(--((((((.((((......)))).))))))).))))... ( -33.50) >DroSim_CAF1 20576 93 + 1 AGCUAUA--UAUAGAUAUAUAUGCACAUAGCA--------------CAUAACGAAGGAUCUCCUGCGACAGC--AGCUGGCGAAGUCGCCUCUUCUCCAGCUGGUGUCAAG .((((((--((((....)))))....))))).--------------......(.(((....))).)((((.(--((((((.((((......)))).))))))).))))... ( -34.60) >DroEre_CAF1 13575 95 + 1 AGAUAUAUAUAUAGAUAUAUAUGCACAUGGCC--------------CAUGACGAUGGGGAUCCUGUGACAGC--AGCUGGCGAAGUCGCCUCUUCUACAGCUGGUGUCAAG ..((((((((....))))))))..(((.((((--------------(((....)))))...)))))((((.(--(((((..((((......))))..)))))).))))... ( -34.30) >DroYak_CAF1 20987 107 + 1 AGAUAUA--UAUAAAUUCAUAUGCACAUAGCACAUAGCACAGAGCACAUAACGAUGGGGAUCCUGUGACAGC--AGCUGGCGGAGUCGCCUCUUCUACAGCUGGUGUCAAG .......--......(((((.(((.....)))....((.....))........))))).......(((((.(--(((((..((((......))))..)))))).))))).. ( -27.40) >DroAna_CAF1 13043 78 + 1 AG----------AGGCAAAGUCGCCCCCCGCC--------------CAU-------GGGAUCCUGUGACAGC--AGCUGGCUGAGUCGCCUCUUAUACAGCUGGUGUCAAG ..----------.(((......))).((((..--------------..)-------)))......(((((.(--(((((..((((......))))..)))))).))))).. ( -29.50) >consensus AGCUAUA__UAUAGAUAUAUAUGCACAUAGCA______________CAUAACGAAGGAGAUCCUGUGACAGC__AGCUGGCGAAGUCGCCUCUUCUACAGCUGGUGUCAAG ......................((.....))...................................((((.(..(((((..((((......))))..)))))).))))... (-17.05 = -16.55 + -0.50)

| Location | 1,756,798 – 1,756,893 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 78.25 |
| Mean single sequence MFE | -31.72 |
| Consensus MFE | -15.22 |
| Energy contribution | -15.05 |
| Covariance contribution | -0.17 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.48 |
| SVM decision value | 1.09 |
| SVM RNA-class probability | 0.913285 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1756798 95 - 22407834 CUUGACACCAGCUGGAGAAGAGGCGACUUCGCCAGCUGUGCUGUCACAGGAGAUCCUUCGUUAUG--------------UGCUAUGUGCAUAUAUAUCUAUA--UAUAGCU ..(((((((((((((.((((......)))).))))))).).))))).(((....)))....((((--------------(((.....)))))))........--....... ( -32.00) >DroSec_CAF1 13914 93 - 1 CUUGACACCAGCUGGAGAAGAGGCGACUUCGCCAGCU--GCUGUCGCAGGAGAUCCUUCGUUAUG--------------UGCUAUGUGCUUAUAUAUCUAUA--UAUAGCU ...((((.(((((((.((((......)))).))))))--).))))(.(((....))).)......--------------.((((((((............))--)))))). ( -32.30) >DroSim_CAF1 20576 93 - 1 CUUGACACCAGCUGGAGAAGAGGCGACUUCGCCAGCU--GCUGUCGCAGGAGAUCCUUCGUUAUG--------------UGCUAUGUGCAUAUAUAUCUAUA--UAUAGCU ...((((.(((((((.((((......)))).))))))--).))))(((((....)))....((((--------------(((.....)))))))........--....)). ( -34.60) >DroEre_CAF1 13575 95 - 1 CUUGACACCAGCUGUAGAAGAGGCGACUUCGCCAGCU--GCUGUCACAGGAUCCCCAUCGUCAUG--------------GGCCAUGUGCAUAUAUAUCUAUAUAUAUAUCU ...((((.((((((..((((......))))..)))))--).))))(((((...(((((....)))--------------)))).)))..((((((((....)))))))).. ( -30.10) >DroYak_CAF1 20987 107 - 1 CUUGACACCAGCUGUAGAAGAGGCGACUCCGCCAGCU--GCUGUCACAGGAUCCCCAUCGUUAUGUGCUCUGUGCUAUGUGCUAUGUGCAUAUGAAUUUAUA--UAUAUCU ..(((((.((((((..(..(((....))))..)))))--).)))))..((....))....((((((((.(...((.....))...).)))))))).......--....... ( -31.90) >DroAna_CAF1 13043 78 - 1 CUUGACACCAGCUGUAUAAGAGGCGACUCAGCCAGCU--GCUGUCACAGGAUCCC-------AUG--------------GGCGGGGGGCGACUUUGCCU----------CU (((((((.((((((.....(((....)))...)))))--).))))).))......-------..(--------------(((((((.....))))))))----------.. ( -29.40) >consensus CUUGACACCAGCUGGAGAAGAGGCGACUUCGCCAGCU__GCUGUCACAGGAGACCCUUCGUUAUG______________UGCUAUGUGCAUAUAUAUCUAUA__UAUAGCU (((((((..(((((..((((......))))..)))))....))))).))...............................((.....))...................... (-15.22 = -15.05 + -0.17)

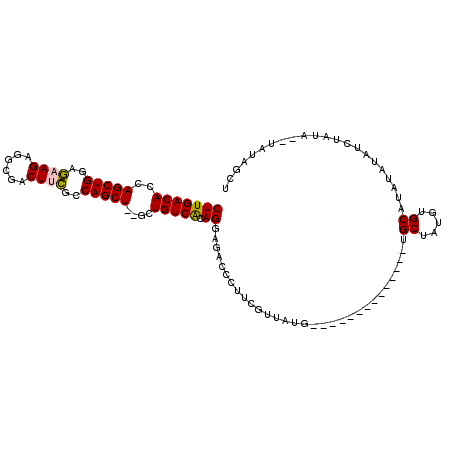
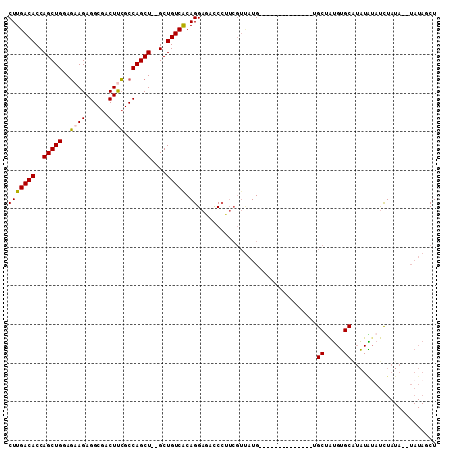
| Location | 1,756,827 – 1,756,933 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.74 |
| Mean single sequence MFE | -36.92 |
| Consensus MFE | -19.53 |
| Energy contribution | -21.20 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.53 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.943504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1756827 106 + 22407834 A--------------CAUAACGAAGGAUCUCCUGUGACAGCACAGCUGGCGAAGUCGCCUCUUCUCCAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAAGGGGCUGACU .--------------.(((((((.((((.((((.(((((.(.(((((((.((((......)))).))))))))))))))))).....))))...)))))))((((.......)))).... ( -33.80) >DroPse_CAF1 15454 91 + 1 -------------------------GCUUGGCUGUGACAGC--CGCUGCCAAAGCCUCCUCGGCUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAGGGGA--GACU -------------------------(((((((.(((.....--))).))).))))(((((((((((((((.((((.((((........)))))))))))))))))))....)))--)... ( -36.90) >DroEre_CAF1 13606 104 + 1 C--------------CAUGACGAUGGGGAUCCUGUGACAGC--AGCUGGCGAAGUCGCCUCUUCUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAAGGGGCUGACU (--------------((..((((((.(((((((.(((((.(--(((((..((((......))))..)))))).)))))))))......))).))))))..)..((....))))....... ( -38.70) >DroYak_CAF1 21016 118 + 1 ACAUAGCACAGAGCACAUAACGAUGGGGAUCCUGUGACAGC--AGCUGGCGGAGUCGCCUCUUCUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAAGGGGCUGACU ...((((.........(((((((((.(((((((.(((((.(--(((((..((((......))))..)))))).)))))))))......))).)))))))))..((....))..))))... ( -41.50) >DroAna_CAF1 13064 97 + 1 C--------------CAU-------GGGAUCCUGUGACAGC--AGCUGGCUGAGUCGCCUCUUAUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAGGGGGACGACU .--------------...-------....((((.(((((.(--(((((..((((......))))..)))))).)))))))))..............(((((..((....))...))))). ( -32.50) >DroPer_CAF1 14487 91 + 1 -------------------------GCUUGGCUGUGACAGC--CGCUGCCAAAGCCUCCUCGGCUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAGGGGG--GACU -------------------------..(((((.(((.....--))).)))))..((((((((((((((((.((((.((((........)))))))))))))))))))..)))))--.... ( -38.10) >consensus _______________CAU_______GGGAUCCUGUGACAGC__AGCUGGCGAAGUCGCCUCUUCUACAGCUGGUGUCAAGGAAUAAUAUUUGCAUUGUUGUAGCUGAAAAGGGGCUGACU .....................................((((...))))....((((((((((((((((((.((((.((((........))))))))))))))).....)))))).))))) (-19.53 = -21.20 + 1.67)

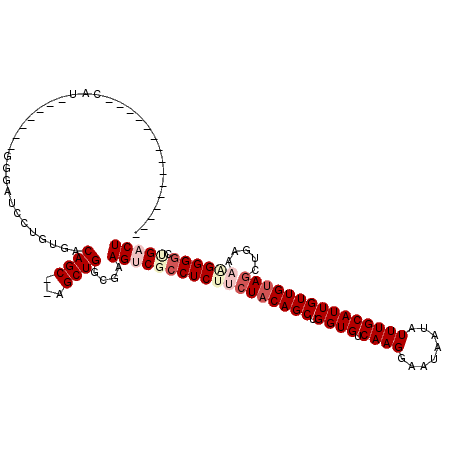
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:39:49 2006