
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
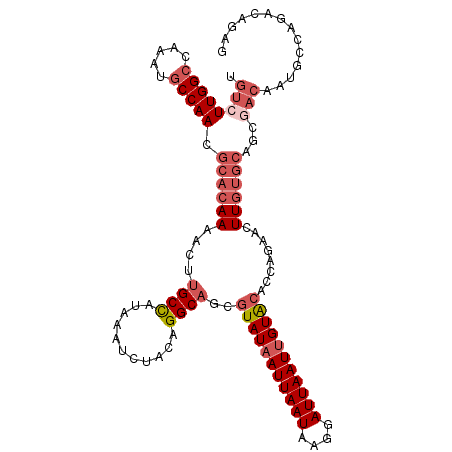
| Location | 15,868,223 – 15,868,334 |
| Length | 111 |
| Max. P | 0.778825 |

| Location | 15,868,223 – 15,868,334 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 87.75 |
| Mean single sequence MFE | -29.71 |
| Consensus MFE | -21.56 |
| Energy contribution | -23.28 |
| Covariance contribution | 1.72 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.21 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.17 |
| SVM RNA-class probability | 0.617584 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
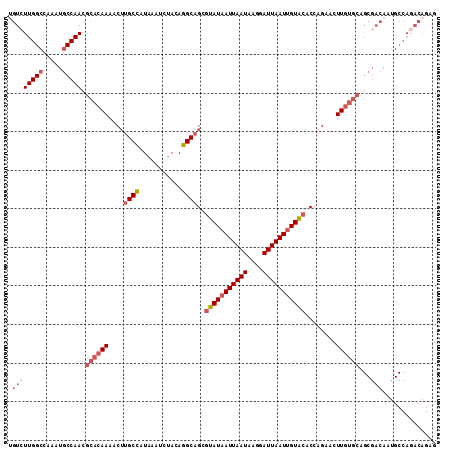
>2L_DroMel_CAF1 15868223 111 + 22407834 CGUCUUGGCCAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAUGCCAGACAGAG .((((.(((....(((....((((((...(((((...........))))).(((((((((((....)))))))))))........)))))).))).....))))))).... ( -31.60) >DroGri_CAF1 13040 89 + 1 UUUCUUGGGCGCAUGCCAACACACAAAACAAGCUAUUAAUCCACAGGCAGCAUAUAAUUAAUAAGGAUUAAUAGUGCACCAGAACUUAC---------------------- .((((.((((....)))..............(((((((((((......................)))))))))))...).)))).....---------------------- ( -17.15) >DroSec_CAF1 41847 111 + 1 UGUCUUGGCCAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAUGCCAGACAGAG (((((.(((....(((....((((((...(((((...........))))).(((((((((((....)))))))))))........)))))).))).....))))))))... ( -32.00) >DroSim_CAF1 41804 111 + 1 UGUCUUGGCCAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAUGCCAGACAGAG (((((.(((....(((....((((((...(((((...........))))).(((((((((((....)))))))))))........)))))).))).....))))))))... ( -32.00) >DroEre_CAF1 31770 111 + 1 UGUGUUGGCUAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAGGCCAGACAGAG (((((((((.....))))))))).......((((...........))))(((((((((((((....))))))))))).......(((((......)))))))......... ( -33.50) >DroYak_CAF1 16320 111 + 1 UGUCUUGGCCAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAUGCCAGACAGAG (((((.(((....(((....((((((...(((((...........))))).(((((((((((....)))))))))))........)))))).))).....))))))))... ( -32.00) >consensus UGUCUUGGCCAAAUGCCAACGCACAAAACUUGCCAUAAAUCUACAGGCAGCGUAUAAUUAAUAAGGAUUAAUUGUACACCAGAACUUGUGCAGCGACAAUGCCAGACAGAG .((((((((.....))))).((((((....((((...........))))..(((((((((((....)))))))))))........))))))...))).............. (-21.56 = -23.28 + 1.72)

| Location | 15,868,223 – 15,868,334 |
|---|---|
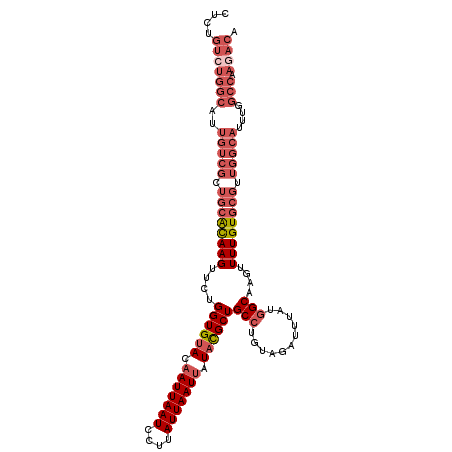
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 87.75 |
| Mean single sequence MFE | -35.09 |
| Consensus MFE | -26.03 |
| Energy contribution | -28.78 |
| Covariance contribution | 2.75 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.41 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.778825 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
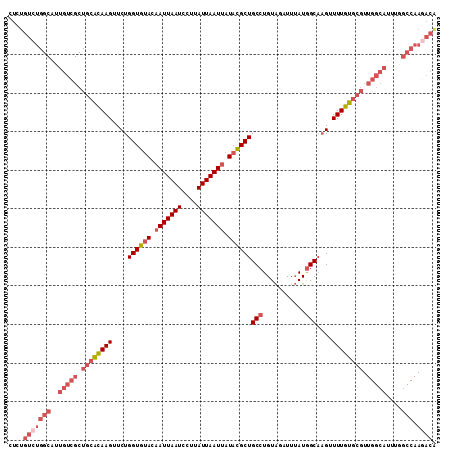
>2L_DroMel_CAF1 15868223 111 - 22407834 CUCUGUCUGGCAUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUGGCCAAGACG ....(((((((..(((((.((((((((..((((((((.(((((((....))))))).))))))(((...........))).)).)))))))).)))))....))).)))). ( -38.60) >DroGri_CAF1 13040 89 - 1 ----------------------GUAAGUUCUGGUGCACUAUUAAUCCUUAUUAAUUAUAUGCUGCCUGUGGAUUAAUAGCUUGUUUUGUGUGUUGGCAUGCGCCCAAGAAA ----------------------.....((((((.((.((((((((((......................))))))))))........(((((....))))))))).)))). ( -21.75) >DroSec_CAF1 41847 111 - 1 CUCUGUCUGGCAUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUGGCCAAGACA ....(((((((..(((((.((((((((..((((((((.(((((((....))))))).))))))(((...........))).)).)))))))).)))))....))).)))). ( -38.60) >DroSim_CAF1 41804 111 - 1 CUCUGUCUGGCAUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUGGCCAAGACA ....(((((((..(((((.((((((((..((((((((.(((((((....))))))).))))))(((...........))).)).)))))))).)))))....))).)))). ( -38.60) >DroEre_CAF1 31770 111 - 1 CUCUGUCUGGCCUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUAGCCAACACA ...(((.((((..(((((.((((((((..((((((((.(((((((....))))))).))))))(((...........))).)).)))))))).)))))....))))))).. ( -34.40) >DroYak_CAF1 16320 111 - 1 CUCUGUCUGGCAUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUGGCCAAGACA ....(((((((..(((((.((((((((..((((((((.(((((((....))))))).))))))(((...........))).)).)))))))).)))))....))).)))). ( -38.60) >consensus CUCUGUCUGGCAUUGUCGCUGCACAAGUUCUGGUGUACAAUUAAUCCUUAUUAAUUAUACGCUGCCUGUAGAUUUAUGGCAAGUUUUGUGCGUUGGCAUUUGGCCAAGACA ....(((((((..(((((.((((((((....((((((.(((((((....))))))).))))))(((...........)))....)))))))).)))))....))).)))). (-26.03 = -28.78 + 2.75)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:48:05 2006