
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,752,956 – 15,753,086 |
| Length | 130 |
| Max. P | 0.988367 |

| Location | 15,752,956 – 15,753,064 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 74.13 |
| Mean single sequence MFE | -39.64 |
| Consensus MFE | -26.30 |
| Energy contribution | -26.80 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.66 |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.987886 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15752956 108 + 22407834 U----GAUUGGCUCU-------GUAAUUGGACGAUUCGGCUGACUCCCUAAGCGCAUCCUGCGCUUAGAGAGCUGUUACUUAUACGGAUGUCUGAGGCACUCCCUCCACCAGGUGGGUU .----...((.(((.-------......((((.(((((((.(.(((.((((((((.....)))))))).)))).))........)))))))))))).))..(((.((....)).))).. ( -34.41) >DroVir_CAF1 4453 99 + 1 UCAUUGAAAG-------------------GGCUAUGUGGAUU-CUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUACUUAGACAGAUGUCUGGGGCACACCCUCCACCAGCUGCGUU ..........-------------------((((..(((((..-((((((((((((.....)))))))))))).(((..(((((((....)))))))....))).))))).))))..... ( -42.30) >DroPse_CAF1 4748 112 + 1 C----GAAAGGCUUAUUGG---UUGGUUGGAUGGUUCGAUUUACUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUUCUUAAACGGAGGUCUGGGGCACGCCUUCUACCAGCUGUGUG (----....).......((---(((((.((...((((......((((((((((((.....))))))))))))((((((((.....))))).)))))))...))....)))))))..... ( -41.80) >DroWil_CAF1 8848 98 + 1 -----GAAAGGAUU----------------AUGGUUUAUUUCACUCUCUAAACGCAUCCCGCGCUUAGAGAGCGGUUACUUAGACUGAUGUCUGAGGUAUGCCCUCCACUAGCUCUGUA -----...((..((----------------((((.........((((((((.(((.....))).)))))))).(((..(((((((....)))))))....)))..))).)))..))... ( -31.50) >DroAna_CAF1 4647 113 + 1 C----GAAAGGCUGU--GGUACGUUGUUGGACGAUUCGGCUGACUCUCUAAGCGCAUCCCGCACUUAGAGAGCUGUUACUUAGACGGACGUCUGGGGCACUCCCUCCACCAGCUGGGUG .----....((((((--((.........(((((.((((.(((((((((((((.((.....)).))))))))).......)))).)))))))))(((.....))).))).)))))..... ( -40.61) >DroPer_CAF1 4779 112 + 1 C----GAAAGGCUUAUUGG---UUGGUUGGAUGGUUCGAUUUACUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUUCUUAGACGGAGGUCUGGGGCACGCCUUCUACCAGCUGUGUG (----....).......((---(((((.(((.(((........((((((((((((.....))))))))))))..((.((((((((....)))))))).))))).))))))))))..... ( -47.20) >consensus C____GAAAGGCUU_________U_GUUGGACGAUUCGGUUUACUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUACUUAGACGGAUGUCUGGGGCACGCCCUCCACCAGCUGUGUG .........((................................((((((((((((.....))))))))))))..((..(((((((....)))))))..))........))......... (-26.30 = -26.80 + 0.50)



| Location | 15,752,956 – 15,753,064 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 74.13 |
| Mean single sequence MFE | -35.27 |
| Consensus MFE | -26.27 |
| Energy contribution | -26.75 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.75 |
| SVM decision value | 2.12 |
| SVM RNA-class probability | 0.988367 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15752956 108 - 22407834 AACCCACCUGGUGGAGGGAGUGCCUCAGACAUCCGUAUAAGUAACAGCUCUCUAAGCGCAGGAUGCGCUUAGGGAGUCAGCCGAAUCGUCCAAUUAC-------AGAGCCAAUC----A ........((((.((((.....)))).(((....((.......)).(((((((((((((.....)))))))))))))..........))).......-------...))))...----. ( -33.60) >DroVir_CAF1 4453 99 - 1 AACGCAGCUGGUGGAGGGUGUGCCCCAGACAUCUGUCUAAGUAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAG-AAUCCACAUAGCC-------------------CUUUCAAUGA ......(((((((((.(((.(((...((((....))))..)))))).((((((((((((.....))))))))))))-..))))).)))).-------------------.......... ( -40.90) >DroPse_CAF1 4748 112 - 1 CACACAGCUGGUAGAAGGCGUGCCCCAGACCUCCGUUUAAGAAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAGUAAAUCGAACCAUCCAACCAA---CCAAUAAGCCUUUC----G .............((((((((..(..((((....))))..)..)).(((((((((((((.....)))))))))))))....................---.......)))))).----. ( -31.30) >DroWil_CAF1 8848 98 - 1 UACAGAGCUAGUGGAGGGCAUACCUCAGACAUCAGUCUAAGUAACCGCUCUCUAAGCGCGGGAUGCGUUUAGAGAGUGAAAUAAACCAU----------------AAUCCUUUC----- (((..........((((.....))))((((....))))..)))..((((((((((((((.....))))))))))))))...........----------------.........----- ( -33.50) >DroAna_CAF1 4647 113 - 1 CACCCAGCUGGUGGAGGGAGUGCCCCAGACGUCCGUCUAAGUAACAGCUCUCUAAGUGCGGGAUGCGCUUAGAGAGUCAGCCGAAUCGUCCAACAACGUACC--ACAGCCUUUC----G ......((((.(((.(((.....))).((((....((...((....(((((((((((((.....)))))))))))))..)).))..))))..........))--))))).....----. ( -38.40) >DroPer_CAF1 4779 112 - 1 CACACAGCUGGUAGAAGGCGUGCCCCAGACCUCCGUCUAAGAAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAGUAAAUCGAACCAUCCAACCAA---CCAAUAAGCCUUUC----G .............((((((((..(..((((....))))..)..)).(((((((((((((.....)))))))))))))....................---.......)))))).----. ( -33.90) >consensus CACACAGCUGGUGGAGGGCGUGCCCCAGACAUCCGUCUAAGUAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAGUAAACCGAACCAUCCAAC_A_________AAGCCUUUC____G ............((((((........((((....))))........(((((((((((((.....)))))))))))))...............................))))))..... (-26.27 = -26.75 + 0.48)

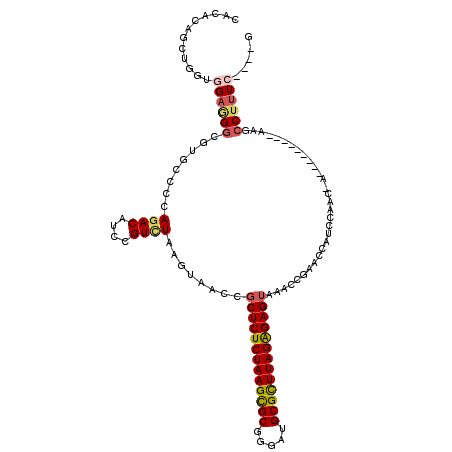
| Location | 15,752,984 – 15,753,086 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 83.91 |
| Mean single sequence MFE | -41.49 |
| Consensus MFE | -33.46 |
| Energy contribution | -34.63 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.754777 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15752984 102 + 22407834 CUGACUCCCUAAGCGCAUCCUGCGCUUAGAGAGCUGUUACUUAUACGGAUGUCUGAGGCACUCCCUCCACCAGGUGGGUUCUCCUCGGCUGAGCCGCUGGCU ....(((.((((((((.....)))))))).)))((((.......))))..(.((((((....(((.((....)).)))....)))))))..((((...)))) ( -38.40) >DroVir_CAF1 4473 101 + 1 AUU-CUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUACUUAGACAGAUGUCUGGGGCACACCCUCCACCAGCUGCGUUCGACGCGGCUGCACCGCUGGCU ...-.(((((((((((.....)))))))))))(((((..(((((((....))))))).............((((((((.....)))))))).)))))..... ( -49.00) >DroPse_CAF1 4780 102 + 1 UUUACUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUUCUUAAACGGAGGUCUGGGGCACGCCUUCUACCAGCUGUGUGCGUCGGGGUUGAGCCGCUGGCU .....(((((((((((.....)))))))))))((((((((((.....))).(((((.((((((............)))))).)))))...)))))))..... ( -42.40) >DroGri_CAF1 4512 102 + 1 AUCACUCCCUAAGCGCAUCGCGCACUUAGAGAGCGGUUACUUAGACAGACGUCUGUGGCACCCCCUCCACCAGCUGCGUGCGUCGUGGCUGGGCAGCCGGCU ....(((.(((((.((.....)).))))).)))(((((.(((((.(((((((.((..((.............))..)).))))).)).))))).)))))... ( -37.82) >DroMoj_CAF1 4500 101 + 1 AUU-CUCUCUAAGCGCAUCGCGCGCCUAGAGAGCGGUUACUUAGACGGAUGUUUGGGGCACGCCCUCCACCAGCUGAGUGCGUCGUGGCUGUACCGCCGGCU ...-(((((((.((((.....)))).)))))))..((.((((((..((.((...((((....)))).))))..))))))))(((((((.....))).)))). ( -39.20) >DroAna_CAF1 4680 102 + 1 CUGACUCUCUAAGCGCAUCCCGCACUUAGAGAGCUGUUACUUAGACGGACGUCUGGGGCACUCCCUCCACCAGCUGGGUGCGUCUCGGCUGCGGCGCCGGCU ....(((((((((.((.....)).)))))))))............(((.(((((((((......))))).((((((((.....)))))))).)))))))... ( -42.10) >consensus AUUACUCUCUAAGCGCAUCCCGCGCUUAGAGAGCGGUUACUUAGACGGAUGUCUGGGGCACGCCCUCCACCAGCUGCGUGCGUCGCGGCUGAGCCGCCGGCU ....((((((((((((.....))))))))))))((((..(((((((....))))))).............((((((((.....))))))))....))))... (-33.46 = -34.63 + 1.17)



| Location | 15,752,984 – 15,753,086 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 83.91 |
| Mean single sequence MFE | -43.07 |
| Consensus MFE | -31.11 |
| Energy contribution | -31.28 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.669670 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15752984 102 - 22407834 AGCCAGCGGCUCAGCCGAGGAGAACCCACCUGGUGGAGGGAGUGCCUCAGACAUCCGUAUAAGUAACAGCUCUCUAAGCGCAGGAUGCGCUUAGGGAGUCAG .....((((.....(.((((....(((.((....)).)))....)))).)....))))..........(((((((((((((.....)))))))))))))... ( -41.30) >DroVir_CAF1 4473 101 - 1 AGCCAGCGGUGCAGCCGCGUCGAACGCAGCUGGUGGAGGGUGUGCCCCAGACAUCUGUCUAAGUAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAG-AAU .((((((((.....))(((.....))).))))))((.(((....))).((((....))))......)).((((((((((((.....))))))))))))-... ( -48.20) >DroPse_CAF1 4780 102 - 1 AGCCAGCGGCUCAACCCCGACGCACACAGCUGGUAGAAGGCGUGCCCCAGACCUCCGUUUAAGAAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAGUAAA .((((((.((((......)).)).....)))))).(.((((........).))).)............(((((((((((((.....)))))))))))))... ( -37.80) >DroGri_CAF1 4512 102 - 1 AGCCGGCUGCCCAGCCACGACGCACGCAGCUGGUGGAGGGGGUGCCACAGACGUCUGUCUAAGUAACCGCUCUCUAAGUGCGCGAUGCGCUUAGGGAGUGAU ....(((.((((..((((.(.((.....))).))))...)))))))((((....)))).........((((((((((((((.....)))))))))))))).. ( -46.90) >DroMoj_CAF1 4500 101 - 1 AGCCGGCGGUACAGCCACGACGCACUCAGCUGGUGGAGGGCGUGCCCCAAACAUCCGUCUAAGUAACCGCUCUCUAGGCGCGCGAUGCGCUUAGAGAG-AAU ....((.(((((.(((.(..(((.(......))))..))))))))))).....................((((((((((((.....))))))))))))-... ( -41.40) >DroAna_CAF1 4680 102 - 1 AGCCGGCGCCGCAGCCGAGACGCACCCAGCUGGUGGAGGGAGUGCCCCAGACGUCCGUCUAAGUAACAGCUCUCUAAGUGCGGGAUGCGCUUAGAGAGUCAG ...((((......))))(((((.((....((((.((........))))))..)).)))))........(((((((((((((.....)))))))))))))... ( -42.80) >consensus AGCCAGCGGCGCAGCCGCGACGCACCCAGCUGGUGGAGGGAGUGCCCCAGACAUCCGUCUAAGUAACCGCUCUCUAAGCGCGGGAUGCGCUUAGAGAGUAAU .((((((.....................))))))...(((....))).((((....))))........(((((((((((((.....)))))))))))))... (-31.11 = -31.28 + 0.17)

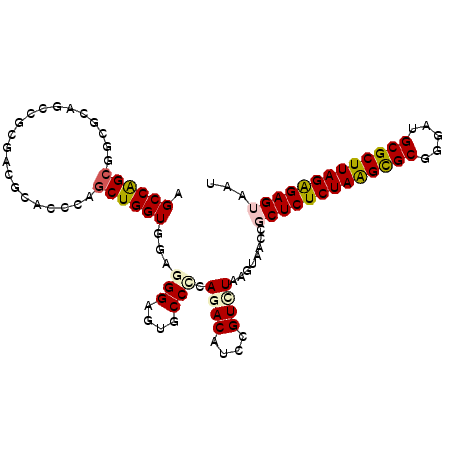

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:45:50 2006