
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,713,110 – 15,713,250 |
| Length | 140 |
| Max. P | 0.940644 |

| Location | 15,713,110 – 15,713,218 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.87 |
| Mean single sequence MFE | -33.23 |
| Consensus MFE | -25.23 |
| Energy contribution | -24.70 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.24 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.677144 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15713110 108 + 22407834 AUGGGGGUUUAUGGGGAGUU--GGGGAAUUGGAAGGCCAGCCAGAGGUUACGGGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU-----AUAGUAC-- ........((((((....(.--.(((........((((((((...(....)..)))..)))))...)))..)....((((((((((.....))))))))))))-----))))...-- ( -31.40) >DroVir_CAF1 72141 101 + 1 AUGGGG----AUGGGGGGUA------UAUUGUAGUGUUUG-CCGAGGUUACGAGGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCUGU---GUAGUAC-- ......----..((.(..((------(......)))..).-))((((((.((((((.........)))))).))))))(((((((..(((.....)))..)))))---)).....-- ( -34.00) >DroSec_CAF1 41494 108 + 1 AUGGGGAUUUAUGUGGAGUU--GGGGAAUUGGAAGGCCAGACAGAGGUUACGGGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU-----AUAGUAC-- ...........(((((....--...(((((..(((((..(((...(((........))).)))..)))))..)))))(((((((((.....))))))))).))-----)))....-- ( -30.30) >DroEre_CAF1 40555 113 + 1 AUGGGGAUUUAUGGGGAGUU--UGGGAAUUGGAAGGCCAACCAGAGGUUACGGGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCUAUGCUAUAGUAC-- ........((((((.(((((--..((((((..(((((..(((((.(.(.....).).)))))...)))))..))))))((((((((.....)))))))))))).).))))))...-- ( -32.30) >DroMoj_CAF1 43411 107 + 1 GUGGGU----AGGGGGAGUGUUGGGCUGUAGUAGUGUUUG-UCGAGGUUACGAGGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCUGU---GUAGUAC-- ......----.....((((.(((((((..(.(((((((..-(((......)))))))))).)..)))).))).))))((((((((..(((.....)))..)))))---)))....-- ( -34.30) >DroAna_CAF1 32870 108 + 1 -UGAGGACUCCAG-GGAACA--GGAGAAGGGGAGGCCAGGCCAGAGGUUACGAGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU-----AUAGUACAC -......((((.(-....).--))))..((.....)).((((.((((((.((((((.........)))))).))))))((((((((.....))))))))))))-----......... ( -37.10) >consensus AUGGGGAUUUAUGGGGAGUU__GGGGAAUUGGAAGGCCAGCCAGAGGUUACGAGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU_____AUAGUAC__ ...................................(((.....((((((.((((((.........)))))).))))))((((((((.....)))))))))))............... (-25.23 = -24.70 + -0.53)

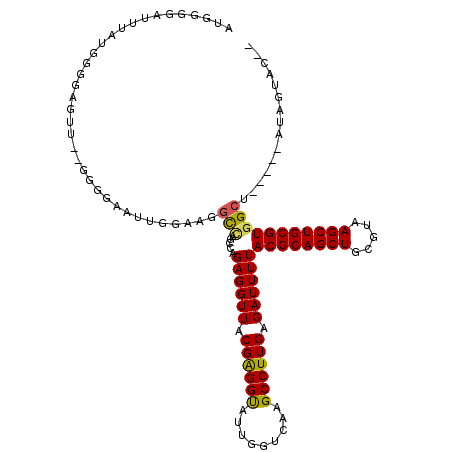
| Location | 15,713,110 – 15,713,218 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.87 |
| Mean single sequence MFE | -22.72 |
| Consensus MFE | -15.33 |
| Energy contribution | -15.33 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940644 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15713110 108 - 22407834 --GUACUAU-----AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCCCGUAACCUCUGGCUGGCCUUCCAAUUCCCC--AACUCCCCAUAAACCCCCAU --.......-----....((((((((.....))))))))........((((((.(.(((...............)))).))))))..........--.................... ( -21.86) >DroVir_CAF1 72141 101 - 1 --GUACUAC---ACAGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUGCCUCGUAACCUCGG-CAAACACUACAAUA------UACCCCCCAU----CCCCAU --.......---...(((((((((((.....)))))))).........((((.........)))).........))-).............------..........----...... ( -23.00) >DroSec_CAF1 41494 108 - 1 --GUACUAU-----AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCCCGUAACCUCUGUCUGGCCUUCCAAUUCCCC--AACUCCACAUAAAUCCCCAU --.......-----....((((((((.....))))))))........((((((.(((..................))).))))))..........--.................... ( -21.57) >DroEre_CAF1 40555 113 - 1 --GUACUAUAGCAUAGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCCCGUAACCUCUGGUUGGCCUUCCAAUUCCCA--AACUCCCCAUAAAUCCCCAU --................((((((((.....))))))))........((((((.(((((...............)))))))))))..........--.................... ( -22.76) >DroMoj_CAF1 43411 107 - 1 --GUACUAC---ACAGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUGCCUCGUAACCUCGA-CAAACACUACUACAGCCCAACACUCCCCCU----ACCCAC --....(((---......((((((((.....)))))))).........((((.........)))).))).......-..............................----...... ( -18.30) >DroAna_CAF1 32870 108 - 1 GUGUACUAU-----AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCUCGUAACCUCUGGCCUGGCCUCCCCUUCUCC--UGUUCC-CUGGAGUCCUCA- .........-----.(((((((((((.....)))))))).........(((((.((....(((...)))...)).))))))))........((((--......-..))))......- ( -28.80) >consensus __GUACUAU_____AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCCCGUAACCUCUGGCUGGCCCUCCAAUUCCCC__AACUCCCCAUAAAUCCCCAU ..............((((((((((((.....))))))))..........))))................................................................ (-15.33 = -15.33 + 0.00)

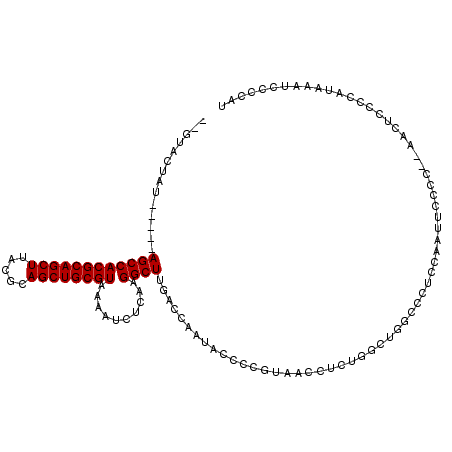

| Location | 15,713,145 – 15,713,250 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 82.00 |
| Mean single sequence MFE | -38.47 |
| Consensus MFE | -24.90 |
| Energy contribution | -23.93 |
| Covariance contribution | -0.97 |
| Combinations/Pair | 1.31 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.905931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15713145 105 + 22407834 CAGCCAGAGGUUACGGGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU--AUAGUAC------ACGAUAA-ACCGUUAUUAUGUGCCAUGUG---CCCC .((((...))))..(((((((((((..((((..........((((((((.....))))))))))))--((((((.------(((....-..)))))))))..)))).)))---)))) ( -37.50) >DroVir_CAF1 72168 109 + 1 UUG-CCGAGGUUACGAGGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCUGUGUAGUAC------ACGAUAAUACCGUUAUUAUGUACUACACGA-AUACC ..(-((((((((.((((((.........)))))).))))))((((((((.....)))))))))))((((((((((------(..(((((...)))))..))))))))))).-..... ( -48.30) >DroPse_CAF1 47243 103 + 1 --GUCAAAGGUUACGAGGUAUUGGUCAAGCCUUGAGAUUUAACGCAGCUGCGUAAGCUGCGUGGCU--GUAGUAC------ACGUUAA-GCCGUUAUUAUGUGCCAUGUA---UAUG --.....(((((.((((((.........)))))).))))).((((((((.....))))))))((((--((((((.------(((....-..)))))))))).))).....---.... ( -31.60) >DroGri_CAF1 44383 109 + 1 CCG-CCUAGGCUAAG-GGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCAUGGCUGUGUAGUAC------ACGAUAAUACCGUUAUUAUGUAUCGCACUAUACACC ..(-((.(((((...-(((....))).)))))...........((((((.....))))))..))).(((..((((------(..(((((...)))))..)))))..)))........ ( -36.70) >DroMoj_CAF1 43444 109 + 1 UUG-UCGAGGUUACGAGGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCUGUGUAGUAC------ACGAUAAUGCCGUUAUUAUAUACUAUACGA-AUACA ..(-((((((((.((((((.........)))))).))))))((((((((.....)))))))))))(((((((((.------(..(((((...)))))..).))))))))).-..... ( -37.70) >DroAna_CAF1 32903 110 + 1 AGGCCAGAGGUUACGAGGUAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU--AUAGUACACGUUCACGAUAA-ACCGUUAUUAUGUGCUAUAUA---CAC- .((((.((((((.((((((.........)))))).))))))((((((((.....))))))))))))--((((((((.....(((....-..))).....))))))))...---...- ( -39.00) >consensus CUG_CAGAGGUUACGAGGCAUUGGUCAAGCCUUGAGAUUUUACGCAGCUGCGUAAGCUGCGUGGCU__GUAGUAC______ACGAUAA_ACCGUUAUUAUGUACCAUACG___CACC ........(((((((((((.........)))))..........((((((.....))))))))))))..((.((((......(((.......)))......)))).)).......... (-24.90 = -23.93 + -0.97)



| Location | 15,713,145 – 15,713,250 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 82.00 |
| Mean single sequence MFE | -30.59 |
| Consensus MFE | -17.45 |
| Energy contribution | -17.53 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.563695 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15713145 105 - 22407834 GGGG---CACAUGGCACAUAAUAACGGU-UUAUCGU------GUACUAU--AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCCCGUAACCUCUGGCUG ((((---...((((.(((..((((....-))))..)------)).))))--((((((((((((.....))))))))..........)))).........)))).............. ( -29.20) >DroVir_CAF1 72168 109 - 1 GGUAU-UCGUGUAGUACAUAAUAACGGUAUUAUCGU------GUACUACACAGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUGCCUCGUAACCUCGG-CAA .....-..((((((((((..((((.....))))..)------))))))))).(((((((((((.....)))))))).........((((.........)))).........))-).. ( -40.60) >DroPse_CAF1 47243 103 - 1 CAUA---UACAUGGCACAUAAUAACGGC-UUAACGU------GUACUAC--AGCCACGCAGCUUACGCAGCUGCGUUAAAUCUCAAGGCUUGACCAAUACCUCGUAACCUUUGAC-- ....---.....(((........(((..-....)))------.......--.)))((((((((.....))))))))......((((((..(((........)))....)))))).-- ( -23.09) >DroGri_CAF1 44383 109 - 1 GGUGUAUAGUGCGAUACAUAAUAACGGUAUUAUCGU------GUACUACACAGCCAUGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUGCC-CUUAGCCUAGG-CGG .(((((..(..(((((.(((.......)))))))).------.)..))))).(((((((((((.....)))))))).........(((((...........-...))))).))-).. ( -33.14) >DroMoj_CAF1 43444 109 - 1 UGUAU-UCGUAUAGUAUAUAAUAACGGCAUUAUCGU------GUACUACACAGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUGCCUCGUAACCUCGA-CAA .((((-(.((.(((((((..((((.....))))..)------))))))((.((((((((((((.....))))))))..........)))))))).))))).(((......)))-... ( -28.30) >DroAna_CAF1 32903 110 - 1 -GUG---UAUAUAGCACAUAAUAACGGU-UUAUCGUGAACGUGUACUAU--AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCUCGUAACCUCUGGCCU -(((---..(((((.((((....((((.-...))))....)))).))))--)..)))((((((.....))))))...........(((((.((....(((...)))...)).))))) ( -29.20) >consensus GGUA___CAUAUAGCACAUAAUAACGGU_UUAUCGU______GUACUAC__AGCCACGCAGCUUACGCAGCUGCGUAAAAUCUCAAGGCUUGACCAAUACCUCGUAACCUCGG_CAA ..........((((.((......((((.....))))......)).))))..((((((((((((.....))))))))..........))))........................... (-17.45 = -17.53 + 0.08)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:45:34 2006