| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,647,169 – 15,647,289 |
| Length | 120 |
| Max. P | 0.874274 |

| Location | 15,647,169 – 15,647,289 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.89 |
| Mean single sequence MFE | -44.97 |
| Consensus MFE | -31.50 |
| Energy contribution | -32.00 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.48 |
| Structure conservation index | 0.70 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.508452 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
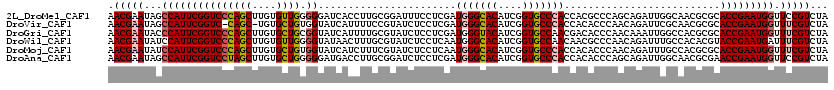
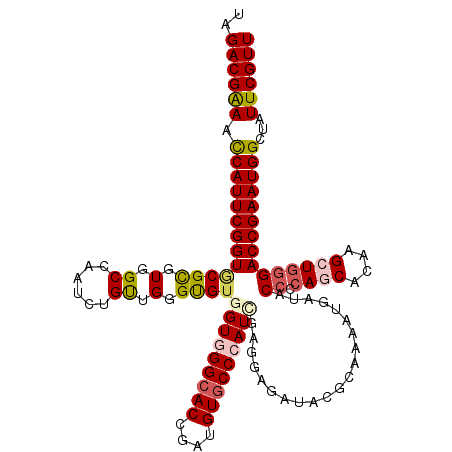
>2L_DroMel_CAF1 15647169 120 + 22407834 AACGAAUAGCCAUUCGGUCCCAGCUUGUGUUGGGGGAUCACCUUGCGGAUUUCCUCGAUGGGCACAUCGGUGCCCACCACGCCCAGCAGAUUGGCAACGCGCACCGAAUGGUUCCGUCUA ...((..(((((((((((....((.(.((((((((((...((....))...)))..(.(((((((....))))))).)...))))))).)..(....)))..)))))))))))...)).. ( -49.40) >DroVir_CAF1 20599 118 + 1 AACGAAUAGCCAUUCGGUC-CAGC-UGUGCUGUGGUAUCAUUUUCCGUAUCUCCUCGAUGGGCACAUCGGUGCCCACCACACCCAACAGAUUCGCAACGCGCACCGAAUGGUUUCGUCUA .(((((..((((((((((.-...(-(((..((((((.........((........))..((((((....))))))))))))....))))....((.....)))))))))))))))))... ( -44.50) >DroGri_CAF1 5894 120 + 1 AACGAAUACCCAUUCGGUCCCAGCUUGUGCUGCGGUAUCAUUUUGCGUAUCUCCUCGAUGGGUACAUCGGUGCCAACGACACCCAACAAAUUGGCCACGCGCACCGAAUGGUUUCGUCUA .(((((...(((((((((((((((....)))).)).........((((.......(((((....)))))(((((((..............)))).)))))))))))))))).)))))... ( -42.84) >DroWil_CAF1 21281 120 + 1 AACGAAUAUCCAUUCGGUCCCAGCUUGUGUUGGGGUAUAACUUUGCGUAUCUCCUCAAUGGGCACAUCGGUGCCAACAACGCCCAACAGAUUUGCCACACGUACCGAAUGAUUUCGUCUA .(((((....(((((((((...((.(.((((((((((......)))..............(((((....))))).......))))))).)...)).....).))))))))..)))))... ( -36.50) >DroMoj_CAF1 3929 120 + 1 AACGAAUAUCCAUUCGGUCCCAGCUUGUGCUGUGGUAUCAUCUUUCGUAUCUCCUCAAUGGGCACAUCGGUGCCCACCACACCCAACAGAUUUGCCACGCGCACCGAAUGGUUUCGUCUA .(((((...(((((((((........((((.((((((..((((...(........)..(((((((....)))))))...........)))).))))))))))))))))))).)))))... ( -43.40) >DroAna_CAF1 7958 120 + 1 AACGAAUAGCCAUUCGGUCCUAGCUUGUGCUGGGGGAUGACCUUGCGGAUCUCCUCGAUGGGCACAUCGGUGCCCACCACACCCAGCAGAUUGGCAACGCGAACCGAAUGGUUCCGUCUA ...((..(((((((((((....((.(.((((((((((.((((....)).)))))..(.(((((((....))))))).)...))))))).)..(....)))..)))))))))))...)).. ( -53.20) >consensus AACGAAUAGCCAUUCGGUCCCAGCUUGUGCUGGGGUAUCACCUUGCGUAUCUCCUCGAUGGGCACAUCGGUGCCCACCACACCCAACAGAUUGGCAACGCGCACCGAAUGGUUUCGUCUA .(((((...(((((((((((((((....)))).)).......................(((((((....)))))))..........................))))))))).)))))... (-31.50 = -32.00 + 0.50)
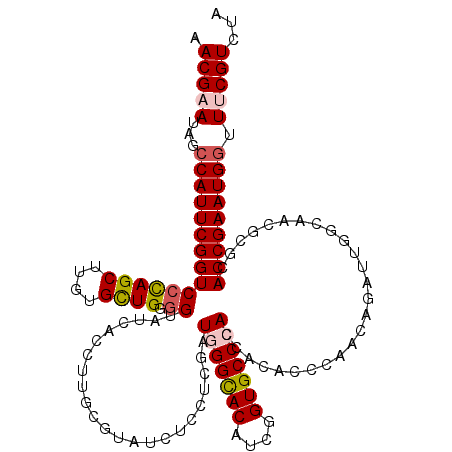
| Location | 15,647,169 – 15,647,289 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.89 |
| Mean single sequence MFE | -51.25 |
| Consensus MFE | -38.08 |
| Energy contribution | -38.62 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.80 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874274 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15647169 120 - 22407834 UAGACGGAACCAUUCGGUGCGCGUUGCCAAUCUGCUGGGCGUGGUGGGCACCGAUGUGCCCAUCGAGGAAAUCCGCAAGGUGAUCCCCCAACACAAGCUGGGACCGAAUGGCUAUUCGUU ..((((((.(((((((((((((.(.((......)).).))))(((((((((....)))))))))..(((...((....))...)))((((........)))))))))))))...)))))) ( -54.20) >DroVir_CAF1 20599 118 - 1 UAGACGAAACCAUUCGGUGCGCGUUGCGAAUCUGUUGGGUGUGGUGGGCACCGAUGUGCCCAUCGAGGAGAUACGGAAAAUGAUACCACAGCACA-GCUG-GACCGAAUGGCUAUUCGUU ..((((((.(((((((((.((.((((......(((((((((((((((((((....)))))))))..(......)........))))).)))))))-))))-.)))))))))...)))))) ( -50.10) >DroGri_CAF1 5894 120 - 1 UAGACGAAACCAUUCGGUGCGCGUGGCCAAUUUGUUGGGUGUCGUUGGCACCGAUGUACCCAUCGAGGAGAUACGCAAAAUGAUACCGCAGCACAAGCUGGGACCGAAUGGGUAUUCGUU ..((((((.(((((((((..(((((.....((((.((((((.(((((....))))))))))).))))....))))).........((.((((....)))))))))))))))...)))))) ( -52.90) >DroWil_CAF1 21281 120 - 1 UAGACGAAAUCAUUCGGUACGUGUGGCAAAUCUGUUGGGCGUUGUUGGCACCGAUGUGCCCAUUGAGGAGAUACGCAAAGUUAUACCCCAACACAAGCUGGGACCGAAUGGAUAUUCGUU ..((((((.(((((((((..(((((.....((...(((((...((((....))))..)))))..)).....)))))..........((((........)))))))))))))...)))))) ( -40.50) >DroMoj_CAF1 3929 120 - 1 UAGACGAAACCAUUCGGUGCGCGUGGCAAAUCUGUUGGGUGUGGUGGGCACCGAUGUGCCCAUUGAGGAGAUACGAAAGAUGAUACCACAGCACAAGCUGGGACCGAAUGGAUAUUCGUU ..((((((.(((((((((((((.(((((....))))).))))(((((((((....))))))))).........(....)......((.((((....)))))))))))))))...)))))) ( -49.00) >DroAna_CAF1 7958 120 - 1 UAGACGGAACCAUUCGGUUCGCGUUGCCAAUCUGCUGGGUGUGGUGGGCACCGAUGUGCCCAUCGAGGAGAUCCGCAAGGUCAUCCCCCAGCACAAGCUAGGACCGAAUGGCUAUUCGUU ..((((((.(((((((((((.....((.....(((((((..((((((((((....)))))))))).(((((.((....)))).))))))))))...))..)))))))))))...)))))) ( -60.80) >consensus UAGACGAAACCAUUCGGUGCGCGUGGCCAAUCUGUUGGGUGUGGUGGGCACCGAUGUGCCCAUCGAGGAGAUACGCAAAAUGAUACCCCAGCACAAGCUGGGACCGAAUGGCUAUUCGUU ..((((((.(((((((((((((.(.((......)).).))))(((((((((....))))))))).....................((.((((....)))))))))))))))...)))))) (-38.08 = -38.62 + 0.53)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:45:13 2006