
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,627,701 – 15,627,818 |
| Length | 117 |
| Max. P | 0.805715 |

| Location | 15,627,701 – 15,627,818 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.85 |
| Mean single sequence MFE | -42.85 |
| Consensus MFE | -27.65 |
| Energy contribution | -27.38 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.805715 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
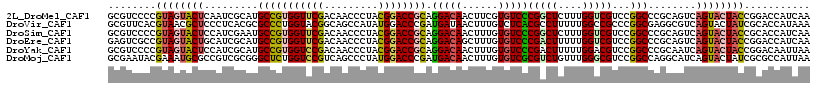
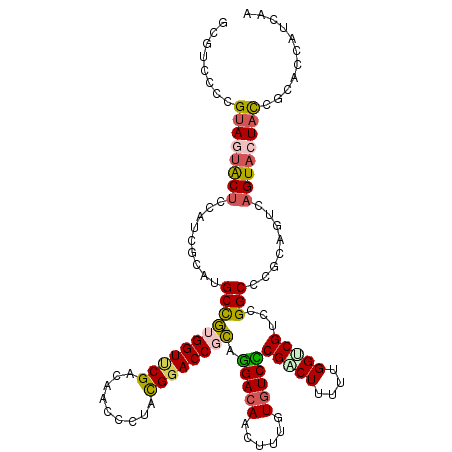
>2L_DroMel_CAF1 15627701 117 + 22407834 GCGUCCCCGUAGUACUCAAUCGCAUGCCGUGGUUCGACAACCCUACGGACCGCAGGACAACUUCGUGUCCCGGCUCUUUGGUCGUCCGGCCCGCAGUCAGUACUACCGGACCAUCAA (.((((..((((((((..((.((..(((((((((((.........)))))))).(((((......))))).))).....((((....)))).)).)).)))))))).)))))..... ( -44.40) >DroVir_CAF1 180879 117 + 1 GCGUUCACGUAACGCUCCCUCACGCGCCCUGGUACGGCAGCCAUAUGGACCCGAUGAUAACUUUGUCUCACGCCUUUUUGGCCGCCCGGCGAGGCGUCAGUACUAUCGCACCAUAAA (((((.....)))))........(((...((((((((...((....))..))((.((((....))))))(((((((....(((....))))))))))..)))))).)))........ ( -34.00) >DroSim_CAF1 127837 117 + 1 GCGUCCCCGUAGUACUCCAUCGAAUGCCGUGGUUCGACAACCCUACGGACCGCAGGACAACUUUGUGUCCCGGCUCUUUGGUCGUCCGGCCCGCAGUCAGUACUACCGCACCAUCAA (((.....((((((((....((...(((((((((((.........)))))))).(((((......))))).))).....((((....)))))).....)))))))))))........ ( -39.50) >DroEre_CAF1 130034 117 + 1 GAGUCGCCGUAGUACUGCAUCGCAUGCCGUGGUUCGACAACCCUACGGACCGCAGGACAGCUUUGUGUCCCGACUUUUUGGUCGUCCGGCCCGCAGUCAGUACUACCGGACCAUCAA ((....(((((((((((.((.((..(((((((((((.........)))))))).(((((......)))))(((((....)))))...)))..)).)))))))))).)))....)).. ( -47.30) >DroYak_CAF1 141975 117 + 1 GCGUCCCCGUAGUACUCCAUCGCAUGCCGUGGUCCGACAACCCUACGGACCGCAGGACAACUUUGUGUCCCGACUUUUUGGACGUCCGGCCCGCAAUCAGUACUACCGGACAAUUAA ..((((..((((((((.....((..(((((((((((.........)))))))).((((.((...))((((.........)))))))))))..))....)))))))).))))...... ( -45.40) >DroMoj_CAF1 179510 117 + 1 GCGAAUACGAAAUGCGCCGUCGCGGGCUCUGGUCCGUCAGCCCUAUGGACCCGAUGACAACUUUGUGUCGCGUCUGUUUGGGCGUCCGGCCAGGCAUCAGUACUAUCGCGCCAUUAA ((((.(((...(((((((......))).((((.(((...((((.((((((.((((.(((....))))))).))))))..))))...)))))))))))..)))...))))........ ( -46.50) >consensus GCGUCCCCGUAGUACUCCAUCGCAUGCCGUGGUUCGACAACCCUACGGACCGCAGGACAACUUUGUGUCCCGACUUUUUGGUCGUCCGGCCCGCAGUCAGUACUACCGCACCAUCAA ........((((((((.........(((((((((((.........)))))))).(((((......)))))(((((....)))))...)))........))))))))........... (-27.65 = -27.38 + -0.27)
| Location | 15,627,701 – 15,627,818 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 80.85 |
| Mean single sequence MFE | -48.15 |
| Consensus MFE | -27.86 |
| Energy contribution | -30.42 |
| Covariance contribution | 2.56 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.602111 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
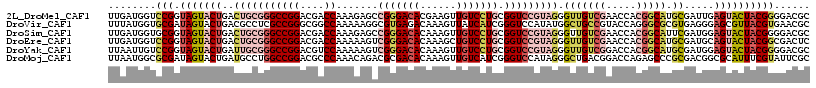
>2L_DroMel_CAF1 15627701 117 - 22407834 UUGAUGGUCCGGUAGUACUGACUGCGGGCCGGACGACCAAAGAGCCGGGACACGAAGUUGUCCUGCGGUCCGUAGGGUUGUCGAACCACGGCAUGCGAUUGAGUACUACGGGGACGC .....(..((.((((((((.((((((((((((....)).....((.((((((......))))))))))))))))).(((((((.....))))).))...).)))))))).))..).. ( -50.70) >DroVir_CAF1 180879 117 - 1 UUUAUGGUGCGAUAGUACUGACGCCUCGCCGGGCGGCCAAAAAGGCGUGAGACAAAGUUAUCAUCGGGUCCAUAUGGCUGCCGUACCAGGGCGCGUGAGGGAGCGUUACGUGAACGC ((((((((((....))))(((((((((((..((((((((....(((.(((((........)).))).)))....))))))))(..(....)..))))))...))))))))))))... ( -41.70) >DroSim_CAF1 127837 117 - 1 UUGAUGGUGCGGUAGUACUGACUGCGGGCCGGACGACCAAAGAGCCGGGACACAAAGUUGUCCUGCGGUCCGUAGGGUUGUCGAACCACGGCAUUCGAUGGAGUACUACGGGGACGC ........((.((((((((..(((((((((((....)).....((.((((((......)))))))))))))))))(..(((((.....)))))..).....))))))))(....))) ( -47.70) >DroEre_CAF1 130034 117 - 1 UUGAUGGUCCGGUAGUACUGACUGCGGGCCGGACGACCAAAAAGUCGGGACACAAAGCUGUCCUGCGGUCCGUAGGGUUGUCGAACCACGGCAUGCGAUGCAGUACUACGGCGACUC .....(((((.(((((((((.((((((((((..((((......))))(((((......)))))..)))))))))).(((((((.....))))).))....))))))))).).)))). ( -54.70) >DroYak_CAF1 141975 117 - 1 UUAAUUGUCCGGUAGUACUGAUUGCGGGCCGGACGUCCAAAAAGUCGGGACACAAAGUUGUCCUGCGGUCCGUAGGGUUGUCGGACCACGGCAUGCGAUGGAGUACUACGGGGACGC ......((((.((((((((.((((((.((((............(.(((((((......))))))))((((((.........)))))).)))).)))))).).)))))))..)))).. ( -51.70) >DroMoj_CAF1 179510 117 - 1 UUAAUGGCGCGAUAGUACUGAUGCCUGGCCGGACGCCCAAACAGACGCGACACAAAGUUGUCAUCGGGUCCAUAGGGCUGACGGACCAGAGCCCGCGACGGCGCAUUUCGUAUUCGC ........((((..((((.((((((((((((...((((.....(((.(((.(((....)))..))).)))....))))...))).)))).(((......))))))))..)))))))) ( -42.40) >consensus UUGAUGGUCCGGUAGUACUGACUGCGGGCCGGACGACCAAAAAGCCGGGACACAAAGUUGUCCUGCGGUCCGUAGGGUUGUCGAACCACGGCAUGCGAUGGAGUACUACGGGGACGC ........(((.(((((((..(((((((((((....)).......(((((((......))))))).))))))))).(((((((.....))))).)).....))))))))))...... (-27.86 = -30.42 + 2.56)
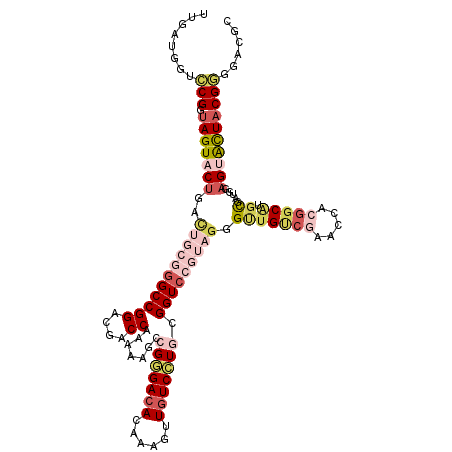
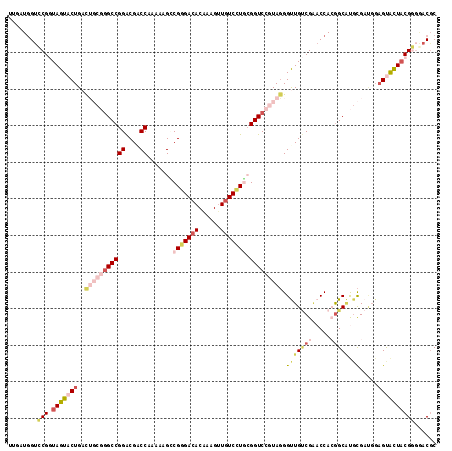
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:45:02 2006