
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,170,285 – 15,170,405 |
| Length | 120 |
| Max. P | 0.956282 |

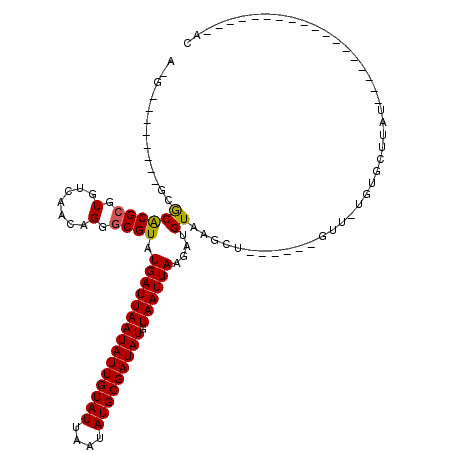
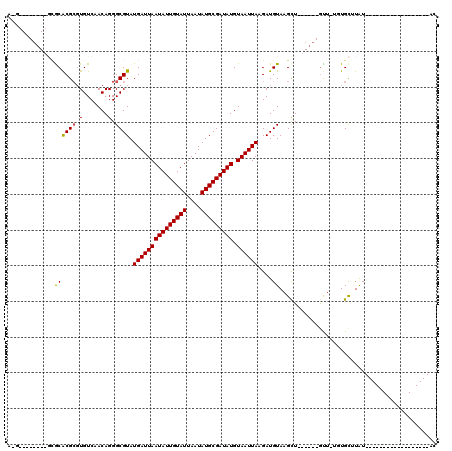
| Location | 15,170,285 – 15,170,377 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.80 |
| Mean single sequence MFE | -25.52 |
| Consensus MFE | -12.98 |
| Energy contribution | -12.70 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.46 |
| SVM RNA-class probability | 0.956282 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15170285 92 - 22407834 AAGG---GA-GGCGCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCU------ACUCUGUGCUUAU------------------AU ....---((-.(((....))).))...........(((((((((((((((....))))))))).))))))..((((((((.------.......))))))------------------)) ( -23.00) >DroPse_CAF1 142311 102 - 1 AGGGCGAGAGCGCACACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCUGGCUCUGUUUUGUGCUUAU------------------AC .((((((..((((....)))).))((((((((...(((((((((((((((....))))))))).))))))((.......)).))))))))....))))..------------------.. ( -28.50) >DroGri_CAF1 135976 81 - 1 G----------UUGCGCG--UGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCU------GUU---UGCGUAU------------------AU .----------.((((((--....(((((.((((.(((((((((((((((....))))))))).))))))..))))...))------)))---)))))).------------------.. ( -22.30) >DroWil_CAF1 187409 104 - 1 AAA----------GCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCU------GUUCUCUGUGUGUGUGUAUAUAUAUAUAUAUAC ...----------(((((((.(..(((((.((((.(((((((((((((((....))))))))).))))))..))))...))------)))..))))))))((((((((....)))))))) ( -30.00) >DroMoj_CAF1 167424 83 - 1 A----------GUGCGCGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGUU------GUU---UGCAUAU------------------AU .----------((((((.(.((....)).)))))))((((((((((((((....))))))))).)))))...(((((((..------.))---)))))..------------------.. ( -20.80) >DroPer_CAF1 145075 102 - 1 AGGGCGAGAGCGCACACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCUGGCUCUGUUUUGUGCUUAU------------------AC .((((((..((((....)))).))((((((((...(((((((((((((((....))))))))).))))))((.......)).))))))))....))))..------------------.. ( -28.50) >consensus A_G________GCGCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGCU______GUU_UGUGCUUAU__________________AC .............((((((.(.......).)))).(((((((((((((((....))))))))).))))))....))............................................ (-12.98 = -12.70 + -0.28)

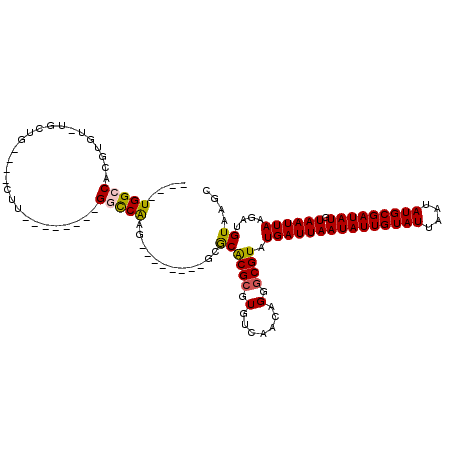
| Location | 15,170,301 – 15,170,405 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.90 |
| Mean single sequence MFE | -30.17 |
| Consensus MFE | -15.06 |
| Energy contribution | -14.73 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.684401 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
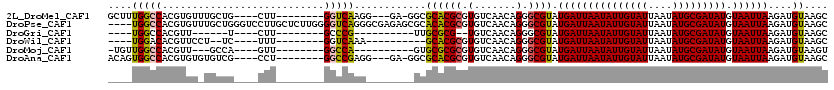
>2L_DroMel_CAF1 15170301 104 - 22407834 GCUUUGGCCACGUGUUUGCUG----CUU--------GGUCAAGG---GA-GGCGCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ((((((..(((((((.((((.----(((--------......))---).-)))).)))))))....))))))...(((((((((((((((....))))))))).)))))).......... ( -32.70) >DroPse_CAF1 142333 116 - 1 ----UGGCCACGUGUUUGCUGGGUCCUUGCUCUUGGGGUCAGGGCGAGAGCGCACACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ----((((.((((((.(((...(..((((((((.......))))))))..)))).)))))))))).....((((.(((((((((((((((....))))))))).))))))..)))).... ( -37.60) >DroGri_CAF1 135989 86 - 1 ----UGGCCACGUU------U----CUU--------GCCCG----------UUGCGCG--UGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ----.....(((((------(----...--------(((((----------((((...--.).)))).))))...(((((((((((((((....))))))))).)))))))))))).... ( -22.60) >DroWil_CAF1 187443 92 - 1 ----UGGACACGUUCCU--UC----UUU--------GGUCAAA----------GCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ----..(((((((....--.(----(((--------....)))----------)...)))))))......((((.(((((((((((((((....))))))))).))))))..)))).... ( -22.50) >DroMoj_CAF1 167437 94 - 1 -UGUUGGCCACGUU---GCCA----GUU--------GGCCA----------GUGCGCGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGU -...(((((((...---....----).)--------)))))----------((((((.(.((....)).)))))))((((((((((((((....))))))))).)))))........... ( -27.60) >DroAna_CAF1 122257 104 - 1 ACAGUGGCCACGUGUGUGUCG----CCU--------GGCCGAGG---GA-GGCGCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ....((((.(((((((((((.----(((--------......))---).-))))))))))))))).....((((.(((((((((((((((....))))))))).))))))..)))).... ( -38.00) >consensus ____UGGCCACGUGU_UGCUG____CUU________GGCCAAG________GCGCACGCGUGUCAACAGGGCGUAUGAUUAAUAUUGUAUUAAUAUGCGAUAUGUAAUUAAGAUGUAAGC ....(((((...........................)))))............((((((.(.......).)))).(((((((((((((((....))))))))).))))))....)).... (-15.06 = -14.73 + -0.33)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:42:09 2006