
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 15,145,027 – 15,145,165 |
| Length | 138 |
| Max. P | 0.999844 |

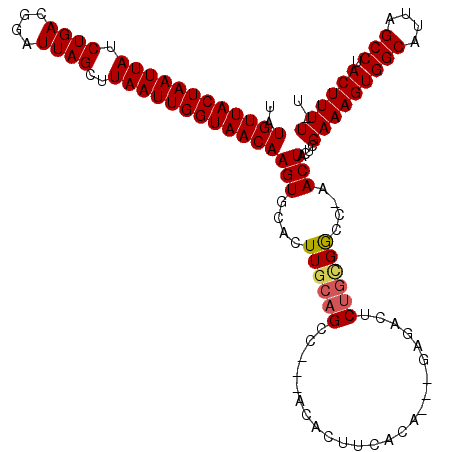
| Location | 15,145,027 – 15,145,147 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.04 |
| Mean single sequence MFE | -28.78 |
| Consensus MFE | -24.24 |
| Energy contribution | -24.49 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.655952 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15145027 120 - 22407834 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCUGCGGCCGAACUACUCGAAAGUGGCAUUAGCCUACUUUUUU ..((((((((((((.((((....))))..))))))))))))(((....((((((........((.....)).......))))))....)))....(((((((((....))).)))))).. ( -26.66) >DroVir_CAF1 121118 112 - 1 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCC---ACACUUCACU---GCGACUCUGCGGCC-AACUACUCGAAAGUGGCAUUAGCCUACUUUU-U ..((((((((((((.((((....))))..))))))))))))..((((.((((((..---........))---))))...))))...-........(((((((((....))).))))))-. ( -29.80) >DroPse_CAF1 109528 120 - 1 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACGAGCGAGACUCUGCGGCCGAACUACUCGAAAGUGGCAUUAGCCUACUUUUUU ..((((((((((((.((((....))))..))))))))))))..(((....)))(((......(.....).(((......))))))..........(((((((((....))).)))))).. ( -28.10) >DroGri_CAF1 105953 112 - 1 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCC---ACACUUCACA---GCGACUCUGCGGCC-AACUACUCGAAAGUGGCAUUAGCCUACUUUU-U ..((((((((((((.((((....))))..))))))))))))..(((....)))(((---.((..((...---..))...)).))).-........(((((((((....))).))))))-. ( -28.60) >DroWil_CAF1 154930 117 - 1 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCC-ACACACUUCACAAGCGAGACUCUGUGGCC-AACUACUCGAAAGUGGCAUUAGCCUACUUUU-U ..((((((((((((.((((....))))..))))))))))))..(((....)))(((-(((...(((......)))....)))))).-........(((((((((....))).))))))-. ( -32.90) >DroMoj_CAF1 128240 112 - 1 UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCC---ACACUUCACU---GCGACUCCUAGACC-AACUACUCGAAAGUGGCAUUAGCCCACUUUU-U ..((((((((((((.((((....))))..)))))))))))).......((((((..---........))---))))..........-........(((((((((....).))))))))-. ( -26.60) >consensus UAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCC___ACACUUCACA___GAGACUCUGCGGCC_AACUACUCGAAAGUGGCAUUAGCCUACUUUU_U ..((((((((((((.((((....))))..))))))))))))(((....((((((........................))))))....)))....(((((((((....))).)))))).. (-24.24 = -24.49 + 0.25)

| Location | 15,145,067 – 15,145,165 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 89.73 |
| Mean single sequence MFE | -25.40 |
| Consensus MFE | -21.28 |
| Energy contribution | -21.16 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.74 |
| SVM RNA-class probability | 0.974864 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15145067 98 + 22407834 AGAGUCUAGCUUGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACACACACA--CU--CG--------A .((((......((((..(((((((((..(((((....)))))..))......((((((..........)))))).....))))))).)))).)--))--).--------. ( -24.10) >DroPse_CAF1 109568 110 + 1 AGAGUCUCGCUCGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACACACUGGCACGCAGGAGGACACAC ...((((..((((((.((((((((((..(((((....)))))..))).....((((((..........))))))..........)))))))..)))).))..)))).... ( -32.60) >DroSec_CAF1 90194 96 + 1 AGAGUCUAGCUUGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACACACACA------CA--------A ........(..((((..(((((((((..(((((....)))))..))......((((((..........)))))).....))))))).))))..------).--------. ( -21.90) >DroSim_CAF1 89718 96 + 1 AGAGUCUAGCUUGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACACACACA------CA--------A ........(..((((..(((((((((..(((((....)))))..))......((((((..........)))))).....))))))).))))..------).--------. ( -21.90) >DroYak_CAF1 105709 96 + 1 AGAGUCUAGCUUGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACAAGCACA------CA--------C ........(((((((...((((((((..(((((....)))))..))......((((((..........))))))))))))...)))))))...------..--------. ( -26.50) >consensus AGAGUCUAGCUUGUGAAGUGUGUUGGCUGCAAGUGCACUUGUUACCAAUUAAGCUAAUCCGUCAGAUAAUUAGUAACAUAACACACACACACA______CA________A .(((.....)))(((..(((((((((..(((((....)))))..))......((((((..........)))))).....))))))).))).................... (-21.28 = -21.16 + -0.12)

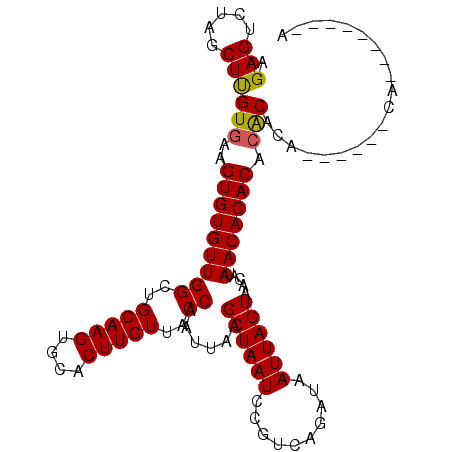
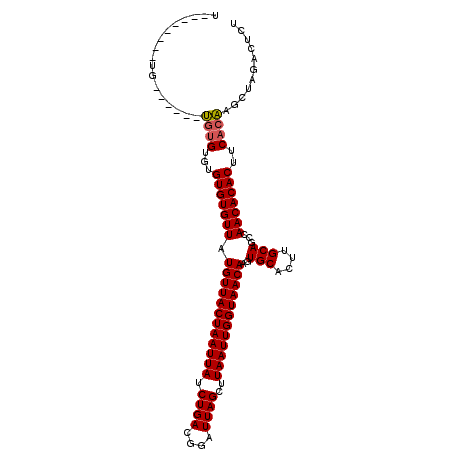
| Location | 15,145,067 – 15,145,165 |
|---|---|
| Length | 98 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 89.73 |
| Mean single sequence MFE | -31.74 |
| Consensus MFE | -27.70 |
| Energy contribution | -27.78 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.87 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 15145067 98 - 22407834 U--------CG--AG--UGUGUGUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCU (--------(.--((--(.((((.(((((((.((((((((((((.((((....))))..))))))))))))..(((....)))...)))))))..)))).))).)).... ( -32.60) >DroPse_CAF1 109568 110 - 1 GUGUGUCCUCCUGCGUGCCAGUGUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACGAGCGAGACUCU ..(.(((.((((.((((..(((((((..(((.((((((((((((.((((....))))..))))))))))))...((....)))))..))))))).)))))).))))).). ( -35.30) >DroSec_CAF1 90194 96 - 1 U--------UG------UGUGUGUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCU .--------.(------(.((((.(((((((.((((((((((((.((((....))))..))))))))))))..(((....)))...)))))))..)))).))........ ( -30.40) >DroSim_CAF1 89718 96 - 1 U--------UG------UGUGUGUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCU .--------.(------(.((((.(((((((.((((((((((((.((((....))))..))))))))))))..(((....)))...)))))))..)))).))........ ( -30.40) >DroYak_CAF1 105709 96 - 1 G--------UG------UGUGCUUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCU (--------(.------((((...(((((((.((((((((((((.((((....))))..))))))))))))..(((....)))...)))))))..)))).))........ ( -30.00) >consensus U________UG______UGUGUGUGUGUGUUAUGUUACUAAUUAUCUGACGGAUUAGCUUAAUUGGUAACAAGUGCACUUGCAGCCAACACACUUCACAAGCUAGACUCU .................((((...(((((((.((((((((((((.((((....))))..))))))))))))..(((....)))...)))))))..))))........... (-27.70 = -27.78 + 0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:42:00 2006