
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 14,603,848 – 14,604,008 |
| Length | 160 |
| Max. P | 0.871792 |

| Location | 14,603,848 – 14,603,968 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -51.80 |
| Consensus MFE | -31.60 |
| Energy contribution | -30.38 |
| Covariance contribution | -1.21 |
| Combinations/Pair | 1.47 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.871792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
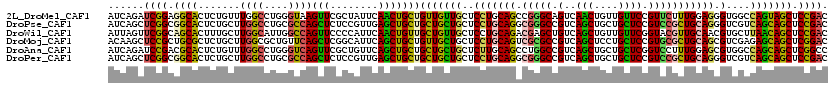
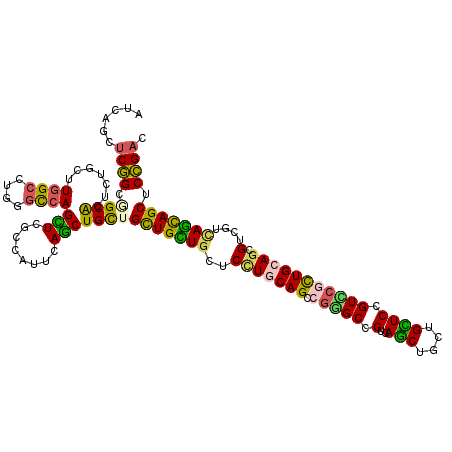
>2L_DroMel_CAF1 14603848 120 + 22407834 AUCAGAUCGGAGGCACUCUGUUUGGCCUGGGUAAGUUCGCUAUUCAACUGCUGUUGUUGCUCCUGCAGCCGGGCAGUCAACUGUUGUUCCGUUCUUUGGAGGGUGGCCAGUAGCUCCGAC ......((((((.(...(((...(..((.....))..)(((((((.((((((...(((((....)))))..))))))..........((((.....))))))))))))))..))))))). ( -37.70) >DroPse_CAF1 16366 120 + 1 AUCAGCUCGGCGGCACUCUGCUUGGCCUGCGCCAGCUCUCCGUUGAGCUGCUGCUGCUGCUCCUGCAGGCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGGGUCGUCAGCAGCUCCGAC .((((((((((((......(((.(((....))))))...))))))))))(((((((.((..(((((((.(((((.(..(((....)))).))))))))))))..)).)))))))...)). ( -63.10) >DroWil_CAF1 163716 120 + 1 AUUAGUUCGGCAGCACUUUGCUUGGCAUUGGCCAGUUCCCCAUUCAACUGUUGCUGUUGCUCCUGCAGACGAGCUGUCAGCUGUUGUUCGGUACGUUGCAACGUGCUUAACAGCUCCGAC ...((((.((((((.((((((..((((..((((((((........)))))..)))..))))...))))).).))))))))))((((((.(((((((....))))))).))))))...... ( -43.60) >DroMoj_CAF1 8755 120 + 1 ACAAGCUCCGCUGCGCUCUGCUUGGCGCUGUUCAGCUCGGCAUUCAGCUGCUGUUGCUGCUCCUGCAGUCGCGCCGUCAGCUCCUGCUCCGUGCGCUGCAGCGUCGAGAGCAGCUCGGAC ......((((((((......(((((((((((...((.(((.....(((((((((.(((((....))))).)))..).)))))......))).))...))))))))))).)))))..))). ( -52.20) >DroAna_CAF1 3083 120 + 1 AUCAGAUCCGACGCACUCUGUUUGGCCUGGGUCAGUUCGCUGUUCAGCUGCUGCUGCUGCUCUUGCAGCCUGGCCGUCAGCUGCUGCUCGGUCCUUUGGAGCGUGGCCAGCAGCUCGGCC ..(((..(.((((.....)))).)..)))((((((...(((((..(((.((....)).)))...)))))))))))(((((((((((((((.(((...))).)).)).)))))))).))). ( -51.10) >DroPer_CAF1 16343 120 + 1 AUCAGCUCGGCGGCACUCUGCUUGGCCUGCGCCAGCUCUCCGUUGAGCUGCUGCUGCUGCUCCUGCAGGCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGGGUCGUCAGCAGCUCCGAC .((((((((((((......(((.(((....))))))...))))))))))(((((((.((..(((((((.(((((.(..(((....)))).))))))))))))..)).)))))))...)). ( -63.10) >consensus AUCAGCUCGGCGGCACUCUGCUUGGCCUGGGCCAGCUCGCCAUUCAGCUGCUGCUGCUGCUCCUGCAGCCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGCGUCGUCAGCAGCUCCGAC ......((((.((((.......((((....))))(((........)))))))(((((((..(((((((.(((((.(..(((....)))).))))))))))).)....))))))).)))). (-31.60 = -30.38 + -1.21)
| Location | 14,603,888 – 14,604,008 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.28 |
| Mean single sequence MFE | -51.85 |
| Consensus MFE | -29.72 |
| Energy contribution | -28.53 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.613742 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 14603888 120 + 22407834 UAUUCAACUGCUGUUGUUGCUCCUGCAGCCGGGCAGUCAACUGUUGUUCCGUUCUUUGGAGGGUGGCCAGUAGCUCCGACAUGCGGGCCUCCCGCAGUGAAGCCUCUGCCCGCAAGGUCU ........(((....(((((....))))).((((((...(((((...((((.....))))(((.((((....((........)).)))).)))))))).......)))))))))...... ( -46.20) >DroPse_CAF1 16406 120 + 1 CGUUGAGCUGCUGCUGCUGCUCCUGCAGGCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGGGUCGUCAGCAGCUCCGACAUUCGUGCCUCCCGCAGGGUGGCCUCUGCGCGCAGCGUCU .((((((((((((..((.((.(((((((.(((((.(..(((....)))).)))))))))))))).)))))))))).))))...(((((....(((((((....))))))).))).))... ( -58.20) >DroWil_CAF1 163756 120 + 1 CAUUCAACUGUUGCUGUUGCUCCUGCAGACGAGCUGUCAGCUGUUGUUCGGUACGUUGCAACGUGCUUAACAGCUCCGACAUGCGAGCCUCGCGUAGAGUGGCCUCCGCUCGAAGCGUUU ...((..((((.((....))....))))..))((((((((((((((...(((((((....)))))))))))))))..)))).))......(((...((((((...))))))...)))... ( -42.60) >DroMoj_CAF1 8795 120 + 1 CAUUCAGCUGCUGUUGCUGCUCCUGCAGUCGCGCCGUCAGCUCCUGCUCCGUGCGCUGCAGCGUCGAGAGCAGCUCGGACAUGCGUGCCUCUCGCACCUUCGCCUCAGUACGCAGUGUUU .....(((((((.(((.((((...((((.(((((.(..(((....)))).))))))))))))).))).))))))).((((((((((((.....((......))....))))))).))))) ( -48.80) >DroAna_CAF1 3123 120 + 1 UGUUCAGCUGCUGCUGCUGCUCUUGCAGCCUGGCCGUCAGCUGCUGCUCGGUCCUUUGGAGCGUGGCCAGCAGCUCGGCCAUACGGGCUUCUCUCAGGGAGGCCUCCGCCCUAAGAGUCU ....((((....))))..(((((((..((.((((((..((((((((((((.(((...))).)).)).))))))))))))))...((((((((.....))))))))..))..))))))).. ( -57.10) >DroPer_CAF1 16383 120 + 1 CGUUGAGCUGCUGCUGCUGCUCCUGCAGGCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGGGUCGUCAGCAGCUCCGACAUUCGUGCCUCCCGCAGGGUGGCCUCUGCGCGCAGCGUCU .((((((((((((..((.((.(((((((.(((((.(..(((....)))).)))))))))))))).)))))))))).))))...(((((....(((((((....))))))).))).))... ( -58.20) >consensus CAUUCAGCUGCUGCUGCUGCUCCUGCAGCCGGGCCGUCAGCUGCUGCUCCGUCCGCUGCAGCGUCGUCAGCAGCUCCGACAUGCGUGCCUCCCGCAGGGUGGCCUCUGCCCGCAGCGUCU ............(((((((..(((((((.(((((.(..(((....)))).))))))))))).)....)))))))...(((...(((((.....((......))....)))))....))). (-29.72 = -28.53 + -1.19)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:37:23 2006