
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
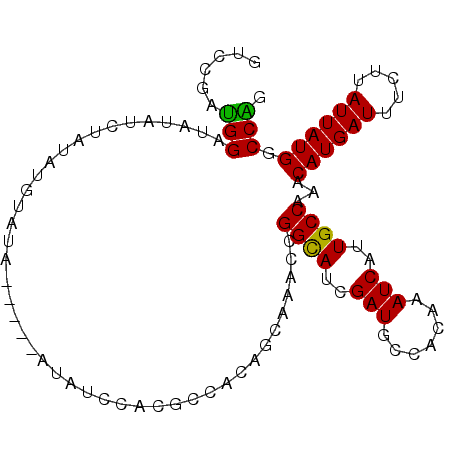
| Location | 14,546,260 – 14,546,354 |
| Length | 94 |
| Max. P | 0.620031 |

| Location | 14,546,260 – 14,546,354 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 83.98 |
| Mean single sequence MFE | -22.65 |
| Consensus MFE | -15.10 |
| Energy contribution | -14.60 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.540299 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 14546260 94 + 22407834 GUCCGAUGGAUAUAUCUAUAUGUAUAU--------CCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCAG ...((.(((((((((......))))))--------))))).....((.....((((..(((.......)))..))))...((((((.....))))))))... ( -23.70) >DroSec_CAF1 78127 102 + 1 GUCCGAUGGAUAUAUCUAUAUGUAUAUCCACAUAUCCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCAG ....(.(((((((((......))))))))))........((((.........((((....))))..(((((((........))))))).......))))... ( -23.00) >DroSim_CAF1 63927 102 + 1 GUCCGAUGGAUAUAUCUAUAUGUAUAUCCACAUAUCCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCAG ....(.(((((((((......))))))))))........((((.........((((....))))..(((((((........))))))).......))))... ( -23.00) >DroEre_CAF1 72790 99 + 1 A-CCAAUGGAUAUAUCUAUACGUACG--AGUAUAUCCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCAG .-....(((((((((...........--.))))))))).((((.........((((....))))..(((((((........))))))).......))))... ( -21.80) >DroYak_CAF1 69058 100 + 1 GUCCAAUGGAUAUAUCCAUAUGUACG--AGUAUAUCCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCGG ......((((((((..(........)--..)))))))).((....))...(((((..(((((........))))).....((((((.....))))))))))) ( -25.10) >DroAna_CAF1 16980 81 + 1 AUCCUACGGAUA--------------------UAUGCACUCCAUUGCG-GCCGGUAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCGG .......(((..--------------------.......)))....((-(((((((..(((.......)))..))))....(((((.....)))))))))). ( -19.30) >consensus GUCCGAUGGAUAUAUCUAUAUGUAUA_____AUAUCCACGCCACAGCAAACCGGCAUCGAUGCCACAAAUCAUUGCCAAACAUGAUUUCUUAUUAUGGCCAG ......(((...........................................((((..(((.......)))..))))...((((((.....)))))).))). (-15.10 = -14.60 + -0.50)


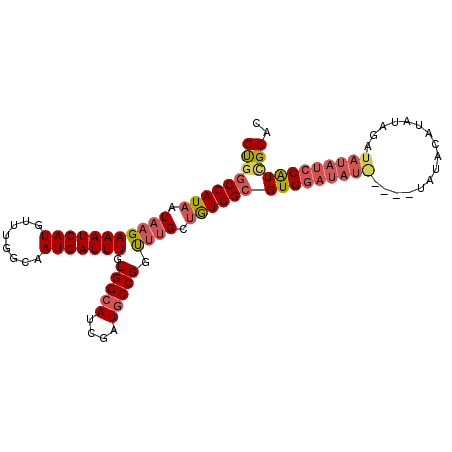
| Location | 14,546,260 – 14,546,354 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 83.98 |
| Mean single sequence MFE | -29.70 |
| Consensus MFE | -20.77 |
| Energy contribution | -21.94 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.620031 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 14546260 94 - 22407834 CUGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGG--------AUAUACAUAUAGAUAUAUCCAUCGGAC (((((((((.(((((((((((........))))))).(((((....))))).)))).))))))((((--------(((((........)))))))))))).. ( -29.70) >DroSec_CAF1 78127 102 - 1 CUGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGGAUAUGUGGAUAUACAUAUAGAUAUAUCCAUCGGAC (((((((((.(((((((((((........))))))).(((((....))))).)))).))))))((((((((((..(((....)))..))))))))))))).. ( -32.30) >DroSim_CAF1 63927 102 - 1 CUGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGGAUAUGUGGAUAUACAUAUAGAUAUAUCCAUCGGAC (((((((((.(((((((((((........))))))).(((((....))))).)))).))))))((((((((((..(((....)))..))))))))))))).. ( -32.30) >DroEre_CAF1 72790 99 - 1 CUGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGGAUAUACU--CGUACGUAUAGAUAUAUCCAUUGG-U ...((((((.(((((((((((........))))))).(((((....))))).)))).))))))(((((((((.(--(.........)))))))))))...-. ( -29.30) >DroYak_CAF1 69058 100 - 1 CCGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGGAUAUACU--CGUACAUAUGGAUAUAUCCAUUGGAC (((((((((.(((((((((((........))))))).(((((....))))).)))).))))))(((((((((..--(((....)))..)))))))))))).. ( -34.10) >DroAna_CAF1 16980 81 - 1 CCGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUACCGGC-CGCAAUGGAGUGCAUA--------------------UAUCCGUAGGAU ((((((......(.(((((((........))))))).)((........)))))-)...(((((.......--------------------..))))).)).. ( -20.50) >consensus CUGGCCAUAAUAAGAAAUCAUGUUUGGCAAUGAUUUGUGGCAUCGAUGCCGGUUUGCUGUGGCGUGGAUAU_____UAUACAUAUAGAUAUAUCCAUCGGAC (((((((((.(((((((((((........))))))).(((((....))))).)))).))))))(((((((((................)))))))))))).. (-20.77 = -21.94 + 1.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:36:29 2006