
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 14,498,728 – 14,498,886 |
| Length | 158 |
| Max. P | 0.981889 |

| Location | 14,498,728 – 14,498,820 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.09 |
| Mean single sequence MFE | -27.82 |
| Consensus MFE | -19.73 |
| Energy contribution | -19.43 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959342 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
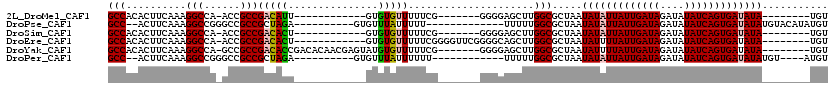
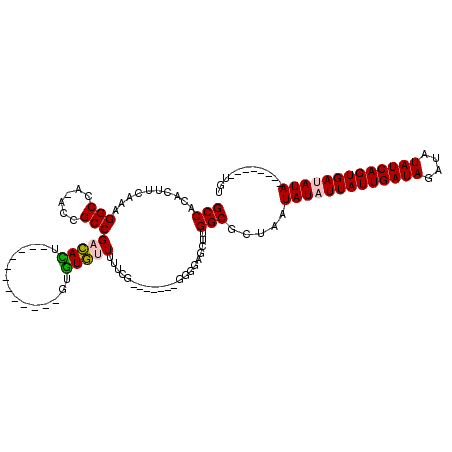
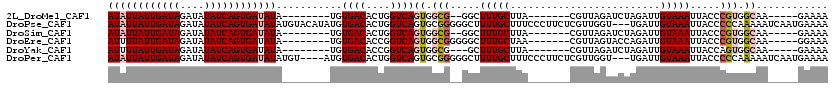
>2L_DroMel_CAF1 14498728 92 + 22407834 GCCACACUUCAAAGGCCA-ACCGCCGACAUU------------GUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGU ((((..((((...(((..-...)))(((((.------------..))))).....-------..))))..))))....((((((((((((((....)))))))))))))--------).. ( -28.60) >DroPse_CAF1 16718 95 + 1 GCC--ACUUCAAAGGCCGGGCCGCCGCUAGA----------GUGUUUAUUUUU-------------UUUUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGUACAUAUGU (((--........)))..(((.((((..(((----------(.........))-------------))..))))))).((((((((((((((....)))))))))))))).......... ( -24.90) >DroSim_CAF1 14467 92 + 1 GCCACACUUCAAAGGCCA-ACCGCCGACACU------------GUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGU ((((..((((...(((..-...)))(((((.------------..))))).....-------..))))..))))....((((((((((((((....)))))))))))))--------).. ( -29.90) >DroEre_CAF1 24470 99 + 1 GCCACACUUCAAAGGCCA-ACCGCCGACACU------------GUGUGUUUUUCGGGGUUCGGGGCAGCUUGGCGCUAAUAUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGU ((((..((....))((((-(((.(((((((.------------..)))....))))))))...)))....))))....((((.(((((((((....))))))))).)))--------).. ( -27.10) >DroYak_CAF1 19683 104 + 1 GCCACACUUCAAAGGCCA-GCCGCCGACACCGACACAACGAGUAUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGU ((((..((((...((...-.)).((((....((((((.......))))))..)))-------).))))..))))....((((.(((((((((....))))))))).)))--------).. ( -31.60) >DroPer_CAF1 16769 92 + 1 GCC--ACUUCAAAGGCCGGGCCGCCGCUAGA----------GUGUUUAUUUUUU------------UUUUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGU----AUGU (((--........)))..(((.((((..(((----------(..........))------------))..))))))).((((((((((((((....))))))))))))))..----.... ( -24.80) >consensus GCCACACUUCAAAGGCCA_ACCGCCGACACU____________GUGUGUUUUUCG_______GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA________UGU (((..........(((......)))(((((...............))))).....................))).....(((((((((((((....)))))))))))))........... (-19.73 = -19.43 + -0.30)
| Location | 14,498,758 – 14,498,851 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.27 |
| Mean single sequence MFE | -29.97 |
| Consensus MFE | -16.10 |
| Energy contribution | -16.77 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.981889 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
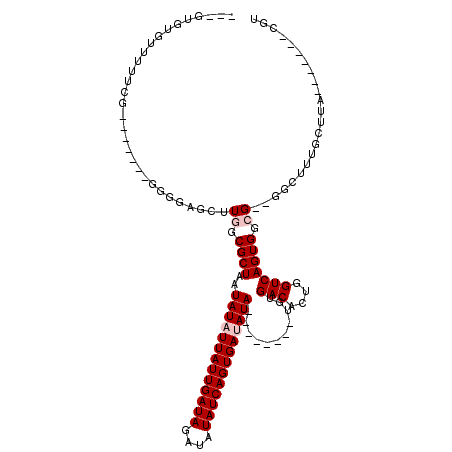
>2L_DroMel_CAF1 14498758 93 + 22407834 ---GUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACUGGUCAGUGGCG--GGCUUUGCUUA-------CGU ---...........(-------(.((((((((.((((.((((((((((((((....)))))))))))))--------)..(((....))))))).))--)))))).))..-------... ( -33.50) >DroPse_CAF1 16747 106 + 1 -GUGUUUAUUUUU-------------UUUUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGUACAUAUGUGACACUGGUCAGUGCGGGGGCUUUUGCUUUCCCUUCUCGU -............-------------......(((((.((((((((((((((....))))))))))))))..........(((....))))))))((((((....)))..)))....... ( -28.90) >DroSim_CAF1 14497 93 + 1 ---GUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACUGGUCAGUGGCG--GGCUUUGCUUA-------CGU ---...........(-------(.((((((((.((((.((((((((((((((....)))))))))))))--------)..(((....))))))).))--)))))).))..-------... ( -33.50) >DroEre_CAF1 24500 102 + 1 ---GUGUGUUUUUCGGGGUUCGGGGCAGCUUGGCGCUAAUAUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACCGGUCAGUGGCGGGGGCUUUGCUAA-------CGU ---..............(((((((((..((((.((((.((((.(((((((((....))))))))).)))--------)..(((....))))))).)))).))))))..))-------).. ( -27.90) >DroYak_CAF1 19722 95 + 1 AGUAUGUGUUUUUCG-------GGGGAGCUUGGCGCUAAUAUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACCGGUCAGUGGCG---GCUUUGCUUA-------CGU .............((-------...((((..(((((((((((.(((((((((....))))))))).)))--------).((((....)))).)))).---)))..)))).-------)). ( -27.10) >DroPer_CAF1 16798 103 + 1 -GUGUUUAUUUUUU------------UUUUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGU----AUGUGACACUGGUCAGUGCGGGGGCUUUUGCUUUCCCUUCUCGU -.............------------......(((((.((((((((((((((....))))))))))))))..----....(((....))))))))((((((....)))..)))....... ( -28.90) >consensus ___GUGUGUUUUUCG_______GGGGAGCUUGGCGCUAAUAUAUUAUUGAUAGAUAUAUCAGUGAUAUA________UGUGACACUGGUCAGUGGCG__GGCUUUGCUUA_______CGU ..............................((.((((..(((((((((((((....)))))))))))))...........(((....))))))).))....................... (-16.10 = -16.77 + 0.67)

| Location | 14,498,788 – 14,498,886 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -27.70 |
| Consensus MFE | -15.23 |
| Energy contribution | -15.73 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.789479 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
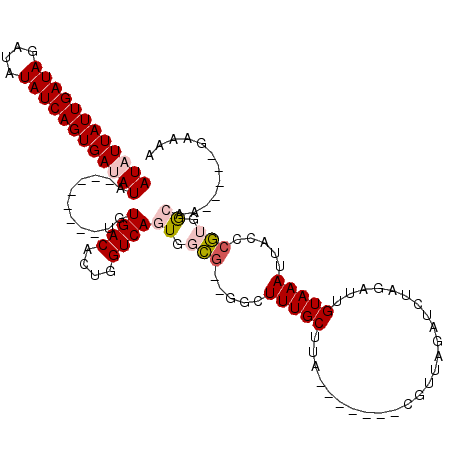
>2L_DroMel_CAF1 14498788 98 + 22407834 AUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACUGGUCAGUGGCG--GGCUUUGCUUA-------CGUUAGAUCUAGAUUGUAAAUUACCCGUGGCAA-----GAAAA ((((((((((((....)))))))))))).--------..((((....))))((.(((--((.(((((...-------..(((....)))...)))))...))))).))..-----..... ( -26.90) >DroPse_CAF1 16773 117 + 1 AUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGUACAUAUGUGACACUGGUCAGUGCGGGGGCUUUUGCUUUCCCUUCUCGUUGGU---UGAUUGUAAAUUACCCCCAAAAAUCAAUGAAAA ...((((((((......(((((((.((((((....)))))).)))))))......(((((..(((((..((((........)).---.))..)))))...)))))....))))))))... ( -34.10) >DroSim_CAF1 14527 98 + 1 AUAUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACUGGUCAGUGGCG--GGCUUUGCUUA-------CGUUAGAUCUAGAUUGUAAAUUACCCGUGGCAA-----GAAAA ((((((((((((....)))))))))))).--------..((((....))))((.(((--((.(((((...-------..(((....)))...)))))...))))).))..-----..... ( -26.90) >DroEre_CAF1 24537 100 + 1 AUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACCGGUCAGUGGCGGGGGCUUUGCUAA-------CGUUAGUACCAGAUUGUAAAUUACCCGUGGCAA-----GGAAA ...(((((((((....)))))))))....--------..((((....))))((.(((((...(((((...-------...............)))))...))))).))..-----..... ( -24.87) >DroYak_CAF1 19755 97 + 1 AUUUUAUUGAUAGAUAUAUCAGUGAUAUA--------UGUGACACCGGUCAGUGGCG---GCUUUGCUUA-------CGUUAGAUCUAGAUUGUAAAUUACCAGUGGCAA-----GAAAA ...(((((((((....)))))))))....--------..........((((.(((..---(.(((((...-------..(((....)))...))))))..))).))))..-----..... ( -19.70) >DroPer_CAF1 16825 113 + 1 AUAUUAUUGAUAGAUAUAUCAGUGAUAUAUGU----AUGUGACACUGGUCAGUGCGGGGGCUUUUGCUUUCCCUUCUCGUUGGU---UGAUUGUAAAUUACCCCCAAAAAUCAAUGAAAA ((((((((((((....))))))))))))..((----((.((((....))))))))(((((..(((((..((((........)).---.))..)))))...)))))............... ( -33.70) >consensus AUAUUAUUGAUAGAUAUAUCAGUGAUAUA________UGUGACACUGGUCAGUGGCG__GGCUUUGCUUA_______CGUUAGAUCUAGAUUGUAAAUUACCCGUGGCAA_____GAAAA ((((((((((((....))))))))))))...........((((....))))((.(((.....(((((.........................))))).....))).))............ (-15.23 = -15.73 + 0.50)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:35:04 2006