
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 14,374,326 – 14,374,441 |
| Length | 115 |
| Max. P | 0.998976 |

| Location | 14,374,326 – 14,374,441 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.21 |
| Mean single sequence MFE | -38.78 |
| Consensus MFE | -31.68 |
| Energy contribution | -32.60 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.86 |
| Structure conservation index | 0.82 |
| SVM decision value | 3.31 |
| SVM RNA-class probability | 0.998976 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
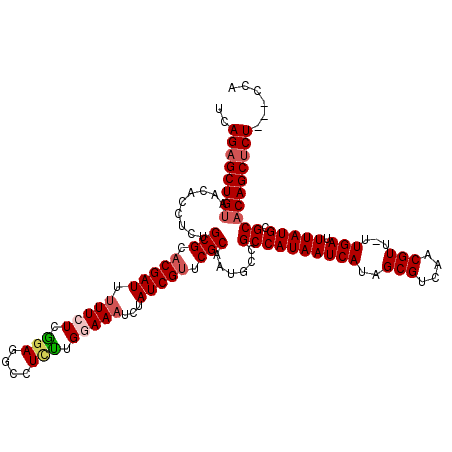
>2L_DroMel_CAF1 14374326 115 + 22407834 UCAGAGCUGUAACACCUAGGCGCACGAUUUUUCUCGGAGGCCU-CUGGAAAUCUAUCGUUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUU-UUGAUUUAUGCGCACAGCUCU---CCA ..((((((((.........(((.(((((.((((.((((....)-)))))))...))))).)))......((((((((((.((((....))))-.))).))))).))))))))))---... ( -41.60) >DroSec_CAF1 22459 116 + 1 UCAGAGCUGUAACACCUCUGCGCACGAUUUUUCUCGGAGGCCUUUUGGAAAUCUAUCGUUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUU-UUGAUUUAUGCGCACAGCUCU---CCA ..((((((((........((((.(((((.(((((..((....))..)))))...))))).)))).....((((((((((.((((....))))-.))).))))).))))))))))---... ( -39.70) >DroSim_CAF1 24561 116 + 1 UCAGAGCUGUAACACCUCUGCGCACGAUUUUUCUCGGAGGCCUUUUGGAAAUCUAUCGUUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUU-UUGAUUUAUGCGCACAGCUCU---CCA ..((((((((........((((.(((((.(((((..((....))..)))))...))))).)))).....((((((((((.((((....))))-.))).))))).))))))))))---... ( -39.70) >DroEre_CAF1 23254 120 + 1 UCAGUGCUGUAACACCUCUGCGCACGAUUUUUCUCAGAGGCCUCUGGAAAUCGUAUCGUUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUUUUUGAUUUAUGCGCACAGCUCUCCUCCA ..((.(((((........((((.(((((.((((.((((....))))))))....))))).)))).....((((((((((.((((....))))..))).))))).))))))).))...... ( -37.10) >DroYak_CAF1 24042 117 + 1 UCAGAGCUGUAACACCUCUGCGCACGAUUUUUCUCGGAGGCCUCUGGAAAACCUAUCGCUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUUUUUGAUUUAUGCGCCCAGCUCU---CCA ..(((((((.........((((..(((((((((.((((....)))))))))...))))..)))).....((((((((((.((((....))))..))).))))).)).)))))))---... ( -35.80) >consensus UCAGAGCUGUAACACCUCUGCGCACGAUUUUUCUCGGAGGCCUCUUGGAAAUCUAUCGUUCGCAAUGCCGCCAUAAUCAUAGCGUCAACGUU_UUGAUUUAUGCGCACAGCUCU___CCA ..((((((((.........(((.(((((.(((((.(((....))).)))))...))))).)))......((((((((((..(((....)))...))).))))).))))))))))...... (-31.68 = -32.60 + 0.92)


| Location | 14,374,326 – 14,374,441 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.21 |
| Mean single sequence MFE | -36.24 |
| Consensus MFE | -31.48 |
| Energy contribution | -32.08 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916421 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 14374326 115 - 22407834 UGG---AGAGCUGUGCGCAUAAAUCAA-AACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAACGAUAGAUUUCCAG-AGGCCUCCGAGAAAAAUCGUGCGCCUAGGUGUUACAGCUCUGA ...---((((((((((((..(((((..-..((((..((((((....))))))((...)).))))...)))))....-((((...(((......)))...))))..))).))))))))).. ( -35.30) >DroSec_CAF1 22459 116 - 1 UGG---AGAGCUGUGCGCAUAAAUCAA-AACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAACGAUAGAUUUCCAAAAGGCCUCCGAGAAAAAUCGUGCGCAGAGGUGUUACAGCUCUGA ...---(((((((((.((((...((..-.....((.((((((....)))))).)).((((.(((((...((((.....((...))..)))).))))).)))))).))))))))))))).. ( -35.60) >DroSim_CAF1 24561 116 - 1 UGG---AGAGCUGUGCGCAUAAAUCAA-AACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAACGAUAGAUUUCCAAAAGGCCUCCGAGAAAAAUCGUGCGCAGAGGUGUUACAGCUCUGA ...---(((((((((.((((...((..-.....((.((((((....)))))).)).((((.(((((...((((.....((...))..)))).))))).)))))).))))))))))))).. ( -35.60) >DroEre_CAF1 23254 120 - 1 UGGAGGAGAGCUGUGCGCAUAAAUCAAAAACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAACGAUACGAUUUCCAGAGGCCUCUGAGAAAAAUCGUGCGCAGAGGUGUUACAGCACUGA ......((.((((((.((((...((........((.((((((....)))))).)).((((.(((((....((((((((....)))).)))).))))).)))))).)))))))))).)).. ( -36.80) >DroYak_CAF1 24042 117 - 1 UGG---AGAGCUGGGCGCAUAAAUCAAAAACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAGCGAUAGGUUUUCCAGAGGCCUCCGAGAAAAAUCGUGCGCAGAGGUGUUACAGCUCUGA ...---((((((((((((...............((.((((((....)))))).))(((((.(((((...(((((..((....)).)))))..))))).)))))..))))).))))))).. ( -37.90) >consensus UGG___AGAGCUGUGCGCAUAAAUCAA_AACGUUGACGCUAUGAUUAUGGCGGCAUUGCGAACGAUAGAUUUCCAAAAGGCCUCCGAGAAAAAUCGUGCGCAGAGGUGUUACAGCUCUGA ......((((((((((((...............((.((((((....)))))).))(((((.(((((...((((............))))...))))).)))))..))).))))))))).. (-31.48 = -32.08 + 0.60)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:33:00 2006