
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 14,186,229 – 14,186,353 |
| Length | 124 |
| Max. P | 0.943031 |

| Location | 14,186,229 – 14,186,321 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 71.77 |
| Mean single sequence MFE | -37.73 |
| Consensus MFE | -18.42 |
| Energy contribution | -17.95 |
| Covariance contribution | -0.47 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.49 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.943031 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
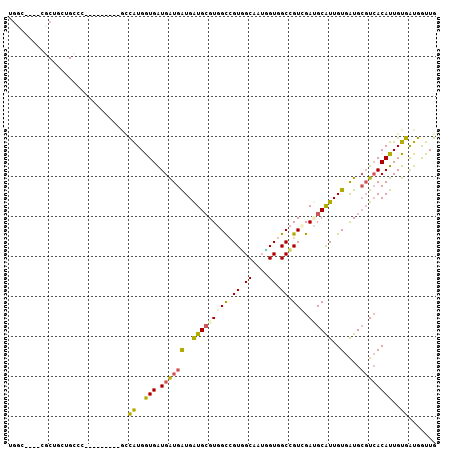
>2L_DroMel_CAF1 14186229 92 + 22407834 -GU-----UGCUGCUGCCC---------ACCAUGGUGGUGAUGGUGGUGCGUGGUCGUUGCAAUGGUGGCAGUCGAUGCAUUGUGGUGCGUCACAUUGUGAUGGUUG -..-----.((..(.((((---------(((((.......))))))).)))..))(((..(((((..(.....)(((((((....))))))).)))))..))).... ( -35.00) >DroVir_CAF1 28452 102 + 1 UGCC----CGCCAACGCCA-ACGAUAUGGUUAUGAUGAUGAUGAUGAUGCGUGGCCGUGGCAAUGGUGGCCGUGGAAGCGUUGUGAUGCGUCACAUUGUGGUGGUUG ...(----(((((..((((-......)))).(((.(((((((.(..((((....(((((((.......)))))))..))))..).)).))))))))..))))))... ( -35.80) >DroGri_CAF1 103 106 + 1 CAAA-CCACUCUGCUGCCCAACAGCGAUGCCAUGGUGAUGAUGAUGAUGCGUUGCUGUCGCUAUGGUUGCCGUGGAGGCAUUGUGAUGCGUGACAUUGUGAUGAUUG ....-((((..(((((.....)))))..(((((((((((..(((((...)))))..)))))))))))....))))...(((((..(((.....)))..))))).... ( -34.40) >DroEre_CAF1 18778 92 + 1 -GU-----UGCUGCUGCCC---------ACCAUGGUGGUGAUGGUGGUGCGUGGUCGUGGCAAUUGUGGCCGUCGAUGCAUUGUGGUGCGUCACAUUGUGAUGGUUG -((-----..(.((..(.(---------(((((.((....)).)))))).)..)).)..))......((((((((((((((....)))))))((...))))))))). ( -36.50) >DroMoj_CAF1 29987 105 + 1 UGGC-CAACGCCCACGCCC-ACGCCCACGCCAUGAUGAUGAUGAUGAUGCGUGGCCGUGGCAAUCGUCGCCGUGGAUGCAUUGUGAUGCGUCACAUUGUGAUGAUUG .(((-....))).....((-(((.(((((((((.((....)).)))..)))))).)))))((((((((((.((((((((((....))))))).))).)))))))))) ( -47.90) >DroPer_CAF1 24886 90 + 1 UGGCC---GACUGCUG--------------CGUGGUGAUGGUGAUGGUGGGUGGCCGUGGCAAGCGUGGCCGUCGACGCGUUGUGGUGGUUCACAUUGUGAUGGUUG .((((---(((.((..--------------(((.((.(((((.((.....)).))))).))..)))..)).)))..((((.(((((....))))).))))..)))). ( -36.80) >consensus UGGC____CGCUGCUGCCC_________GCCAUGGUGAUGAUGAUGAUGCGUGGCCGUGGCAAUGGUGGCCGUCGAUGCAUUGUGAUGCGUCACAUUGUGAUGGUUG ..............................((..(((.((((((..(((((((((.((.((....)).)).))).))))))..)....))))))))..))....... (-18.42 = -17.95 + -0.47)


| Location | 14,186,242 – 14,186,353 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.49 |
| Mean single sequence MFE | -41.92 |
| Consensus MFE | -18.62 |
| Energy contribution | -18.29 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.44 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.748152 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
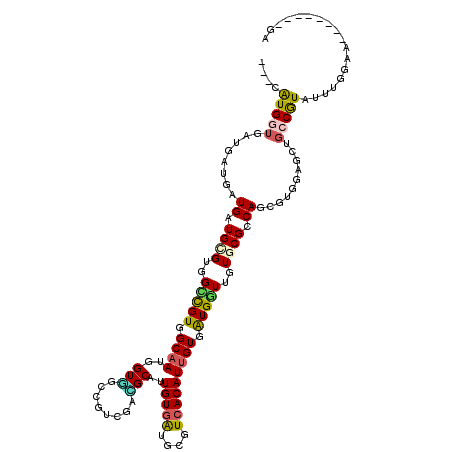
>2L_DroMel_CAF1 14186242 111 + 22407834 -ACCAUGGUGGUGAUGGUGGUGCGUGGUCGUUGCAAUGGUGGCAGUCGAUGCAUUGUGGUGCGUCACAUUGUGAUGGUUGUGCGCCAACGUUGAGCUGCCGUAUUUGGAA--------GA -.((((((..((((((.(((((((..(((((..(((((..(.....)(((((((....))))))).)))))..)))))..))))))).))))..))..)))....)))..--------.. ( -46.50) >DroVir_CAF1 28474 120 + 1 GGUUAUGAUGAUGAUGAUGAUGCGUGGCCGUGGCAAUGGUGGCCGUGGAAGCGUUGUGAUGCGUCACAUUGUGGUGGUUGUGCGCCAGCGUGGUGCUGCCGUAUUUGCUGCUGCUGCCGA (((.(((.(((((((.(..((((....(((((((.......)))))))..))))..).)).)))))))).(..(..((.(((((.((((.....)))).)))))..))..)..).))).. ( -46.20) >DroPse_CAF1 24935 109 + 1 ---CGUGGUGAUGGUGAUGGUGGGUGGCCGUGGCAAGCGUGGCCGUCGACGCGUUGUGGUGGUUCACAUUGUGAUGGUUGUGCGCCAGUGUCGAGCUGCCGUAUUUGGAA--------GA ---.(..((..((..(.(((((.(..(((((.((((((((.(....).))))..(((((....))))))))).)))))..).))))).)..)).))..)...........--------.. ( -40.20) >DroGri_CAF1 129 120 + 1 UGCCAUGGUGAUGAUGAUGAUGCGUUGCUGUCGCUAUGGUUGCCGUGGAGGCAUUGUGAUGCGUGACAUUGUGAUGAUUGUGCGCCAGCGUGCUGCUGCCAUAUUUGCUGCUGUUGCUGA .((((..((.((.(..(((((((((..(((((.((((((...)))))).))))..)..))))))..)))..).)).))..)).))(((((.((.((.((.......)).)).))))))). ( -43.80) >DroWil_CAF1 29033 111 + 1 -AUGAUGAUGGUGAUGAUGAUGUGUGGUUGUCGCAAUGGUCGCUGUCGAGGCAUUGUGAUGAGUCACAUUGUGAUGAUUGUGCGCCAAGGUGGAUGAUCCAUAUUUGGAU--------GA -......(..((.((.(..(((((.(.(..((((((((.((........))))))))))..).))))))..).)).))..).(((((((((((.....)))).))))).)--------). ( -34.60) >DroPer_CAF1 24899 109 + 1 ---CGUGGUGAUGGUGAUGGUGGGUGGCCGUGGCAAGCGUGGCCGUCGACGCGUUGUGGUGGUUCACAUUGUGAUGGUUGUGCGCCAGUGUCGAGCUGCCGUAUUUGGAA--------GA ---.(..((..((..(.(((((.(..(((((.((((((((.(....).))))..(((((....))))))))).)))))..).))))).)..)).))..)...........--------.. ( -40.20) >consensus ___CAUGGUGAUGAUGAUGAUGCGUGGCCGUGGCAAUGGUGGCCGUCGACGCAUUGUGAUGCGUCACAUUGUGAUGGUUGUGCGCCAGCGUGGAGCUGCCGUAUUUGGAA________GA ....(((((........((.((((..(((((.((((..(((........)))..(((((....))))))))).)))))..)))).))..........))))).................. (-18.62 = -18.29 + -0.33)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:31:40 2006