
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,944,833 – 13,944,966 |
| Length | 133 |
| Max. P | 0.997703 |

| Location | 13,944,833 – 13,944,942 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.10 |
| Mean single sequence MFE | -32.03 |
| Consensus MFE | -19.11 |
| Energy contribution | -18.27 |
| Covariance contribution | -0.84 |
| Combinations/Pair | 1.35 |
| Mean z-score | -3.50 |
| Structure conservation index | 0.60 |
| SVM decision value | 2.91 |
| SVM RNA-class probability | 0.997703 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13944833 109 - 22407834 GGGCCAAGAACUCCAGGUUUAGGAUAAAUAC-CAGCUAACUGUUUGUAAA----UUUCGCCA-UUUAUGAACAG--UUUAGCUGGCCA--AGUUUAUCAGUGAAAGGUUUCGG-CGCACA (.(((..(((..((...((((.(((((((.(-((((((((((((..((((----(......)-))))..)))))--.))))))))...--.)))))))..)))).)).)))))-).)... ( -37.40) >DroVir_CAF1 42769 111 - 1 GU---AUGAAAUAAUAGCUUAGGAUAAAUUC-CAGCUAAUUGUUUUUAAAAGAAUUAACAAAUUUUAUAAACAU--UUUAGCUGCUGU--UGUUUAUCAGUGAAAGGUUUCAA-CGUACA ((---(((((((((((((...(((.....))-)((((((.(((((.(((((...........))))).))))).--.)))))))))))--))))).....((((....)))).-))))). ( -29.60) >DroPse_CAF1 38677 105 - 1 GG---AUGAGUGUCCCGUUUAGGGUAAACAG-CAGCUAAAUGUUCGUAAA----UUCCCAUA-UUUAUGGACAU--UUUAGCUG-UCA--CGUUUAUCAGUGAAAGGUUUCAA-CGCACA ..---....((((.((.((((.(((((((.(-((((((((((((((((((----(......)-)))))))))).--))))))))-)..--.)))))))..)))).)).....)-)))... ( -34.00) >DroWil_CAF1 42995 113 - 1 GUG--AUGAAAUUCAAAUUUAGGAUAAAUUCUCAGUUAAGUGUUUGUAAA----UUCAUAUA-UUUAUGAACAUUCUUUAGCUGUGCGUUAGUUUAUCAGUGAAAGGUUUCAAAAGCACA (((--.(((((((....((((.(((((((((.((((((((((((..((((----(......)-))))..)))))...))))))).)....))))))))..)))).)))))))....))). ( -27.00) >DroAna_CAF1 33028 107 - 1 G--CUUGAAAUUCUAUGUUUAGGAUAAAUAC-CAGCUAAAUGUUUGUAGA----UUUUGCCA-UUUAUGGACAA--CUUAGCUGGCCA--AGUUUAUCAGUUAAAGGUUUCAC-CGCACA (--(.((((((((((....)))(((((((.(-(((((((.((((..((((----(......)-))))..)))).--.))))))))...--.))))))).......))))))).-.))... ( -30.20) >DroPer_CAF1 40605 105 - 1 GG---AUGAGUGUCCCGUUUAGGGUAAACAG-CAGCUAAAUGUUCGUAAA----UUCCCAUA-UUUAUGGACAU--UUUAGCUG-UCA--CGUUUAUCAGUGAAAGGUUUCAA-CGCACA ..---....((((.((.((((.(((((((.(-((((((((((((((((((----(......)-)))))))))).--))))))))-)..--.)))))))..)))).)).....)-)))... ( -34.00) >consensus GG___AUGAAUUUCAAGUUUAGGAUAAAUAC_CAGCUAAAUGUUUGUAAA____UUCCCAUA_UUUAUGAACAU__UUUAGCUG_UCA__AGUUUAUCAGUGAAAGGUUUCAA_CGCACA ......................(((((((...(((((((((((((((((((...........))))))))))))...))))))).......)))))))...................... (-19.11 = -18.27 + -0.84)


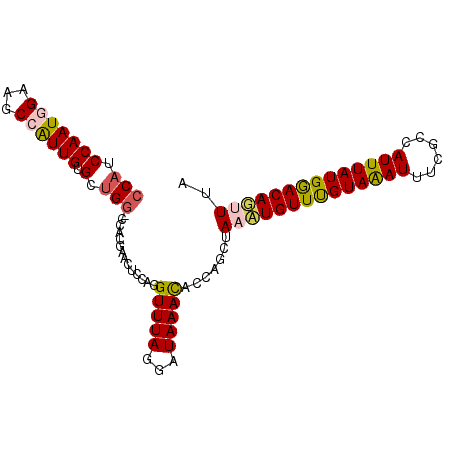
| Location | 13,944,870 – 13,944,966 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 82.23 |
| Mean single sequence MFE | -24.90 |
| Consensus MFE | -15.82 |
| Energy contribution | -15.68 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611137 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13944870 96 - 22407834 CCAUCCAAUGGAAGCCAUUGUGCUGGGCCAAGAACUCCAGGUUUAGGAUAAAUACCAGCUAACUGUUUGUAAAUUUCGCCAUUUAUGAACAGUUUA .....((((((...)))))).(((((.((..((((.....)))).)).......))))).(((((((..(((((......)))))..))))))).. ( -26.00) >DroPse_CAF1 38713 93 - 1 CCAUCCAAUGGAAGCAGUUGUGUUGG---AUGAGUGUCCCGUUUAGGGUAAACAGCAGCUAAAUGUUCGUAAAUUCCCAUAUUUAUGGACAUUUUA .(((((((((.((....)).))))))---))).((..(((.....)))...)).......((((((((((((((......)))))))))))))).. ( -28.60) >DroEre_CAF1 33866 96 - 1 CCAUCCAAUGGAAGCCAUUGUGCUGGGCCACGAACUCCAGGUUUAGGAUAAAUAGCAGCUAACUGUUUGUAAAUUUCACCAUUUAUGGACAGUUUA ((((...))))..((..((((.(((((((..........))))))).))))...))....(((((((..(((((......)))))..))))))).. ( -24.90) >DroYak_CAF1 34211 96 - 1 CCAUCCAAUGGAAGCCAUUGUGCUGGGCCACGAACUCCAGGUUUAGGAUAAACACCAGCUAACUGUUUGUAAAUUUCGCCAUUUAUGGACAGCUUA .....((((((...)))))).(((((.((..((((.....)))).)).......)))))...(((((..(((((......)))))..))))).... ( -24.60) >DroAna_CAF1 33065 94 - 1 CCAUCCAAUGGAAGCCAUUGUGUCG--CUUGAAAUUCUAUGUUUAGGAUAAAUACCAGCUAAAUGUUUGUAGAUUUUGCCAUUUAUGGACAACUUA .....((((((...))))))....(--((.(..((((((....)))))).....).)))....((((..(((((......)))))..))))..... ( -16.70) >DroPer_CAF1 40641 93 - 1 CCAUCCAAUGGAAGCAGUUGUGUUGG---AUGAGUGUCCCGUUUAGGGUAAACAGCAGCUAAAUGUUCGUAAAUUCCCAUAUUUAUGGACAUUUUA .(((((((((.((....)).))))))---))).((..(((.....)))...)).......((((((((((((((......)))))))))))))).. ( -28.60) >consensus CCAUCCAAUGGAAGCCAUUGUGCUGG_CCACGAACUCCAGGUUUAGGAUAAACACCAGCUAAAUGUUUGUAAAUUUCGCCAUUUAUGGACAGUUUA (((.(((((((...)))))).).)))..............(((((...))))).......((((((((((((((......)))))))))))))).. (-15.82 = -15.68 + -0.14)


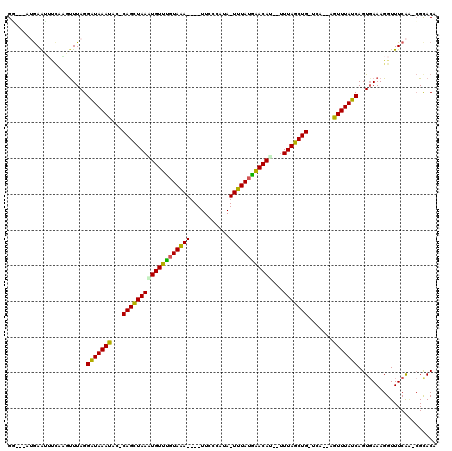
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:30:04 2006