
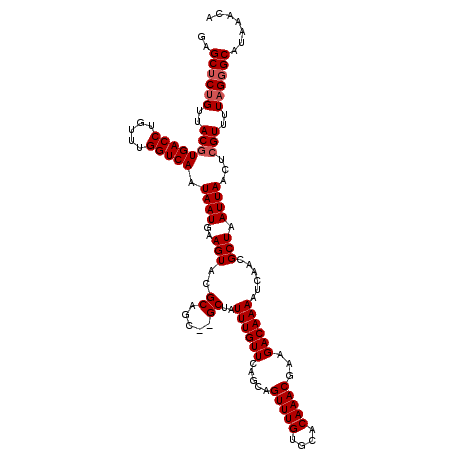
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,760,665 – 13,760,823 |
| Length | 158 |
| Max. P | 0.667936 |

| Location | 13,760,665 – 13,760,783 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.75 |
| Mean single sequence MFE | -29.50 |
| Consensus MFE | -26.28 |
| Energy contribution | -26.78 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.29 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.667936 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
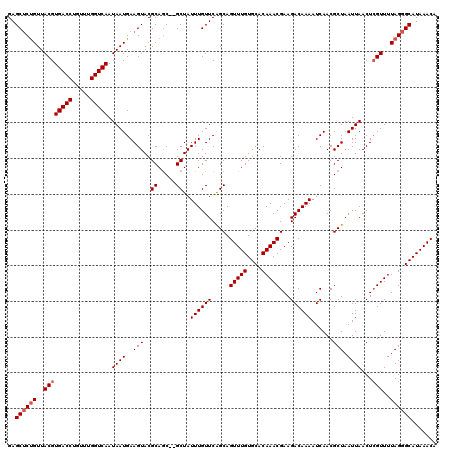
>2L_DroMel_CAF1 13760665 118 - 22407834 GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC--GCUAUUUGUUCGGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....)))))..............(((--(...((((((.....(((((....)))))...)))))).....))))........)))..))))))....... ( -30.10) >DroSec_CAF1 24253 118 - 1 GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC--GCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....)))))..............(((--(...((((((.....(((((....)))))...)))))).....))))........)))..))))))....... ( -30.10) >DroAna_CAF1 45322 120 - 1 GAGCCCCGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAACGAGCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUGGUUUUAGAGCAUAAACA ((((..(((((..(((((.....))))).)))))(((((.((.....)))))))))))....((((((((.....((..((.....))..))((((..........)))).)))))))). ( -26.50) >DroSim_CAF1 27299 118 - 1 GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC--GCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....)))))..............(((--(...((((((.....(((((....)))))...)))))).....))))........)))..))))))....... ( -30.10) >DroEre_CAF1 25944 118 - 1 GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC--GCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....)))))..............(((--(...((((((.....(((((....)))))...)))))).....))))........)))..))))))....... ( -30.10) >DroYak_CAF1 27376 118 - 1 GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC--GCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....)))))..............(((--(...((((((.....(((((....)))))...)))))).....))))........)))..))))))....... ( -30.10) >consensus GAGCUCUGUUACGUGACCUGUUUGGUCAAUAAUGAAGUACGCAGC__GCUAUUUGUUCAGCAGUUUGUGCACAAACGAAGACAAAAUCAACGCUAAUUAACUCGUUUUAGGGCAUAAACA ..((((((..((((((((.....))))).((((..(((..((.....))..((((((.....(((((....)))))...))))))......))).))))...)))..))))))....... (-26.28 = -26.78 + 0.50)

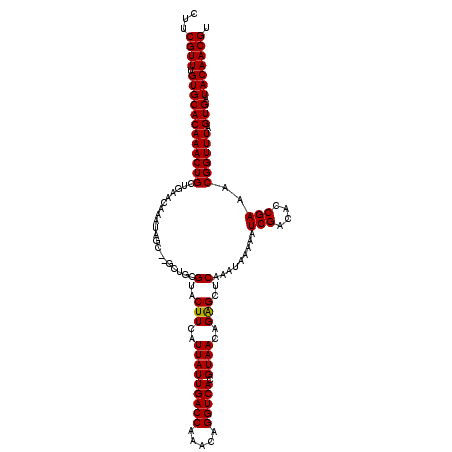
| Location | 13,760,705 – 13,760,823 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.31 |
| Mean single sequence MFE | -25.77 |
| Consensus MFE | -23.52 |
| Energy contribution | -23.38 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.05 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.578621 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
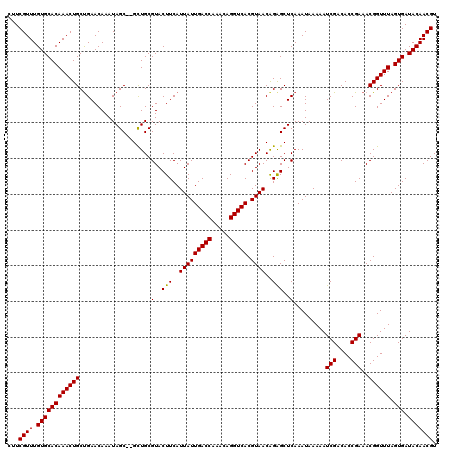
>2L_DroMel_CAF1 13760705 118 + 22407834 CUUCGUUUGUGCACAAACUGCCGAACAAAUAGC--GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((..........(((--.(((.........(((((((((.....))))).))))))).)))..........(((....)))..)))))).))).))))))). ( -24.70) >DroSec_CAF1 24293 118 + 1 CUUCGUUUGUGCACAAACUGCUGAACAAAUAGC--GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((..........(((--.(((.........(((((((((.....))))).))))))).)))..........(((....)))..)))))).))).))))))). ( -24.70) >DroAna_CAF1 45362 120 + 1 CUUCGUUUGUGCACAAACUGCUGAACAAAUAGCUCGUUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACGGGGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((............((((((((((...........((((.....)))))))))))))).............(((....)))..)))))).))).))))))). ( -31.10) >DroSim_CAF1 27339 118 + 1 CUUCGUUUGUGCACAAACUGCUGAACAAAUAGC--GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((..........(((--.(((.........(((((((((.....))))).))))))).)))..........(((....)))..)))))).))).))))))). ( -24.70) >DroEre_CAF1 25984 118 + 1 CUUCGUUUGUGCACAAACUGCUGAACAAAUAGC--GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((..........(((--.(((.........(((((((((.....))))).))))))).)))..........(((....)))..)))))).))).))))))). ( -24.70) >DroYak_CAF1 27416 118 + 1 CUUCGUUUGUGCACAAACUGCUGAACAAAUAGC--GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((..........(((--.(((.........(((((((((.....))))).))))))).)))..........(((....)))..)))))).))).))))))). ( -24.70) >consensus CUUCGUUUGUGCACAAACUGCUGAACAAAUAGC__GCUGCGUACUUCAUUAUUGACCAAACAGGUCACGUAACAGAGCUCAAAUAAAAAUCGACACCGAAACGGUUUAGUGAUACAACGU ...((((.((((((((((((....................(..(((..(((((((((.....))))).))))..)))..).........(((....)))..)))))).))).))))))). (-23.52 = -23.38 + -0.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:28:51 2006