
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,390,019 – 13,390,136 |
| Length | 117 |
| Max. P | 0.527856 |

| Location | 13,390,019 – 13,390,136 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 78.75 |
| Mean single sequence MFE | -40.05 |
| Consensus MFE | -19.66 |
| Energy contribution | -21.33 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.49 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.523596 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13390019 117 + 22407834 GCCGCUCUCGCUCUUUAUGCGCAUUUGCCGCAUUGAGCGCGAAGCCAACGACGAUUUGCGUCUACAGCUUCAGCAUCCCAUCCGUCAGCACUAUCUGCUUUCCCAGCUGAAGCUGGA ...(((.((((.(((.((((((....).))))).))).)))))))....((((.....))))..((((((((((....(....)..((((.....))))......)))))))))).. ( -41.20) >DroVir_CAF1 2467 117 + 1 GCCGCUGUCGCUGAUGUUGCGUAUAUUUCGCGUUGAGCGGGAUAUUAUGGACAAAUUGCGACCACAACUGCAGCAUCCGGUGCGUCAGCAUUAUCUGCUUAGCCAGUUGAAACUGCA ...((.(((((...((((.(((((((..(((.....)))..))).))))))))....)))))..(((((((((((...(((((....)))))...))))..).)))))).....)). ( -39.00) >DroSec_CAF1 2226 117 + 1 GCCGCUCUCGCUCUUUAUGCGCAUUUGCCGCAUUGAACGCGAAGCCAGCGACGAUUUGCGUCUACAGCUUCAGCAUCCCAUCCGUCAGCACUAUCUACUUUCCCAGCUGAAGCUGGA ((.(((.((((..((.((((((....).))))).))..)))))))..))((((.....))))..((((((((((...............................)))))))))).. ( -36.58) >DroEre_CAF1 2232 117 + 1 GCCGCUCUCGCUCUUUAUGCGCAUUUGUCGCAUCGAGCGUGAAGCCAGCGAUGAGUUGCGCCUACAGCUUCAGCAUCCAAUCCGUCAACACUACCUGCUUUCCCAGCUCAAUCUGGA ((.((((.(((((...((((((....).))))).))))).).)))..((...((((((......))))))..))......................))....((((......)))). ( -29.70) >DroYak_CAF1 2242 117 + 1 GCCGCUCUCGCUCUUUCUGCGCAUCUGUCGCAUCGAGCGUGAAGCCAGCGACGAGUUGCGUCUGCAGCUUCAGCAUCCCAUCCGUCAGCACUACCUGCUGUCCCAGCUCAAGCUGGA ...((...(((((....(((((....).))))..)))))((((((..((((((.....)))).)).)))))))).........(.(((((.....))))).)(((((....))))). ( -41.70) >DroMoj_CAF1 2614 117 + 1 GCCACUCUCGCUGAUGCUGCGCAUCUUCCGCAUCGACCGAGAUGUGGUGGAGAAGCUGCGCCCGCAGCUGCAACACCCGGUGCGACAGCACUAUCUGCUCCAGCAGCUGCAGCUGCA ((((((((((.((((((............))))))..))))).)))))((((.((((((....)))))).........(((((....))))).....)))).(((((....))))). ( -52.10) >consensus GCCGCUCUCGCUCUUUAUGCGCAUUUGCCGCAUCGAGCGCGAAGCCAGCGACGAGUUGCGUCUACAGCUUCAGCAUCCCAUCCGUCAGCACUAUCUGCUUUCCCAGCUGAAGCUGGA ...((((.(((((....((((.......))))..))))).)).))...................((((((((((...(.....)..((((.....))))......)))))))))).. (-19.66 = -21.33 + 1.67)

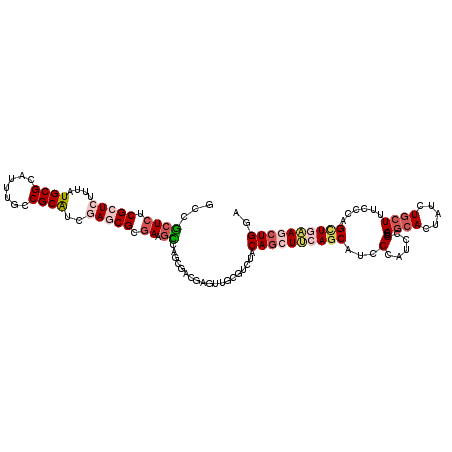
| Location | 13,390,019 – 13,390,136 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.75 |
| Mean single sequence MFE | -43.38 |
| Consensus MFE | -25.85 |
| Energy contribution | -26.25 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.527856 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13390019 117 - 22407834 UCCAGCUUCAGCUGGGAAAGCAGAUAGUGCUGACGGAUGGGAUGCUGAAGCUGUAGACGCAAAUCGUCGUUGGCUUCGCGCUCAAUGCGGCAAAUGCGCAUAAAGAGCGAGAGCGGC ((((((....))))))...(((.....(((((.((..((((.(((.((((((...((((.....))))...))))))))))))).)))))))..))).........((....))... ( -43.70) >DroVir_CAF1 2467 117 - 1 UGCAGUUUCAACUGGCUAAGCAGAUAAUGCUGACGCACCGGAUGCUGCAGUUGUGGUCGCAAUUUGUCCAUAAUAUCCCGCUCAACGCGAAAUAUACGCAACAUCAGCGACAGCGGC ((((((.((.....((..((((.....))))...))....)).))))))(((((.(((((....(((.....((((..(((.....)))..)))).....)))...))))).))))) ( -34.90) >DroSec_CAF1 2226 117 - 1 UCCAGCUUCAGCUGGGAAAGUAGAUAGUGCUGACGGAUGGGAUGCUGAAGCUGUAGACGCAAAUCGUCGCUGGCUUCGCGUUCAAUGCGGCAAAUGCGCAUAAAGAGCGAGAGCGGC ((((((....))))))...(((.....(((((.((....((((((.((((((...((((.....))))...))))))))))))..)))))))..))).........((....))... ( -42.50) >DroEre_CAF1 2232 117 - 1 UCCAGAUUGAGCUGGGAAAGCAGGUAGUGUUGACGGAUUGGAUGCUGAAGCUGUAGGCGCAACUCAUCGCUGGCUUCACGCUCGAUGCGACAAAUGCGCAUAAAGAGCGAGAGCGGC ..(((..((((.((.(...((((.(((((((.(.....).)))))))...))))...).)).))))...)))((((..(((((.(((((.(....))))))...))))).))))... ( -39.20) >DroYak_CAF1 2242 117 - 1 UCCAGCUUGAGCUGGGACAGCAGGUAGUGCUGACGGAUGGGAUGCUGAAGCUGCAGACGCAACUCGUCGCUGGCUUCACGCUCGAUGCGACAGAUGCGCAGAAAGAGCGAGAGCGGC ((((((....)))))).(((((.....)))))..........((((((((((((.((((.....)))))).)))))).(((((..((((.(....)))))....)))))..)))).. ( -46.40) >DroMoj_CAF1 2614 117 - 1 UGCAGCUGCAGCUGCUGGAGCAGAUAGUGCUGUCGCACCGGGUGUUGCAGCUGCGGGCGCAGCUUCUCCACCACAUCUCGGUCGAUGCGGAAGAUGCGCAGCAUCAGCGAGAGUGGC .(((((((((((..((((.((.((((....)))))).))))..)))))))))))(((.........))).((((.(((((.(.(((((............)))))).))))))))). ( -53.60) >consensus UCCAGCUUCAGCUGGGAAAGCAGAUAGUGCUGACGGAUGGGAUGCUGAAGCUGUAGACGCAACUCGUCGCUGGCUUCACGCUCAAUGCGACAAAUGCGCAUAAAGAGCGAGAGCGGC .(((((....)))))...((((.....))))...........(((((((((((..((((.....))))..))))))).(((((..((((.......))))....)))))..)))).. (-25.85 = -26.25 + 0.40)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:24:05 2006