
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,228,264 – 13,228,369 |
| Length | 105 |
| Max. P | 0.853698 |

| Location | 13,228,264 – 13,228,369 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 75.26 |
| Mean single sequence MFE | -36.77 |
| Consensus MFE | -15.98 |
| Energy contribution | -16.12 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.43 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.853698 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
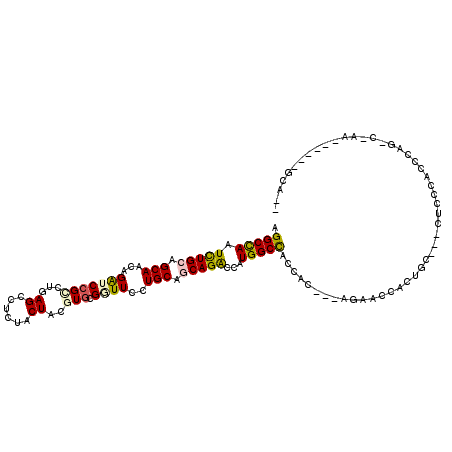
>2L_DroMel_CAF1 13228264 105 + 22407834 AGGCCAAUCUGCAGCAGCAGAUCCGCCUGAGUCUCUACUAUGUGCGCUUCCUGCAGCAGGCGAUGGCCUCCAC---GGAGCCACUGC---CUCCCACACAGCCAAG------GCACA ((((..((((((....))))))..))))..((((...((.((((.(((......)))(((((.((((.((...---.)))))).)))---))..)))).))...))------))... ( -39.80) >DroPse_CAF1 4134 106 + 1 AGGCCAAUCUGCAGCAACAGGUCCGGCUUAGCCUCUACUACGUGCGGUUCCUGCAGCAGACCAUGGCCGCCACCAGAGAACCUCUGCAGGCUUCCAGCCAU---CA------GCA-- .(((((.(((((.(((...((.(((((.(((......))).)).)))..))))).)))))...)))))(((..(((((...)))))..))).....((...---..------)).-- ( -36.80) >DroGri_CAF1 5136 112 + 1 AGGCUAAUUUGCAGCAACAGAUCCGUCUCAGCCUGUACUAUGUGCGGUUCCUGCAACAGACAAUGGCCACGACACGAACUCCACUGU---CUGACACACAAUCCACGCUGCAGCA-- ........(((((((..(((..((((..((..........)).))))...)))...((((((.(((...((...))....))).)))---))).............)))))))..-- ( -27.80) >DroYak_CAF1 4870 105 + 1 AGGCCAAUCUGCAGCAGCAGAUCCGCCUGAGCCUCUACUACGUCCGCUUCCUGCAGCAGGCGAUGGCUUCCAC---GGAGCCACUGC---CUCCCACGCAGUCGAG------GCACU ((((..((((((....))))))..))))..(((((.(((.(((..(((......)))(((((.((((((....---.)))))).)))---))...))).))).)))------))... ( -48.20) >DroAna_CAF1 5777 103 + 1 AGGCCAAUCUGGAGCAGCAGAUCCGCCUGAGCCUCUACUAUGUGCGAUUCCUGCAGCAGACCAUGGCCACCAU---GGAACCACUGG---CCCCUACCCACCACCA------GCA-- .(((((.((((..((((..(((((((...((......))..))).)))).))))..))))((((((...))))---))......)))---))..............------...-- ( -31.30) >DroPer_CAF1 4131 106 + 1 AGGCCAAUCUGCAGCAACAGGUCCGGCUAAGCCUCUACUACGUGAGGUUCCUGCAGCAGACCAUGGCCGCCACCAGAGAACCUCUGCAGGCUUCCAGCCAU---CA------GCA-- .(((((.(((((.(((...((.....)).((((((........))))))..))).)))))...)))))(((..(((((...)))))..))).....((...---..------)).-- ( -36.70) >consensus AGGCCAAUCUGCAGCAACAGAUCCGCCUGAGCCUCUACUACGUGCGGUUCCUGCAGCAGACCAUGGCCACCAC___AGAACCACUGC___CUCCCACCCAG_C_AA______GCA__ .(((((.(((((.(((...(((((((...((......))..))).))))..))).)))))...)))))................................................. (-15.98 = -16.12 + 0.14)

| Location | 13,228,264 – 13,228,369 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 75.26 |
| Mean single sequence MFE | -46.48 |
| Consensus MFE | -18.60 |
| Energy contribution | -19.30 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.40 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.709077 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13228264 105 - 22407834 UGUGC------CUUGGCUGUGUGGGAG---GCAGUGGCUCC---GUGGAGGCCAUCGCCUGCUGCAGGAAGCGCACAUAGUAGAGACUCAGGCGGAUCUGCUGCUGCAGAUUGGCCU ((.(.------(((.((((((((..((---((.(((((((.---...)).))))).))))(((......))).)))))))).))).).))(((.(((((((....))))))).))). ( -51.90) >DroPse_CAF1 4134 106 - 1 --UGC------UG---AUGGCUGGAAGCCUGCAGAGGUUCUCUGGUGGCGGCCAUGGUCUGCUGCAGGAACCGCACGUAGUAGAGGCUAAGCCGGACCUGUUGCUGCAGAUUGGCCU --.((------(.---...((..(((((((....))))).))..)))))(((((..((((((.(((((..(.((...((((....)))).)).)..)))))....))))))))))). ( -43.10) >DroGri_CAF1 5136 112 - 1 --UGCUGCAGCGUGGAUUGUGUGUCAG---ACAGUGGAGUUCGUGUCGUGGCCAUUGUCUGUUGCAGGAACCGCACAUAGUACAGGCUGAGACGGAUCUGUUGCUGCAAAUUAGCCU --...((((((((((.((.((((.(((---(((((((....((...))...)))))))))).)))).)).)))).......((((((((...))).))))).))))))......... ( -41.90) >DroYak_CAF1 4870 105 - 1 AGUGC------CUCGACUGCGUGGGAG---GCAGUGGCUCC---GUGGAAGCCAUCGCCUGCUGCAGGAAGCGGACGUAGUAGAGGCUCAGGCGGAUCUGCUGCUGCAGAUUGGCCU .(.((------(((.(((((((...((---((.(((((((.---...).)))))).)))).((((.....))))))))))).))))).)((((.(((((((....))))))).)))) ( -59.70) >DroAna_CAF1 5777 103 - 1 --UGC------UGGUGGUGGGUAGGGG---CCAGUGGUUCC---AUGGUGGCCAUGGUCUGCUGCAGGAAUCGCACAUAGUAGAGGCUCAGGCGGAUCUGCUGCUCCAGAUUGGCCU --...------....(((.(((.((((---(..((((((((---...(..((........))..).))))))))....((((((.((....))...)))))))))))..))).))). ( -37.90) >DroPer_CAF1 4131 106 - 1 --UGC------UG---AUGGCUGGAAGCCUGCAGAGGUUCUCUGGUGGCGGCCAUGGUCUGCUGCAGGAACCUCACGUAGUAGAGGCUUAGCCGGACCUGUUGCUGCAGAUUGGCCU --.((------..---(((((((..(((((...(((((((.((.(..(((((....).))))..)))))))))).(......)))))))))))....((((....))))))..)).. ( -44.40) >consensus __UGC______UG_G_AUGGGUGGGAG___GCAGUGGUUCC___GUGGAGGCCAUGGUCUGCUGCAGGAACCGCACAUAGUAGAGGCUCAGGCGGAUCUGCUGCUGCAGAUUGGCCU .................................................(((((..((((((.(((((..(((.....(((....)))....))).)))))....))))))))))). (-18.60 = -19.30 + 0.70)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:01 2006