
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,182,267 – 13,182,429 |
| Length | 162 |
| Max. P | 0.753104 |

| Location | 13,182,267 – 13,182,363 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 81.03 |
| Mean single sequence MFE | -33.88 |
| Consensus MFE | -16.73 |
| Energy contribution | -17.71 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.641116 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
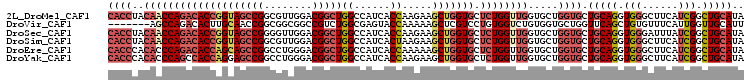
>2L_DroMel_CAF1 13182267 96 + 22407834 UGGUCCAUUGCACUGCGCUGCUGCGACUCCUGGCCAC---GGCUCAUCUGCAGCAGCCAUAUGCAGCCGAUGAAGCCCACCUGCAGCACCAGCACCAAC ((((.....((...))((((.(((..(....((....---((((((((.((.(((......))).)).)))).))))..)).)..))).)))))))).. ( -31.50) >DroPse_CAF1 3126 93 + 1 UGAUCCAGG---CUUUGCUGCUG---AUCCUGGCUGCUGUAGUUCAUCUGCAACAGCCAUAUGCAGCCUAUGAAGCCAACCUGAAGUAUCAGCACGGAC ...(((.((---(((((((((..---....((((((.(((((.....))))).))))))...)))))....))))))...((((....))))...))). ( -36.80) >DroGri_CAF1 2784 96 + 1 AGGUCCAGUUCAGGUUGCUGCUGCAUCUGCUGGCUGC---GAUUAUUCUGCAGCAGCCAAAUGCAGCCAAUGAAACCCAACUGGACCAGGACCACAGCC .(((((((((..((((...(((((((..((((.((((---((....)).))))))))...)))))))......)))).)))))))))............ ( -42.80) >DroSim_CAF1 2788 96 + 1 UGGUCCAUUGCACCGCGCUGCUGGGACUCCUGGCCAC---GACUCAUCUGCAGCAGCCAUAUGCAGCCGAUGAAGCCCACCUGCAGCACCAGCACCAAC ((((.....((.....)).(((((..((.(.((....---(..(((((.((.(((......))).)).)))))..)...)).).))..))))))))).. ( -28.80) >DroEre_CAF1 2819 96 + 1 UGGUCCACUGCACUCCGCUGCUGUGACUCCUGGCCAC---GGCUCAUCUGCAGCAGCCAUAUGCAGCCGAUGAAGCCCACCUGCAGCACCAGCACCAAC ((((...(((((....(((((((((.(.....).)))---)))(((((.((.(((......))).)).)))))))).....))))).))))........ ( -31.70) >DroYak_CAF1 2850 96 + 1 UGGUCCACUGCACUUCGCUGCUGUGACUCCUGGCCAC---GGCUCAUCUGCAGCAGCCAUAUGCAGCCGAUGAAGCCCACCUGCAGCACCAGCACCAAC ((((...(((((....(((((((((.(.....).)))---)))(((((.((.(((......))).)).)))))))).....))))).))))........ ( -31.70) >consensus UGGUCCACUGCACUUCGCUGCUGCGACUCCUGGCCAC___GGCUCAUCUGCAGCAGCCAUAUGCAGCCGAUGAAGCCCACCUGCAGCACCAGCACCAAC ((((.(..........(((((((((......((((.....))))....)))))))))....(((((..............)))))).))))........ (-16.73 = -17.71 + 0.98)


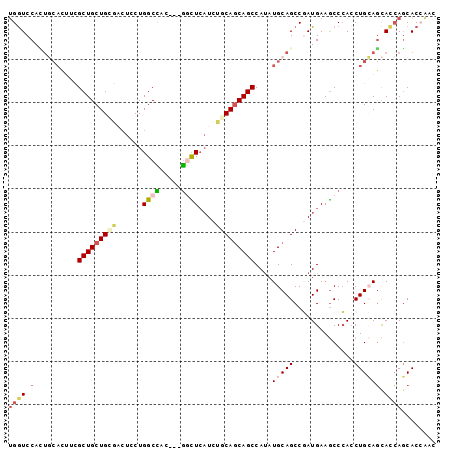
| Location | 13,182,267 – 13,182,363 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 81.03 |
| Mean single sequence MFE | -41.55 |
| Consensus MFE | -22.50 |
| Energy contribution | -23.23 |
| Covariance contribution | 0.73 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.753104 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13182267 96 - 22407834 GUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC---GUGGCCAGGAGUCGCAGCAGCGCAGUGCAAUGGACCA ..((((.(..(..((((..(..(.(......).)..).....((((((((.(((...((---.......)).)))))))))))))))..)...).)))) ( -43.00) >DroPse_CAF1 3126 93 - 1 GUCCGUGCUGAUACUUCAGGUUGGCUUCAUAGGCUGCAUAUGGCUGUUGCAGAUGAACUACAGCAGCCAGGAU---CAGCAGCAAAG---CCUGGAUCA ((((...((((....))))...(((((.....(((((...(((((((((.((.....)).)))))))))....---..))))).)))---)).)))).. ( -36.90) >DroGri_CAF1 2784 96 - 1 GGCUGUGGUCCUGGUCCAGUUGGGUUUCAUUGGCUGCAUUUGGCUGCUGCAGAAUAAUC---GCAGCCAGCAGAUGCAGCAGCAACCUGAACUGGACCU (((....)))..((((((((((((((.(....((((((((((.(((((((.((....))---)))).))))))))))))).).))))).))))))))). ( -48.00) >DroSim_CAF1 2788 96 - 1 GUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGUC---GUGGCCAGGAGUCCCAGCAGCGCGGUGCAAUGGACCA ..((((.(..(..((((.((((.....)))).(((((...((((..(.((......)).---)..))))((....)).)))))))))..)...).)))) ( -41.90) >DroEre_CAF1 2819 96 - 1 GUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC---GUGGCCAGGAGUCACAGCAGCGGAGUGCAGUGGACCA ..((((.((((((..((......))..)))))(((((((.(.(((((((..(((...((---.......)).))).))))))).).)))))))).)))) ( -39.60) >DroYak_CAF1 2850 96 - 1 GUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC---GUGGCCAGGAGUCACAGCAGCGAAGUGCAGUGGACCA ....(..((..((((((.....((((.((((.((.(((......))).)).))))))))---(((((.....))))).)))))).))..)......... ( -39.90) >consensus GUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC___GUGGCCAGGAGUCACAGCAGCGAAGUGCAAUGGACCA ...(((.(..(((((...((((.....)))).(((((...(((((((.((......))....)))))))(......).)))))..)))))...).))). (-22.50 = -23.23 + 0.73)

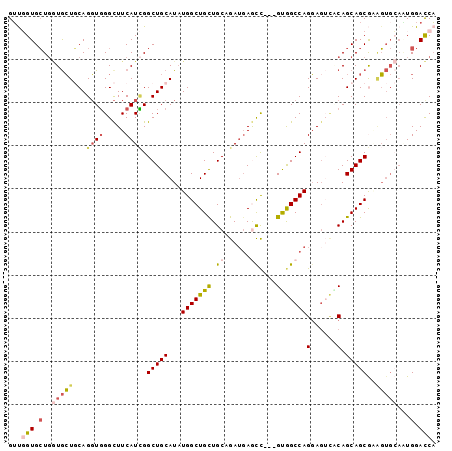
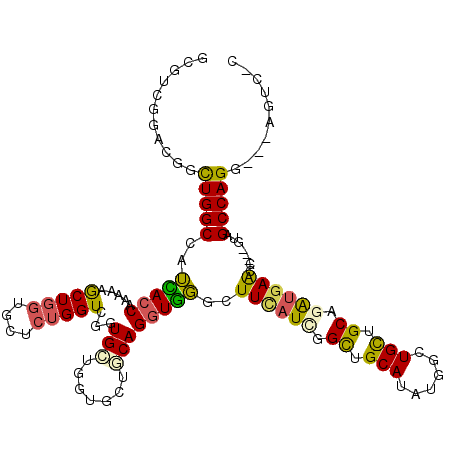
| Location | 13,182,289 – 13,182,403 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.27 |
| Mean single sequence MFE | -48.03 |
| Consensus MFE | -23.31 |
| Energy contribution | -22.45 |
| Covariance contribution | -0.86 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707861 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13182289 114 - 22407834 GCGUUGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC---GUGGCCAGG---AGUCGC ......(((..((((((((((((.((..((..(..((......))..)..)).))...))))((((.((((.((.(((......))).)).))))))))---)))))))).---.))).. ( -51.60) >DroVir_CAF1 2874 117 - 1 GCGGCGGCCGUCUGGCGAGUACCAAAAAGCUCGUCCUGUGGUCUGUGGUGCUGGUUCAGCUGUGUUUCAUUGGUUGCAUUUGGCUGCUGCAGGCUAAUC---AUAGCCAGCUGCAGCUGC (((((.((.(.(((((((((........))))......(((((((..((((..(..((((((........))))))...)..)).))..)))))))...---...)))))).)).))))) ( -48.00) >DroPse_CAF1 3145 114 - 1 GCAUCGGGCGUUUGGCCAUAACCAAAAAACUGGUGCUCUGGUCCGUGCUGAUACUUCAGGUUGGCUUCAUAGGCUGCAUAUGGCUGUUGCAGAUGAACUACAGCAGCCAGG---AU---C (((((((...(((((......)))))...)))))))...((((((((((((....)))....((((.....)))))))).(((((((((.((.....)).)))))))))))---))---) ( -44.10) >DroSec_CAF1 2820 114 - 1 GGGUUGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGAUUUAUCGGCUGCAUAUGGCUGCUGCAGAUGAGUC---GUGGCCAGG---AGUCAC ......(((..((((((((((((.((..((..(..((......))..)..)).))...)))).((((((((.((.(((......))).)).))))))))---)))))))).---.))).. ( -48.80) >DroEre_CAF1 2841 114 - 1 GCCUGGGACGGCUGGCCAUCACCAAAAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAGCC---GUGGCCAGG---AGUCAC ......(((..((((((((((((.....((..(..((......))..)..))......))))((((.((((.((.(((......))).)).))))))))---)))))))).---.))).. ( -51.60) >DroPer_CAF1 3133 114 - 1 GCAUCGGGCGUUUGGCCAUAACCAAAAAACUGGUGCUCUGGUCCGUGCUGAUUCUUCAGGUUGGCUUCAUAGGCUGCAUAUGGCUGUUGCAGAUGAACUACAGCAGCCAGG---AU---C (((((((...(((((......)))))...)))))))...((((((((((((....)))....((((.....)))))))).(((((((((.((.....)).)))))))))))---))---) ( -44.10) >consensus GCGUCGGACGGCUGGCCAUCACCAAAAAGCUGGUGCUCUGGUCGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUAUGGCUGCUGCAGAUGAACC___GUAGCCAGG___AGUC_C ...........(((((..(((((.....(((((....)))))...(((.......))))))))..((((((.((.(((......))).)).))))))........))))).......... (-23.31 = -22.45 + -0.86)


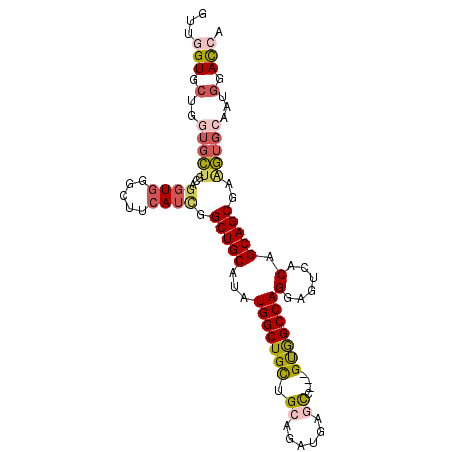
| Location | 13,182,323 – 13,182,429 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 84.91 |
| Mean single sequence MFE | -46.23 |
| Consensus MFE | -34.63 |
| Energy contribution | -36.80 |
| Covariance contribution | 2.17 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.599169 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
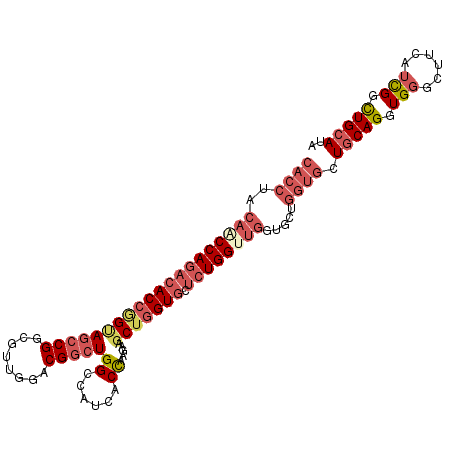
>2L_DroMel_CAF1 13182323 106 - 22407834 CACCUACAACCAGACACCGGUAGCCGGCGUUGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUA ((((..(((((((((((((((.((((((........))))))..((.....)).))))))).)))))))).....)))).(((((.(((......))).))))).. ( -47.90) >DroVir_CAF1 2911 99 - 1 -------AGCCAGACACUUGCAACCGGCGGCGGCCGUCUGGCGAGUACCAAAAAGCUCGUCCUGUGGUCUGUGGUGCUGGUUCAGCUGUGUUUCAUUGGUUGCAUU -------((((((((((..(((((((((((((((((.(.(((((((........)))))))..)))).))))..))))))))..)).)))))....)))))..... ( -37.70) >DroSec_CAF1 2854 106 - 1 CACCUACAACCAGACACCGGUAGCCGGGGUUGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGAUUUAUCGGCUGCAUA ((((..((((((((((((((((((((........)))))((......)).....))))))).)))))))).....)))).(((((.(((......))).))))).. ( -47.50) >DroSim_CAF1 2844 106 - 1 CACCUACAACCAGACACCGGUAGCCGGCGUUGGACGGCUGGCCAUCACUAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUA ((((..(((((((((((((((.((((((........))))))..((.....)).))))))).)))))))).....)))).(((((.(((......))).))))).. ( -47.90) >DroEre_CAF1 2875 106 - 1 CACCCACACCCAGACACCAGCAGCCGGCCUGGGACGGCUGGCCAUCACCAAAAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUA ..(((((...(((.((((((((.((((((.(((..((....))..(((((......)))))))).)))))))))))))))))...)))))................ ( -48.70) >DroYak_CAF1 2906 106 - 1 CACCCACACCCAGCCACCAGGAGCCGGCCUGGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUA ..(((((...((((.(((((..(((((((......)))))))((((((((.((.((....))))))).))))))))))))))...)))))................ ( -47.70) >consensus CACCUACAACCAGACACCGGUAGCCGGCGUUGGACGGCUGGCCAUCACCAAGAAGCUGGUGCUCUGGUUGGUGCUGGUGCUGCAGGUGGGCUUCAUCGGCUGCAUA ((((..((((((((((((((((((((........)))))((......)).....))))))).)))))))).....)))).(((((.(((......))).))))).. (-34.63 = -36.80 + 2.17)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:17 2006