
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,144,216 – 13,144,367 |
| Length | 151 |
| Max. P | 0.927490 |

| Location | 13,144,216 – 13,144,330 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.49 |
| Mean single sequence MFE | -37.04 |
| Consensus MFE | -30.42 |
| Energy contribution | -31.45 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.699025 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
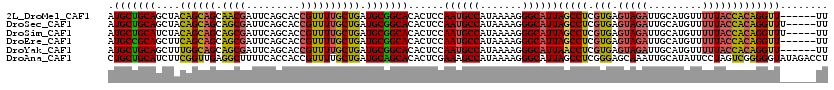
>2L_DroMel_CAF1 13144216 114 + 22407834 AA------AACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGUUGCUGCUGUAGCUGCAGCAU ..------....(((((((...((..........((....)).((((((((.......)))))))).)).)))))))((((((......))))))...(((((.((....)).))))).. ( -37.50) >DroSec_CAF1 11306 115 + 1 AA-----AAACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGUAGCUGCAGCAU ..-----.....(((((((...((..........((....)).((((((((.......)))))))).)).)))))))((((((......))))))...(((((.((....)).))))).. ( -39.90) >DroSim_CAF1 12105 115 + 1 AA-----AAACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGUAGAUGCAGCAU ..-----.....(((((((...((..........((....)).((((((((.......)))))))).)).)))))))((((((......)))))).....((((((.......)))))). ( -37.20) >DroEre_CAF1 13208 114 + 1 AA------AACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGAAGCUGCGGCAU ..------..(((((((((.............))).))).)))((((((((.......))))))))...((((((((((((((...(((((......))))).))))))...)))))))) ( -40.52) >DroYak_CAF1 11454 114 + 1 AA------AACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGUUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCCAAAGCUGCAGCAU ..------....(((((((...((..........((....)).((((((((.......)))))))).)).)))))))((((((......))))))...(((((.((....)).))))).. ( -36.80) >DroAna_CAF1 10604 120 + 1 AGGUCUAUACCCCCGACUAGGAAUAUGCAAUUUGCUCCCGAGGCUAAUGCCCUUUUAUGGCUUUCGAGUGUGCUGCAUCAGCAAAACGGUGGUGAAAAGCCUCAACCGAAGAUGCAGCAG ..((.(((.((........)).))).)).....((((..(((((((...........))))))).)))).(((((((((.......(((((((.....)))...))))..))))))))). ( -30.30) >consensus AA______AACCUGUGGUAAAAACAUGCAAUCUACUCACGAGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGAAGCUGCAGCAU ............(((((((...((..........((....)).((((((((.......)))))))).)).)))))))((((((......))))))...(((((.((....)).))))).. (-30.42 = -31.45 + 1.03)


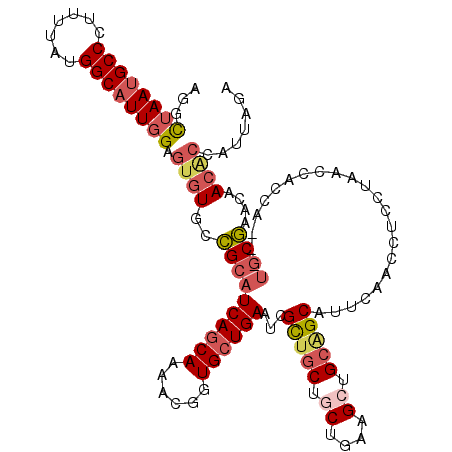
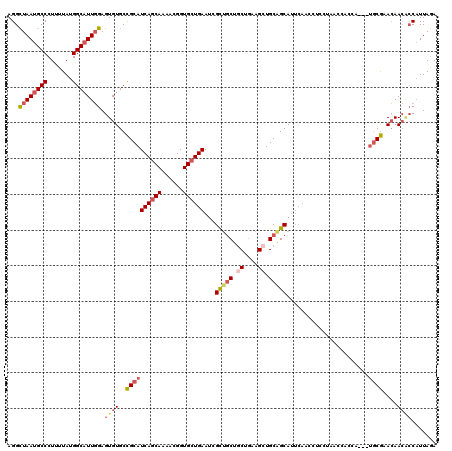
| Location | 13,144,216 – 13,144,330 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.49 |
| Mean single sequence MFE | -37.78 |
| Consensus MFE | -32.43 |
| Energy contribution | -32.22 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.927490 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
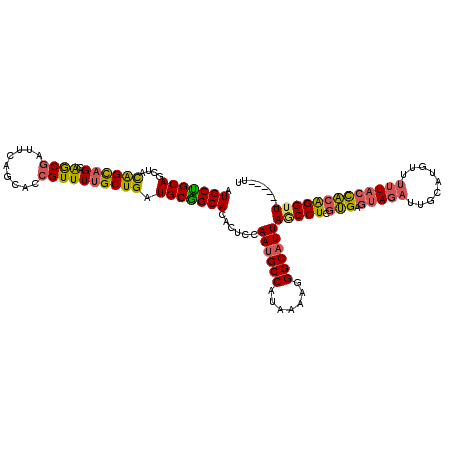
>2L_DroMel_CAF1 13144216 114 - 22407834 AUGCUGCAGCUACAGCAGCAACGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUU------UU .(((((((....((((((.((((.........)))))))))).)))))))......((((((.......))))))(((((.(((.(((((.........)))))))))))))------.. ( -37.60) >DroSec_CAF1 11306 115 - 1 AUGCUGCAGCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUUU-----UU .(((((((....((((((.((((.........)))))))))).)))))))......((((((.......))))))(((((.(((.(((((.........))))))))))))).-----.. ( -37.60) >DroSim_CAF1 12105 115 - 1 AUGCUGCAUCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUUU-----UU .(((((((((...(((((.((((.........))))))))))))))))))......((((((.......))))))(((((.(((.(((((.........))))))))))))).-----.. ( -38.20) >DroEre_CAF1 13208 114 - 1 AUGCCGCAGCUUCAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUU------UU .(((((((...(((((((.((((.........))))))))))))))))))......((((((.......))))))(((((.(((.(((((.........)))))))))))))------.. ( -40.90) >DroYak_CAF1 11454 114 - 1 AUGCUGCAGCUUUGGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAACCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUU------UU .(((((((((....))..((((((..(......)..)))))).)))))))......((((((.......))))))(((((.(((.(((((.........)))))))))))))------.. ( -37.30) >DroAna_CAF1 10604 120 - 1 CUGCUGCAUCUUCGGUUGAGGCUUUUCACCACCGUUUUGCUGAUGCAGCACACUCGAAAGCCAUAAAAGGGCAUUAGCCUCGGGAGCAAAUUGCAUAUUCCUAGUCGGGGGUAUAGACCU .(((((((((..((((((((....))))..)))).......)))))))))..(((((...........((((....)))).((((((.....))....))))..)))))(((....))). ( -35.10) >consensus AUGCUGCAGCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCUCGUGAGUAGAUUGCAUGUUUUUACCACAGGUU______UU .(((((((....((((((.((((.........)))))))))).)))))))......((((((.......))))))(((((.(((.(((((.........)))))))))))))........ (-32.43 = -32.22 + -0.22)
| Location | 13,144,250 – 13,144,367 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.86 |
| Mean single sequence MFE | -36.15 |
| Consensus MFE | -28.54 |
| Energy contribution | -29.77 |
| Covariance contribution | 1.23 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.787307 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13144250 117 + 22407834 AGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGUUGCUGCUGUAGCUGCAGCAUUCAACCAUCUAACCACCA---UGCGAACAACACCAUUAGA ...((((((((.......)))))))).((((..((((((((((......))))))...((((.((((((....))))))..)))).............---))))....))))....... ( -36.70) >DroSec_CAF1 11341 117 + 1 AGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGUAGCUGCAGCAUUCAACCUCCUAACCACCA---UGCGAACAACACCAUUAGA ...((((((((.......)))))))).((((..((((((((((......))))))...(((((.((....)).)))))....................---))))....))))....... ( -38.40) >DroSim_CAF1 12140 117 + 1 AGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGUAGAUGCAGCAUUCAACCAUCUAACCACCA---UGCGAACAACACCAUUAGA ...((((((((.......)))))))).((((..((((((((((......)))))).....((((((.......))))))...................---))))....))))....... ( -35.70) >DroEre_CAF1 13242 117 + 1 AGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGAAGCUGCGGCAUUCAACCUGCUAACCACCA---UGCGAACAACACCAUUAGA (((((((((((.......)))))))).(.((((((((((((((...(((((......))))).))))))...)))))))).)..)))((.........---.))................ ( -38.50) >DroYak_CAF1 11488 120 + 1 AGGUUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCCAAAGCUGCAGCAUUCAACCUCCUAACCACCAACCUGCGAACAACGCCAUUAGA (((((((((((.......))))))((((.((...(((((........)))))(((((.(((((.((....)).))))))))))))))))........)))))(((.....)))....... ( -39.80) >DroAna_CAF1 10644 94 + 1 AGGCUAAUGCCCUUUUAUGGCUUUCGAGUGUGCUGCAUCAGCAAAACGGUGGUGAAAAGCCUCAACCGAAGAUGCAGCAG-----------------------CAAAGAA---CCUUAAA .(((....)))..((((.((..(((...(((((((((((.......(((((((.....)))...))))..))))))))).-----------------------))..)))---)).)))) ( -27.80) >consensus AGGCUAAUGCCCUUUUAUGGCAUUGGAGUGUGCCGCAUCAGCAAAACGGUGCUGAAUCGCUGCUGCUGAAGCUGCAGCAUUCAACCUCCUAACCACCA___UGCGAACAACACCAUUAGA ...((((((((.......)))))))).((((..((((((((((......))))))...(((((.((....)).))))).......................))))....))))....... (-28.54 = -29.77 + 1.23)

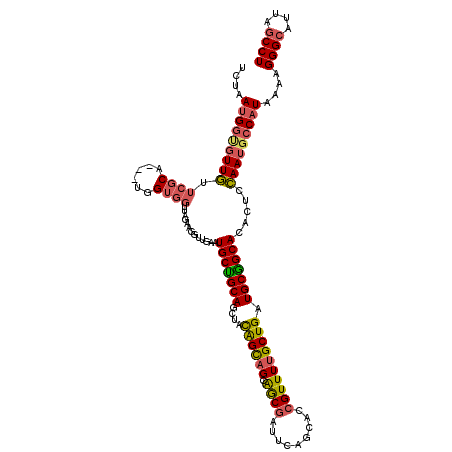
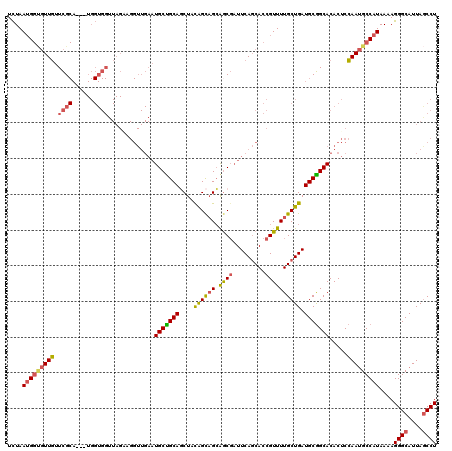
| Location | 13,144,250 – 13,144,367 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.86 |
| Mean single sequence MFE | -40.58 |
| Consensus MFE | -31.00 |
| Energy contribution | -31.27 |
| Covariance contribution | 0.27 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.739390 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 13144250 117 - 22407834 UCUAAUGGUGUUGUUCGCA---UGGUGGUUAGAUGGUUGAAUGCUGCAGCUACAGCAGCAACGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCU ......(((..(((((...---..(((((....(((.....(((((((....((((((.((((.........)))))))))).)))))))....)))..)))))....)))))...))). ( -38.40) >DroSec_CAF1 11341 117 - 1 UCUAAUGGUGUUGUUCGCA---UGGUGGUUAGGAGGUUGAAUGCUGCAGCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCU ......(((..(((((...---..(((((..((((((((((((((((.((....)).))))).)))))))...((.((((....)))).)).))))...)))))....)))))...))). ( -45.50) >DroSim_CAF1 12140 117 - 1 UCUAAUGGUGUUGUUCGCA---UGGUGGUUAGAUGGUUGAAUGCUGCAUCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCU ...((((((((((.((((.---((.((.(((((((((.....))..)))))).).)).))))))..))))))))))..((((((((((((........))))........)))))))).. ( -41.80) >DroEre_CAF1 13242 117 - 1 UCUAAUGGUGUUGUUCGCA---UGGUGGUUAGCAGGUUGAAUGCCGCAGCUUCAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCU ...((((((((((.((((.---((.((.(.(((.(((.....)))...)))..).)).))))))..))))))))))..((((((((((((........))))........)))))))).. ( -42.50) >DroYak_CAF1 11488 120 - 1 UCUAAUGGCGUUGUUCGCAGGUUGGUGGUUAGGAGGUUGAAUGCUGCAGCUUUGGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAACCU .(((((.(((.....)))..))))).((((.((((((((((((((((.((....)).))))).)))))))...((.((((....)))).)).))))((((((.......)))))))))). ( -46.60) >DroAna_CAF1 10644 94 - 1 UUUAAGG---UUCUUUG-----------------------CUGCUGCAUCUUCGGUUGAGGCUUUUCACCACCGUUUUGCUGAUGCAGCACACUCGAAAGCCAUAAAAGGGCAUUAGCCU ((((.((---((.((((-----------------------.(((((((((..((((((((....))))..)))).......)))))))))....)))))))).)))).((((....)))) ( -28.70) >consensus UCUAAUGGUGUUGUUCGCA___UGGUGGUUAGAAGGUUGAAUGCUGCAGCUACAGCAGCAGCGAUUCAGCACCGUUUUGCUGAUGCGGCACACUCCAAUGCCAUAAAAGGGCAUUAGCCU ....(((((((((.((((......)))).............(((((((....((((((.((((.........)))))))))).))))))).....)))))))))....((((....)))) (-31.00 = -31.27 + 0.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:58 2006