
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 13,141,703 – 13,141,861 |
| Length | 158 |
| Max. P | 0.976018 |

| Location | 13,141,703 – 13,141,822 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 92.61 |
| Mean single sequence MFE | -24.52 |
| Consensus MFE | -20.46 |
| Energy contribution | -21.38 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.976018 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
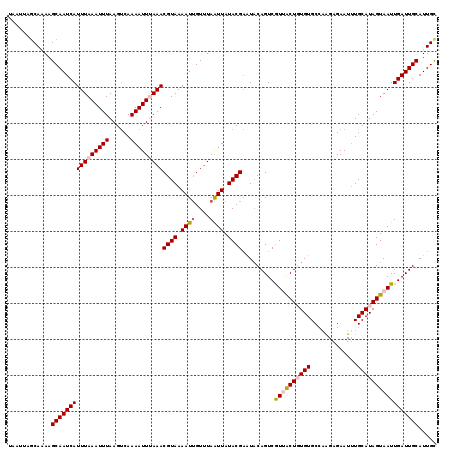
>2L_DroMel_CAF1 13141703 119 + 22407834 UAAUUAGCAAAAGCAAUCAUUUAAAUUUAAGUCAAAAUUUAAACGUAAAAUUGUUUAAUUAUACGAAUUCAGUCAUUGCUGUGUGCGAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGU ......((((..((((((((((((((((......)))))))))((((.((((....)))).)))).........((((((..(((((((.....)))))))))))))))))))).)))) ( -25.60) >DroSec_CAF1 8780 119 + 1 UAAUAAUCAAAAGCAAUCAUUUAAAUUUAAGUCAAAAUUUAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUGUGCAUAGUAAUUGAUUGCAUUGC ............((((((((((((((((......)))))))))((((.((((....)))).)))).........(((((((((..(.........)..))))))))))))))))..... ( -27.30) >DroSim_CAF1 9585 119 + 1 UAAUUAGCAAAAGCAAUCAUUUAAAUUUAAGUCAAAAUUUAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGC ......((((..((((((((((((((((......)))))))))((((.((((....)))).)))).........((((((((((.............))))))))))))))))).)))) ( -25.92) >DroEre_CAF1 10890 119 + 1 UAAUUAGCACAAGCAAUCAUUUAAAUUUAAGCCAAAAUUAAAACGUAAAAUGGUUUAGUUAUACGUGUACAGUCGUAACUUUGUGCCAGGAGAAUUUGCAUAGUAAUUGAUUGCAUUGC ......(((...(((((((.(((..(((((.......)))))..(((((.((((..((((((((.......)).))))))....))))......)))))....))).)))))))..))) ( -22.20) >DroYak_CAF1 8888 119 + 1 UAACUAGCACAAGCAAUCAUUUAAAUUUAAGUCAAAAUUAAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUAACUUUGUGCCCAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGC ......(((...(((((((.(((..(((((.......)))))..(((((............(((((......))))).(((((....)))))..)))))....))).)))))))..))) ( -21.60) >consensus UAAUUAGCAAAAGCAAUCAUUUAAAUUUAAGUCAAAAUUUAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGC ............((((((((((((((((......)))))))))((((.((((....)))).)))).........((((((((((.............)))))))))))))))))..... (-20.46 = -21.38 + 0.92)


| Location | 13,141,742 – 13,141,861 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 91.76 |
| Mean single sequence MFE | -27.48 |
| Consensus MFE | -21.58 |
| Energy contribution | -21.98 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.834548 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
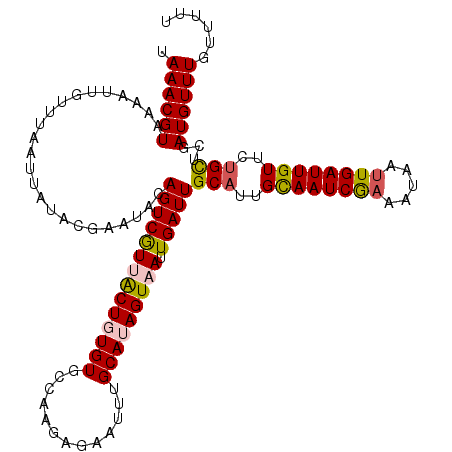
>2L_DroMel_CAF1 13141742 119 + 22407834 UAAACGUAAAAUUGUUUAAUUAUACGAAUUCAGUCAUUGCUGUGUGCGAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGUAAUCGAAAUAAUUGAUUGUUCCGCUCGAUGUUUGUUUUU (((((((..((((((((.....(((((...(((((((((((..(((((((.....)))))))))))).))))))..)))))....))))))))((....))......)))))))..... ( -24.80) >DroSec_CAF1 8819 119 + 1 UAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUGUGCAUAGUAAUUGAUUGCAUUGCAAUCGAAAUAAUUGAUUGUUCUGCUCGAUGUUUGUUUUU ((((((......)))))).....(((((((((((((((((((((..(.........)..)))))))).)))))(((..((((((((.....))))))))..)))..).))))))).... ( -32.20) >DroSim_CAF1 9624 119 + 1 UAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGCAAUCGAAAUAAUUGAUUGUUCUGCUCGAUGUUUGUUUUU ((((((......)))))).....((((((((((((((((((((((.............))))))))).)))))(((..((((((((.....))))))))..)))..).))))))).... ( -29.32) >DroEre_CAF1 10929 119 + 1 AAAACGUAAAAUGGUUUAGUUAUACGUGUACAGUCGUAACUUUGUGCCAGGAGAAUUUGCAUAGUAAUUGAUUGCAUUGCAAUCGCAGUAAUUGAUUGUUCUGCUCGAUGUUUGUUUUU ...(((((.(((......))).)))))((((((........))))))...((((((..((((.......((((((...))))))((((.(((.....)))))))...))))..)))))) ( -26.40) >DroYak_CAF1 8927 119 + 1 AAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUAACUUUGUGCCCAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGCAAUCAAAAUAGUUGAUUGUUUUGUUUGAUGUUUGUUUUU .(((((......))))).((((.(((((.(((((((((((((((....)))))...)))).......((((((((...)))))))).......))))))))))).)))).......... ( -24.70) >consensus UAAACGUAAAAUUGUUUAAUUAUACGAAUACAGUCGUUACUGUGUGCCAAGAGAAUUUGCAUAGUAAUUGAUUGCAUUGCAAUCGAAAUAAUUGAUUGUUCUGCUCGAUGUUUGUUUUU .((((((........................((((((((((((((.............))))))))).)))))(((..((((((((.....))))))))..)))...))))))...... (-21.58 = -21.98 + 0.40)

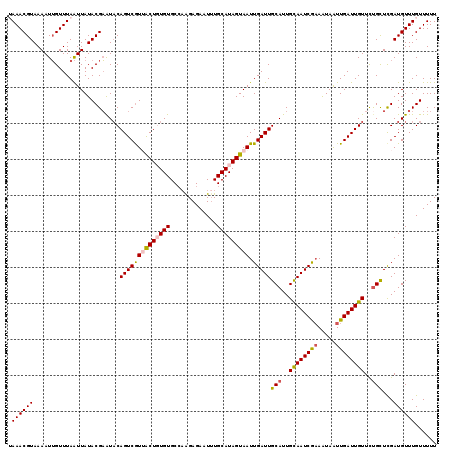
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:51 2006