
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,773,020 – 12,773,167 |
| Length | 147 |
| Max. P | 0.936136 |

| Location | 12,773,020 – 12,773,135 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 76.05 |
| Mean single sequence MFE | -34.17 |
| Consensus MFE | -18.83 |
| Energy contribution | -19.70 |
| Covariance contribution | 0.87 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.25 |
| SVM RNA-class probability | 0.936136 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
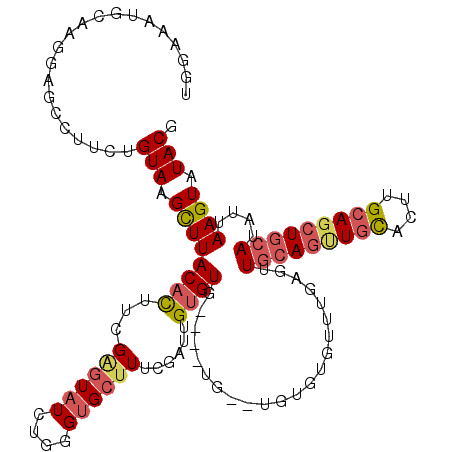
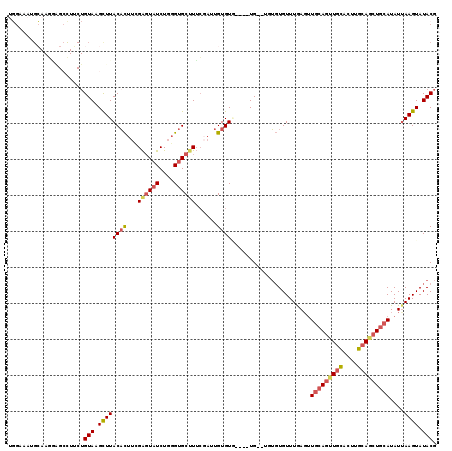
>2L_DroMel_CAF1 12773020 115 + 22407834 UGGAAAAGCAAGGAGCCUGCUGUAAGCUUACACUUCGAGUAUCUGGGUGCUUUCGAUUGUGUG----UGUGUGUGUGUUUAAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACG ......((((.(....)))))....((.(((((.((((((((....))))..))))..)))))----.))..((((((((((..(((((((((....)))))))))..)))))))))). ( -38.00) >DroSec_CAF1 50012 93 + 1 U--------------------GUAAGCUUACACUUCGAGUAUCUGGGUGCUUUCGAUUGUGUG----UG--UGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACG .--------------------....((.(((((.((((((((....))))..))))..)))))----.)--)((((((((((..(((((((((....)))))))))..)))))))))). ( -34.40) >DroSim_CAF1 50533 115 + 1 UGGAAAAGCAAGGAGCCUGCUGUAAGCUUACACUUCGAGUAUCUGGGUGCUUUCGAUUGUGUG----UGUGUGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACG ......((((.(....)))))....((.(((((.((((((((....))))..))))..)))))----.))..((((((((((..(((((((((....)))))))))..)))))))))). ( -38.10) >DroEre_CAF1 52069 111 + 1 UGGAAAUGCAAGUAGGUUUCUGUAAGCUUACACUUCGGGUAUCUGGGUGCUUGCCAUUGUGUU----UG----UGUGUUUGAGUUGCAGCUGCACUUGCAGCUGCAUAUUAAGUAUACG ...((((((((((((((((....))))))))....(((((((....)))))))...)))))))----)(----(((((((((..(((((((((....)))))))))..)))))))))). ( -41.50) >DroYak_CAF1 52416 110 + 1 UGGAAAUGCAAGGCGCCUUCUGUAAGCUUACACUUGGAGUAUCUGGGUGCUUUCGAUUGUGUA----UG----UGUGUUUGAC-UCCAGUUGCACUGGCAGCUGCAUAUUAAGUAUACG ......((.(((((((((.((.((((......)))).)).....)))))))))))...(((((----((----(((((..(.(-(((((.....)))).))).))))))...)))))). ( -31.10) >DroAna_CAF1 49989 117 + 1 CCGAACUGGAACAGGCCUUCUGUAUGUUUACCUCCAGACUAUCUCUGAGCCUUG--CUGUGUUCUGCUGUGGUGCUGUUUAAGUUGCAUUUUUGGUAGCACUUUCAUAUCAAGUAUACG ...((((.(((((((((....(((.(..(((...((((.....))))(((...)--)))))..))))...))).)))))).)))).....(((((((.........)))))))...... ( -21.90) >consensus UGGAAAUGCAAGGAGCCUUCUGUAAGCUUACACUUCGAGUAUCUGGGUGCUUUCGAUUGUGUG____UG__UGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACG .....................(((.((((((((...((((((....))))))......))))......................(((((((((....)))))))))....)))).))). (-18.83 = -19.70 + 0.87)

| Location | 12,773,059 – 12,773,167 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 81.80 |
| Mean single sequence MFE | -39.26 |
| Consensus MFE | -24.38 |
| Energy contribution | -25.97 |
| Covariance contribution | 1.59 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.27 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925126 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
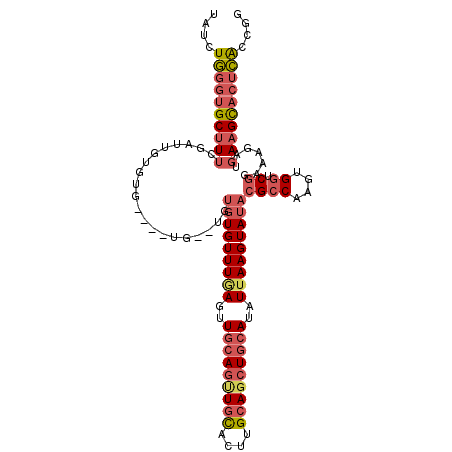
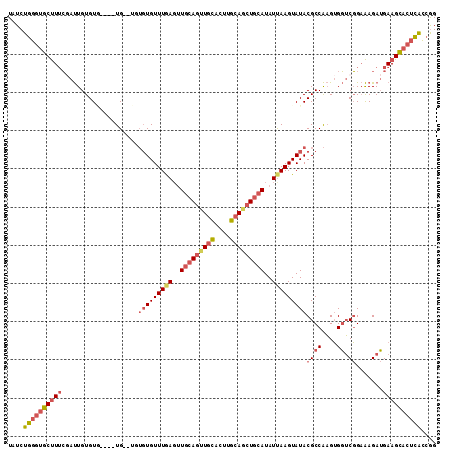
>2L_DroMel_CAF1 12773059 108 + 22407834 UAUCUGGGUGCUUUCGAUUGUGUG----UGUGUGUGUGUUUAAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....((((((((((((((..(...----((.((((((((((((..(((((((((....)))))))))..)))))))))))))).)..)))))).......)))))))).... ( -45.41) >DroSec_CAF1 50031 106 + 1 UAUCUGGGUGCUUUCGAUUGUGUG----UG--UGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....((((((((((((((..(.((----.(--(((((((((((..(((((((((....)))))))))..)))))))))))))).)..)))))).......)))))))).... ( -45.71) >DroSim_CAF1 50572 108 + 1 UAUCUGGGUGCUUUCGAUUGUGUG----UGUGUGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....((((((((((((((..(...----((.((((((((((((..(((((((((....)))))))))..)))))))))))))).)..)))))).......)))))))).... ( -45.51) >DroEre_CAF1 52108 104 + 1 UAUCUGGGUGCUUGCCAUUGUGUU----UG----UGUGUUUGAGUUGCAGCUGCACUUGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....(((((((((((((((.....----((----(((((((((..(((((((((....)))))))))..)))))))))))...))))))..........))))))))).... ( -41.10) >DroYak_CAF1 52455 103 + 1 UAUCUGGGUGCUUUCGAUUGUGUA----UG----UGUGUUUGAC-UCCAGUUGCACUGGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....((((((((((((((..(...----((----(((((((((.-..(((((((....)))))))....)))))))))))....)..)))))).......)))))))).... ( -33.21) >DroAna_CAF1 50028 110 + 1 UAUCUCUGAGCCUUG--CUGUGUUCUGCUGUGGUGCUGUUUAAGUUGCAUUUUUGGUAGCACUUUCAUAUCAAGUAUACGCCGUGGGUACCGUGAGAAGAAGUCAUGGAAAG ...........((..--(.((((..((((.(((((.((.....(((((.......))))).....))))))))))).)))).)..))..((((((.......)))))).... ( -24.60) >consensus UAUCUGGGUGCUUUCGAUUGUGUG____UG__UGUGUGUUUGAGUUGCAGUUGCACUUGCAGCUGCAUAUUAAGUAUACGCCAAGUGGUCGGAAAGAUGAAGCACUCACCGG ....((((((((((....................(((((((((..(((((((((....)))))))))..)))))))))((((....)).)).......)))))))))).... (-24.38 = -25.97 + 1.59)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:17:57 2006