
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,625,992 – 12,626,242 |
| Length | 250 |
| Max. P | 0.922867 |

| Location | 12,625,992 – 12,626,112 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -53.13 |
| Consensus MFE | -35.39 |
| Energy contribution | -35.53 |
| Covariance contribution | 0.15 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.785204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
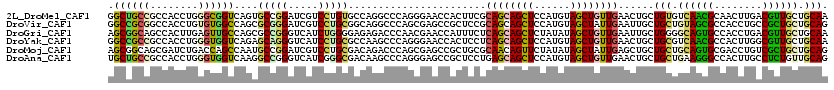
>2L_DroMel_CAF1 12625992 120 + 22407834 AUCACUGUCGCCACAGGAUUUUGCCCAGUUCCAUCAGCUAUUGCAGCAACGUCAAGUUGCGUUGACACAGCAGUUCAACAGCUACAUGGAGCUGCUGCGAAGUGGUUCCCUGGGCCUGGC ......((((((.(((((((((((.(((((((((.((((......(((((.....)))))(((((.((....)))))))))))..)))))))))..))))))).....)))))))..))) ( -45.10) >DroVir_CAF1 64117 120 + 1 CUCCAUUUCGCCGCAGGACUUCGCCCAGUUCCAGCAGCUGCUGCAGCAGCGGCAGGUGGCGCUACAGCAGCAAUUCAAUAGCUACAUGGAGCUGCUGCGGAGCGGCUCGCUGGGCCUGCC ............(((((....)(((((((...(((.(((.((((((((((..((.(((((((....))(....)......))))).))..))))))))))))).))).))))))))))). ( -54.30) >DroPse_CAF1 6984 120 + 1 CUCGCUGUCCCCCCAGGACUUUGCCCAGUUCCAGCAGCUGCUGCAGCAGCGGCAGGUGGCCCUCCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUGCGCAGCGGAUCGCUGGGCCUGGC ...((.((((.....))))...))((((..(((((..(((((((.((((((((.((....))((((..(((.(.....).)))...)))))))))))))))))))...)))))..)))). ( -60.60) >DroMoj_CAF1 60519 120 + 1 CUCCAUUUCGCAACAGGAUUUCUCUCAGCUCCAGCAGGUGCUGCAGCAGCGACAGGUCGCACUGCAGCAGCAGCUCAAUAGCUAUAUAGAACUGUUGCGCAGCGGCUCGCUGGGUCUGUC .(((...........))).......(((..(((((((.((((((.(((((((....))))..))).(((((((.((.(((....))).)).))))))))))))).)).)))))..))).. ( -49.30) >DroAna_CAF1 1675 120 + 1 AUCCCUGUCCCCGCAGGACUUUGCCCAGUUCCAGCAGCUACUGCAACAGAGGCAAGUGGCCCUUCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUCAGGAGCGGCUCCCUGGGCUUGUC ...(((((....)))))((...((((((....(((((((.((((...((.(((.....)))))...)))).)))).....)))....(((((((((....)))))))))))))))..)). ( -48.90) >DroPer_CAF1 7229 120 + 1 CUCGCUGUCCCCCCAGGACUUUGCCCAGUUCCAGCAGCUGCUGCAGCAGCGGCAGGUGGCCCUCCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUGCGCAGCGGAUCGCUGGGCCUGGC ...((.((((.....))))...))((((..(((((..(((((((.((((((((.((....))((((..(((.(.....).)))...)))))))))))))))))))...)))))..)))). ( -60.60) >consensus CUCCCUGUCCCCACAGGACUUUGCCCAGUUCCAGCAGCUGCUGCAGCAGCGGCAGGUGGCCCUCCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUGCGCAGCGGCUCGCUGGGCCUGGC ............(((((......((((((.((....(((((((...))))))).....((......((((((((((..((......))))))))))))...))))...))))))))))). (-35.39 = -35.53 + 0.15)

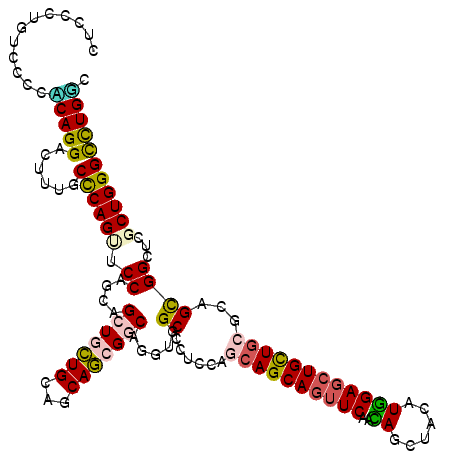
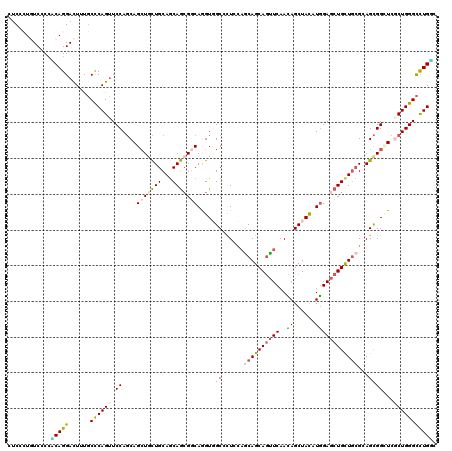
| Location | 12,625,992 – 12,626,112 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -55.80 |
| Consensus MFE | -33.15 |
| Energy contribution | -33.85 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.72 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.660094 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12625992 120 - 22407834 GCCAGGCCCAGGGAACCACUUCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGUGUCAACGCAACUUGACGUUGCUGCAAUAGCUGAUGGAACUGGGCAAAAUCCUGUGGCGACAGUGAU .....((((((((((....)))((..(((.(((((.(((((((((....((..((((((......))))))..))..)))))))))))))).))).)).....)))).)))......... ( -45.30) >DroVir_CAF1 64117 120 - 1 GGCAGGCCCAGCGAGCCGCUCCGCAGCAGCUCCAUGUAGCUAUUGAAUUGCUGCUGUAGCGCCACCUGCCGCUGCUGCAGCAGCUGCUGGAACUGGGCGAAGUCCUGCGGCGAAAUGGAG .......(((....(((((((((((((((((......)))).......((((((.((((((.(....).)))))).))))))))))).)))...((((...)))).)))))....))).. ( -54.00) >DroPse_CAF1 6984 120 - 1 GCCAGGCCCAGCGAUCCGCUGCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGCUGGAGGGCCACCUGCCGCUGCUGCAGCAGCUGCUGGAACUGGGCAAAGUCCUGGGGGGACAGCGAG .((((..((((((....(((((((((((((((((.(((((.(.....).))))))))).(((.....))))))))))).)))))))))))..))))((...((((.....)))).))... ( -64.60) >DroMoj_CAF1 60519 120 - 1 GACAGACCCAGCGAGCCGCUGCGCAACAGUUCUAUAUAGCUAUUGAGCUGCUGCUGCAGUGCGACCUGUCGCUGCUGCAGCACCUGCUGGAGCUGAGAGAAAUCCUGUUGCGAAAUGGAG ......(((((((...)))))((((((((((((...(((((....(((.(.(((((((((((((....)))).))))))))).).)))..)))))..))))...))))))))....)).. ( -52.50) >DroAna_CAF1 1675 120 - 1 GACAAGCCCAGGGAGCCGCUCCUGAGCAGCUCCAUGUAGCUGUUGAACUGCUGCUGAAGGGCCACUUGCCUCUGUUGCAGUAGCUGCUGGAACUGGGCAAAGUCCUGCGGGGACAGGGAU .....(((((((((((.(((....))).)))))..((((((.....(((((.((....((((.....))))..)).))))))))))).....))))))...(((((..(....).))))) ( -53.80) >DroPer_CAF1 7229 120 - 1 GCCAGGCCCAGCGAUCCGCUGCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGCUGGAGGGCCACCUGCCGCUGCUGCAGCAGCUGCUGGAACUGGGCAAAGUCCUGGGGGGACAGCGAG .((((..((((((....(((((((((((((((((.(((((.(.....).))))))))).(((.....))))))))))).)))))))))))..))))((...((((.....)))).))... ( -64.60) >consensus GCCAGGCCCAGCGAGCCGCUGCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGCUGCAGGGCCACCUGCCGCUGCUGCAGCAGCUGCUGGAACUGGGCAAAGUCCUGCGGCGACAGGGAG .((((((((((....((((....((((((((......))))))))..(((((((.(.((((........)))).).)))))))..)).))..))))))......))))............ (-33.15 = -33.85 + 0.70)

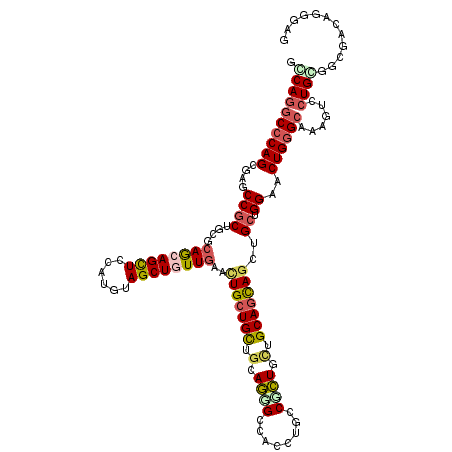
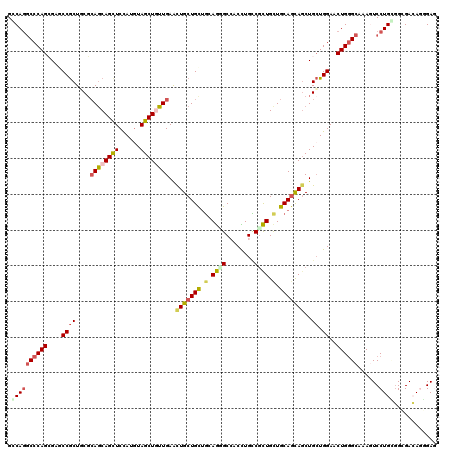
| Location | 12,626,032 – 12,626,152 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.72 |
| Mean single sequence MFE | -53.40 |
| Consensus MFE | -27.29 |
| Energy contribution | -26.10 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.60 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.814870 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
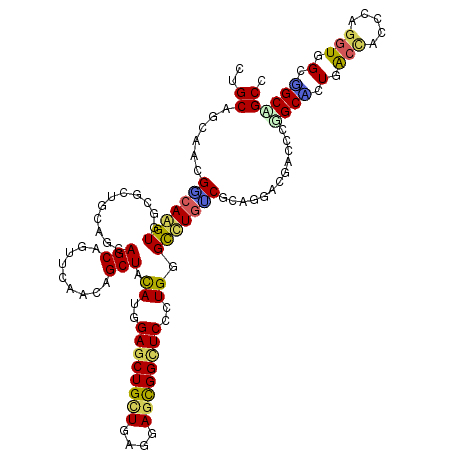
>2L_DroMel_CAF1 12626032 120 + 22407834 UUGCAGCAACGUCAAGUUGCGUUGACACAGCAGUUCAACAGCUACAUGGAGCUGCUGCGAAGUGGUUCCCUGGGCCUGGCACAGGACGAUCCGGCACUGACCGCCCAGGUGGCGGCAGCC ((((.(((((.....)))))(((((.((....))))))).(((((..(((((..((....))..)))))((((((..((..(((..(......)..))).))))))))))))).)))).. ( -49.70) >DroVir_CAF1 64157 120 + 1 CUGCAGCAGCGGCAGGUGGCGCUACAGCAGCAAUUCAAUAGCUACAUGGAGCUGCUGCGGAGCGGCUCGCUGGGCCUGCCGCAGGACGAUCCCGCGCUGGCCACACAGGUGGCCGCGGCC (((((((.((((((((((((((((..(.(....).)..))))......((((((((....)))))))))))..))))))))).((......))..)))((((((....)))))))))).. ( -60.80) >DroGri_CAF1 27195 120 + 1 UUGCAGCAACGUCAGGUGGCACUGCCCCAGCAAUUCAACAGCUAUAUAGAGCUGCUGAGAAAUGGUUCGUUGGGUCUCUCCCCAGAUGACCCGGCGCUGGCAACUCAAGUGGCUGCCGCU ..............(((((((..((((((((..((((.(((((......))))).)))).........(((((((((((....))).)))))))))))))..((....)))))))))))) ( -46.40) >DroYak_CAF1 7125 120 + 1 UUGCAGCAACGCCAAGUGGCGUUGACGCAGCAGUUCAACAGCUACAUGGAGCUGCUGAGGAGUGGUUCCCUGGGCUUGGCGCAGGAUGACCCUGCUCUGACCACCCAGGUGGCGGCGGCC ..((..(((((((....)))))))..))((((((((..((......))))))))))..((.((.((..((((((.(..(.(((((.....))))).)..)...))))))..)).))..)) ( -56.80) >DroMoj_CAF1 60559 120 + 1 CUGCAGCAGCGACAGGUCGCACUGCAGCAGCAGCUCAAUAGCUAUAUAGAACUGUUGCGCAGCGGCUCGCUGGGUCUGUCGCAGGACGAUCCGGCAUUGGCUGGUCAGAUCGCUGCCGCU ..((.(((((((..((((((..(((.(((((((.((.(((....))).)).)))))))))))))))).((((((((.(((....))))))))))).((((....)))).))))))).)). ( -56.20) >DroAna_CAF1 1715 120 + 1 CUGCAACAGAGGCAAGUGGCCCUUCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUCAGGAGCGGCUCCCUGGGCUUGUCGCCCGAUGACCCGGCCUUGACCACCCAGGUGGCGGCAGCA ((((....(((((..(((((..((.(((....))).))..)))))..(((((((((....))))))))).((((.(..((....))..))))))))))(.((((....))))).)))).. ( -50.50) >consensus CUGCAGCAACGGCAAGUGGCGCUGCAGCAGCAGUUCAACAGCUACAUGGAGCUGCUGAGGAGCGGCUCCCUGGGCCUGUCGCAGGACGACCCGGCACUGACCACCCAGGUGGCGGCAGCC ..((......(((((((...........(((.........))).((..((((((((....))))))))..)).)))))))............((((.(.(((.....))).).)))))). (-27.29 = -26.10 + -1.19)

| Location | 12,626,032 – 12,626,152 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.72 |
| Mean single sequence MFE | -53.33 |
| Consensus MFE | -27.32 |
| Energy contribution | -26.88 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.64 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.866501 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
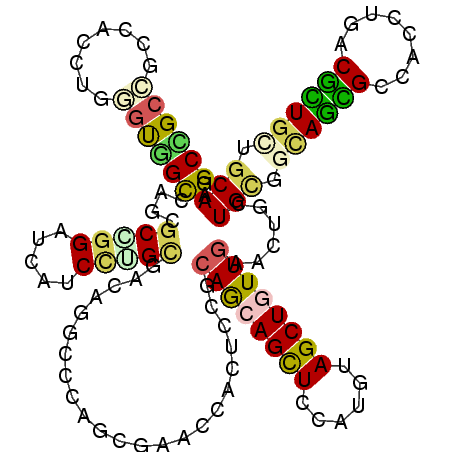
>2L_DroMel_CAF1 12626032 120 - 22407834 GGCUGCCGCCACCUGGGCGGUCAGUGCCGGAUCGUCCUGUGCCAGGCCCAGGGAACCACUUCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGUGUCAACGCAACUUGACGUUGCUGCAA (((....))).(((((((((((((..(......)..))).)))..)))))))..........((((((((..((.((.((.(((((((....)).))))))).)).))..)))))))).. ( -50.30) >DroVir_CAF1 64157 120 - 1 GGCCGCGGCCACCUGUGUGGCCAGCGCGGGAUCGUCCUGCGGCAGGCCCAGCGAGCCGCUCCGCAGCAGCUCCAUGUAGCUAUUGAAUUGCUGCUGUAGCGCCACCUGCCGCUGCUGCAG ((((((((((((....))))))..((((((.....)))))))).)))).((((...))))..((((((((.....(((((.........))))).((((......)))).)))))))).. ( -59.10) >DroGri_CAF1 27195 120 - 1 AGCGGCAGCCACUUGAGUUGCCAGCGCCGGGUCAUCUGGGGAGAGACCCAACGAACCAUUUCUCAGCAGCUCUAUAUAGCUGUUGAAUUGCUGGGGCAGUGCCACCUGACGUUGCUGCAA .((((((((...........(((((...(((((.(((....)))))))).............(((((((((......)))))))))...)))))(((...))).......)))))))).. ( -49.50) >DroYak_CAF1 7125 120 - 1 GGCCGCCGCCACCUGGGUGGUCAGAGCAGGGUCAUCCUGCGCCAAGCCCAGGGAACCACUCCUCAGCAGCUCCAUGUAGCUGUUGAACUGCUGCGUCAACGCCACUUGGCGUUGCUGCAA (((((((........)))))))..((((((((..(((((.((...)).))))).))).....(((((((((......))))))))).)))))(((.(((((((....))))))).))).. ( -57.70) >DroMoj_CAF1 60559 120 - 1 AGCGGCAGCGAUCUGACCAGCCAAUGCCGGAUCGUCCUGCGACAGACCCAGCGAGCCGCUGCGCAACAGUUCUAUAUAGCUAUUGAGCUGCUGCUGCAGUGCGACCUGUCGCUGCUGCAG .((((((((((((((.((.((....)).)).(((.....)))))))..(((...((.((((((((.((((((.(((....))).)))))).))).)))))))...))))))))))))).. ( -53.00) >DroAna_CAF1 1715 120 - 1 UGCUGCCGCCACCUGGGUGGUCAAGGCCGGGUCAUCGGGCGACAAGCCCAGGGAGCCGCUCCUGAGCAGCUCCAUGUAGCUGUUGAACUGCUGCUGAAGGGCCACUUGCCUCUGUUGCAG ..((((.(((((....)))))..((((..((((.((((((.....))))..(((((.(((....))).)))))..(((((.(.....).))))).))..))))....)))).....)))) ( -50.40) >consensus GGCCGCCGCCACCUGGGUGGCCAGCGCCGGAUCAUCCUGCGACAGGCCCAGCGAACCACUCCGCAGCAGCUCCAUGUAGCUGUUGAACUGCUGCGGCAGCGCCACCUGACGCUGCUGCAA .((((((........))))))....(((((.....))))).......................((((((((......))))))))......(((.((((((........)))))).))). (-27.32 = -26.88 + -0.44)
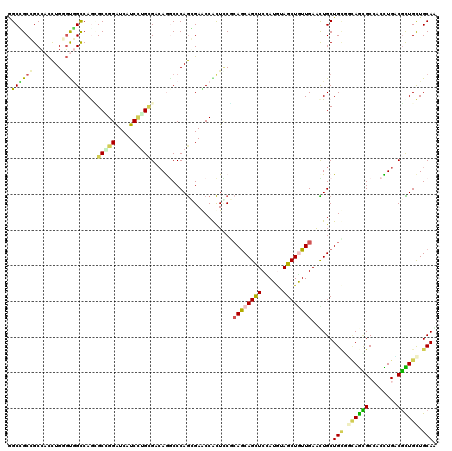
| Location | 12,626,152 – 12,626,242 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 80.72 |
| Mean single sequence MFE | -30.57 |
| Consensus MFE | -24.78 |
| Energy contribution | -24.67 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.47 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.755046 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12626152 90 + 22407834 CAGUUCCUAAUGCAGAGCCAACUGCAGGCCCUCAGUCAGGCCAGUCAGCAAUUGCAGGCACUGCAGAAGCAACAGCAGCGCCAGGUGGAU-- ....(((...(((((......)))))((((........)))).....((....)).(((.((((..........)))).)))....))).-- ( -28.20) >DroVir_CAF1 64277 83 + 1 CAGUUCCUCAUGCAGAGCCAGAUGCAGGCACUGGCCCAGGCCACGCAGCAGCUGCAGGCGCUGCAGAAGCAGCAGGA---------AUCGCU ..((((((........(((...(((((((..((((....))))....))..))))))))(((((....)))))))))---------)).... ( -31.80) >DroSec_CAF1 7103 90 + 1 CAGUUCCUAAUGCAGAGUCAACUGCAGGCCCUCAGCCAGGCCAGUCAGCAAUUGCAGGCACUGCAGAAGCAACAGCAGCGCCAAAUGGAU-- ....(((...(((((......)))))((((........)))).....((....)).(((.((((..........)))).)))....))).-- ( -28.20) >DroSim_CAF1 7188 90 + 1 CAGUUCCUAAUGCAGAGCCAACUGCAGGCCCUCAGCCAGGCCAGUCAGCAAUUGCAGGCACUGCAGAAGCAACAGCAGCGCCAAAUGGAU-- ....(((...(((((......)))))((((........)))).....((....)).(((.((((..........)))).)))....))).-- ( -28.20) >DroEre_CAF1 7047 90 + 1 CAGUUCCUAAUGCAGAGUCAACUGCAGGCCCUGAGCCAGGCCACCCAGCAGCUGCAGGCACUGCAGAAGCAGCAGCAGCGCCAGGCGGAU-- (((..(((...((((......)))))))..))).(((.(((......((..((((((...))))))..)).((....))))).)))....-- ( -33.20) >DroMoj_CAF1 60679 83 + 1 CAGUUCCUGAUGCAGAGCCAGCUGCAGGCGCUGUCCCAGGCCACACAGCAGCUGCAGGCGCUGCAGAAGCAACAGGA---------GACGCU ..(((.((..(((((.(((((((((.(((.(((...)))))).....))))))...))).)))))..)).))).(..---------..)... ( -33.80) >consensus CAGUUCCUAAUGCAGAGCCAACUGCAGGCCCUCAGCCAGGCCACUCAGCAACUGCAGGCACUGCAGAAGCAACAGCAGCGCCA__UGGAU__ ..(((.((..(((((.(((.......((((........)))).....((....)).))).)))))..)).)))................... (-24.78 = -24.67 + -0.11)

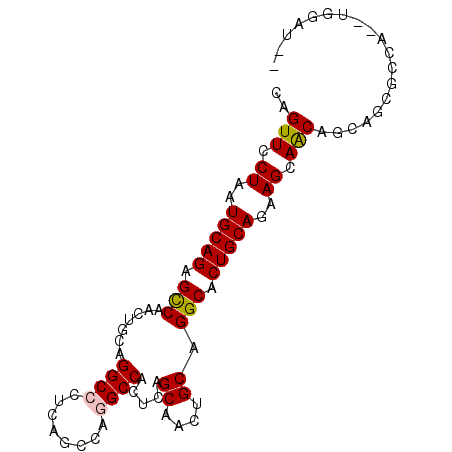
| Location | 12,626,152 – 12,626,242 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 80.72 |
| Mean single sequence MFE | -38.03 |
| Consensus MFE | -27.67 |
| Energy contribution | -28.20 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.922867 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12626152 90 - 22407834 --AUCCACCUGGCGCUGCUGUUGCUUCUGCAGUGCCUGCAAUUGCUGACUGGCCUGACUGAGGGCCUGCAGUUGGCUCUGCAUUAGGAACUG --.(((....((((((((.(......).))))))))((((...((..((((((((......)))))...)))..))..))))...))).... ( -39.20) >DroVir_CAF1 64277 83 - 1 AGCGAU---------UCCUGCUGCUUCUGCAGCGCCUGCAGCUGCUGCGUGGCCUGGGCCAGUGCCUGCAUCUGGCUCUGCAUGAGGAACUG .....(---------(((((((((....)))))((((((((...))))).)))..(((((((((....)).)))))))......)))))... ( -36.10) >DroSec_CAF1 7103 90 - 1 --AUCCAUUUGGCGCUGCUGUUGCUUCUGCAGUGCCUGCAAUUGCUGACUGGCCUGGCUGAGGGCCUGCAGUUGACUCUGCAUUAGGAACUG --.(((....((((((((.(......).))))))))((((...((((...(((((......)))))..))))......))))...))).... ( -35.60) >DroSim_CAF1 7188 90 - 1 --AUCCAUUUGGCGCUGCUGUUGCUUCUGCAGUGCCUGCAAUUGCUGACUGGCCUGGCUGAGGGCCUGCAGUUGGCUCUGCAUUAGGAACUG --.(((....((((((((.(......).))))))))((((...((..((((((((......)))))...)))..))..))))...))).... ( -39.20) >DroEre_CAF1 7047 90 - 1 --AUCCGCCUGGCGCUGCUGCUGCUUCUGCAGUGCCUGCAGCUGCUGGGUGGCCUGGCUCAGGGCCUGCAGUUGACUCUGCAUUAGGAACUG --.((((((..(((((((.(((((....)))))....))))).))..)))(((((......)))))(((((......)))))...))).... ( -44.60) >DroMoj_CAF1 60679 83 - 1 AGCGUC---------UCCUGUUGCUUCUGCAGCGCCUGCAGCUGCUGUGUGGCCUGGGACAGCGCCUGCAGCUGGCUCUGCAUCAGGAACUG ...((.---------(((((.(((...(((((...)))))(((((((((.((((((...))).))))))))).)))...))).))))))).. ( -33.50) >consensus __AUCCA__UGGCGCUGCUGUUGCUUCUGCAGUGCCUGCAACUGCUGACUGGCCUGGCUCAGGGCCUGCAGUUGGCUCUGCAUUAGGAACUG ...................(((.((..(((((.((...(((((((.....(((((......))))).))))))))).)))))..)).))).. (-27.67 = -28.20 + 0.53)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:16:33 2006