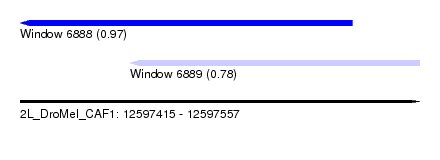
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,597,415 – 12,597,557 |
| Length | 142 |
| Max. P | 0.968315 |

| Location | 12,597,415 – 12,597,533 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.69 |
| Mean single sequence MFE | -38.62 |
| Consensus MFE | -16.93 |
| Energy contribution | -16.26 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.44 |
| SVM decision value | 1.62 |
| SVM RNA-class probability | 0.968315 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
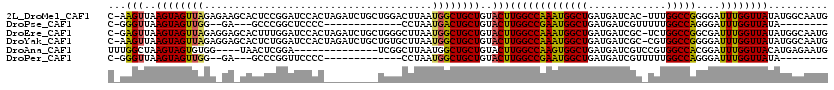
>2L_DroMel_CAF1 12597415 118 - 22407834 C-AAGUUAAGUAGUUAGAGAAGCACUCCGGAUCCACUAGAUCUGCUGGACUUAAUGGCUGCUGUACUUGGCCAAAUGGCUGAUGAUCAC-UUUGGCCGGGGAUUUGGUUAUAUGGCAAUG (-((((..((((((((...(((..(..((((((.....))))))..)..)))..))))))))..)))))((((.((((((...((((.(-((.....))))))).)))))).)))).... ( -38.40) >DroPse_CAF1 216704 93 - 1 C-GGGUUAAGUAGUUGG--GA---GCCCGGCUCCCC-------------CCUAAUGACUGCUGUACUUGGCCGAAUGGCUGAUGAUCGUUUUUGGCCAGGGAUUUGGUUAUA-------- (-(((((.(((((((((--((---((...)))))).-------------......)))))))...((((((((((.(((........)))))))))))))))))))......-------- ( -33.61) >DroEre_CAF1 162378 118 - 1 C-GAGUUAAGUAGUUAGAGGAGCACUUUGGAUCCACUAGAUCUGCUGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCUGAUGAUCGC-UCUGGCCGGCGAUUUGGUUAUAUGGCAAUG (-((((..((((((((...((((.((..(((((.....)))))...))))))..))))))))..)))))((((.((((((...((((((-(......))))))).)))))).)))).... ( -44.60) >DroYak_CAF1 167998 118 - 1 C-AAGUUAAGUAGUUAGAGGAGCACUCUGGAUCCACUAGAUCUGCUGUGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCUGAUGAUCGC-CGUGGCCGGGGAUUUGGUUAUAUGGCAAUG (-((((..((((((((...((((((..((((((.....))))))..))))))..))))))))..)))))((((.((((((...((((.(-((....))).)))).)))))).)))).... ( -49.60) >DroAna_CAF1 163297 102 - 1 UUUGGCUAAGUAGUGUGG----UAACUCGGA--------------UCGGCUUAAUGGCUGCUGUACUUGGCCAAGUGGCUGAUGAUCGUCCGUGGCCACGGAUUUGGUUACAUGAGAAUG .(((((((((((((....----..)).(((.--------------.(((((....)))))))))))))))))))(((((..(.....((((((....)))))))..)))))......... ( -34.80) >DroPer_CAF1 213839 93 - 1 C-GGGUUAAGUAGUUGG--GA---GCCCGGUUCCCC-------------CCUAAUGGCUGCUGUACUUGGCCGAAUGGCUGAUGAUCGUUUUUGGCCAGGGAUUUGGUUAUA-------- (-(((((.(((((((((--((---((...)))))).-------------......)))))))...((((((((((.(((........)))))))))))))))))))......-------- ( -30.71) >consensus C_GAGUUAAGUAGUUAGA_GA_CACCCCGGAUCCAC_________UGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCUGAUGAUCGC_UUUGGCCAGGGAUUUGGUUAUAUGGCAAUG ...(((..((((((((......................................))))))))..)))(((((((((((((.............)))))....)))))))).......... (-16.93 = -16.26 + -0.66)
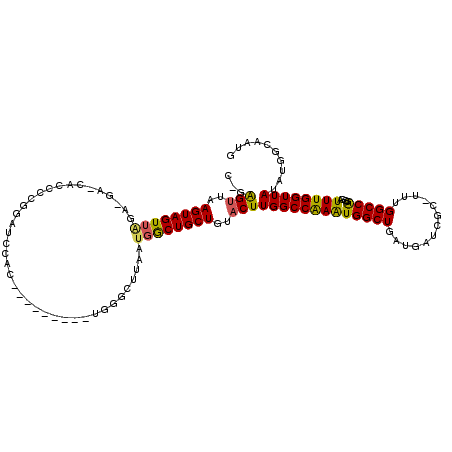

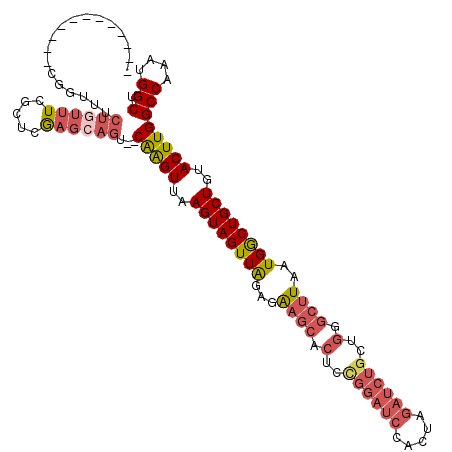
| Location | 12,597,454 – 12,597,557 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.32 |
| Mean single sequence MFE | -34.65 |
| Consensus MFE | -25.42 |
| Energy contribution | -26.93 |
| Covariance contribution | 1.52 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.776896 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12597454 103 - 22407834 ------------CGGUUUCUGUUUCGCUCGAGCAGU--CAAGUUAAGUAGUUAGAGAAGCACUCCGGAUCCACUAGAUCUGCUGGACUUAAUGGCUGCUGUACUUGGCCAAAUGGCU ------------......((((((.....)))))).--(((((..((((((((...(((..(..((((((.....))))))..)..)))..))))))))..)))))(((....))). ( -32.40) >DroPse_CAF1 216736 85 - 1 ------------UGGUUG--GACUCGCAGACGGAGUCCCGGGUUAAGUAGUUGG--GA---GCCCGGCUCCCC-------------CCUAAUGACUGCUGUACUUGGCCGAAUGGCU ------------.....(--(((((.......))))))(((((..(((((((((--((---((...)))))).-------------......)))))))..)))))(((....))). ( -32.31) >DroSec_CAF1 164470 103 - 1 ------------CGGUUUCUGUUUCGCUCGAGCAGU--CAAGUUAAGUAGUUAGAGAAGCACUCCGGAUCCACUAGAUCUGCUGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCU ------------......((((((.....)))))).--(((((..((((((((...((((.(..((((((.....))))))..).))))..))))))))..)))))(((....))). ( -34.60) >DroSim_CAF1 167856 103 - 1 ------------CGGUUUCUGUUUCGCUCGAGCAGU--CAAGUUAAGUAGUUAGAGAAGCACUCCGGAUCCACUAGAUCUGCUGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCU ------------......((((((.....)))))).--(((((..((((((((...((((.(..((((((.....))))))..).))))..))))))))..)))))(((....))). ( -34.60) >DroEre_CAF1 162417 115 - 1 AGGUUUCGGUUUCGGUUUCUUUUUCGCUCGAGCAGU--CGAGUUAAGUAGUUAGAGGAGCACUUUGGAUCCACUAGAUCUGCUGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCU (((..(((....)))..))).....(((......((--(((((..((((((((...((((.((..(((((.....)))))...))))))..))))))))..))))))).....))). ( -32.60) >DroYak_CAF1 168037 114 - 1 -GGGUUCGGGUUCGGUUUCUGUUUCGUUCGAGCAGU--CAAGUUAAGUAGUUAGAGGAGCACUCUGGAUCCACUAGAUCUGCUGUGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCU -..((((((...(((........))).))))))...--(((((..((((((((...((((((..((((((.....))))))..))))))..))))))))..)))))(((....))). ( -41.40) >consensus ____________CGGUUUCUGUUUCGCUCGAGCAGU__CAAGUUAAGUAGUUAGAGAAGCACUCCGGAUCCACUAGAUCUGCUGGGCUUAAUGGCUGCUGUACUUGGCCAAAUGGCU ..................((((((.....))))))...(((((..((((((((...((((.(..((((((.....))))))..).))))..))))))))..)))))(((....))). (-25.42 = -26.93 + 1.52)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:16:16 2006