| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,593,740 – 12,593,837 |
| Length | 97 |
| Max. P | 0.960850 |

| Location | 12,593,740 – 12,593,837 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 113 |
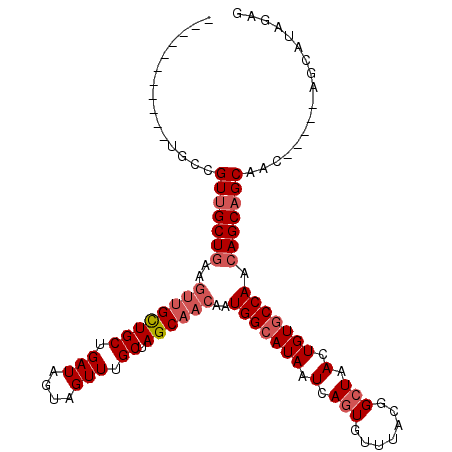
| Reading direction | forward |
| Mean pairwise identity | 84.63 |
| Mean single sequence MFE | -35.22 |
| Consensus MFE | -26.86 |
| Energy contribution | -27.83 |
| Covariance contribution | 0.97 |
| Combinations/Pair | 1.10 |
| Mean z-score | -4.20 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.960850 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12593740 97 + 22407834 UUCUUUGCU-----GUUGCUGCUGUUGGCACAGUUAGCCGUAAACACUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAGCAACUUCAGCAACGGCA----------- .....((((-----(((((((....(((((.((((((.........)))))).)))))..((((((.((............))))))))..)))))))))))----------- ( -36.40) >DroSec_CAF1 160698 97 + 1 CUCUAUGUU-----GUUGCUGCUGUUGGCACAGUUAGCCGUAAACACUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAGCAACUUCAGCAACGGCA----------- .....((((-----(((((((....(((((.((((((.........)))))).)))))..((((((.((............))))))))..)))))))))))----------- ( -34.00) >DroEre_CAF1 158781 97 + 1 CCCUGUGCU-----GUUGCUGCUGUUGGCACAGUUAGCCGUACACACUGAUUAUGCCAUUGUUGCUAGCAAACCACUAUCAGCAGCAACUUCAGCGACGGCA----------- .....((((-----(((((((....(((((.((((((.........)))))).)))))..((((((.((............))))))))..)))))))))))----------- ( -36.10) >DroYak_CAF1 164125 97 + 1 CUCUAUGCU-----GUUGCUGCUGUUGGCACAGUUAGCCGUAAACACUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAGCAACUUCAGCAACGGCA----------- .....((((-----(((((((....(((((.((((((.........)))))).)))))..((((((.((............))))))))..)))))))))))----------- ( -36.50) >DroAna_CAF1 159646 90 + 1 -------AU-----GUUGCUGCUGUUGGAACAGUUAGCUGUAAACAUUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAGCAACUACAGCAACAUCG----------- -------..-----((((((((((.(((...((((.((((..((((.((.......)).))))..)))).)))).))).)))))))))).............----------- ( -30.20) >DroPer_CAF1 209650 107 + 1 CUCUAUGCUGUUCUGUUGCUGCUGUUGGCACAGUUAGCUGUAAGAACUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAAC------AGCAACGGCACGGGAAACGGC .((..((((((((((((((((....(((((.((((((.........)))))).)))))((((.....))))........)))))))------)).)))))))..))....... ( -38.10) >consensus CUCUAUGCU_____GUUGCUGCUGUUGGCACAGUUAGCCGUAAACACUGAUUAUGCCAUUGUUGCUAGCAAACUACUAUCAGCAGCAACUUCAGCAACGGCA___________ .....((((.....((((((((((.(((((.((((((.........)))))).)))))((((.....))))........))))))))))...))))................. (-26.86 = -27.83 + 0.97)

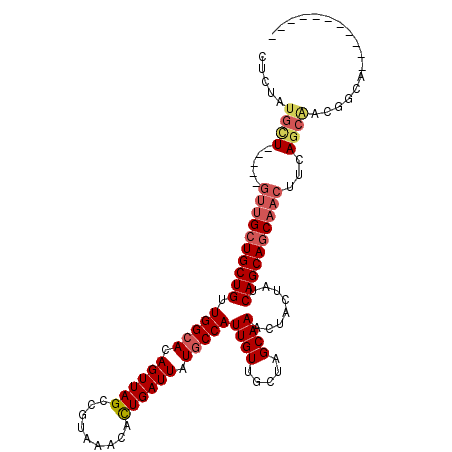
| Location | 12,593,740 – 12,593,837 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 84.63 |
| Mean single sequence MFE | -34.08 |
| Consensus MFE | -26.59 |
| Energy contribution | -27.62 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.917080 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12593740 97 - 22407834 -----------UGCCGUUGCUGAAGUUGCUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAGUGUUUACGGCUAACUGUGCCAACAGCAGCAAC-----AGCAAAGAA -----------(((.(((((((..((((((((.(((....))).)).))))))..(((((((.(.(((.......))).).)))))))....)))))))-----.)))..... ( -35.20) >DroSec_CAF1 160698 97 - 1 -----------UGCCGUUGCUGAAGUUGCUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAGUGUUUACGGCUAACUGUGCCAACAGCAGCAAC-----AACAUAGAG -----------....(((((((..((((((((.(((....))).)).))))))..(((((((.(.(((.......))).).))))))).)))))))...-----......... ( -32.10) >DroEre_CAF1 158781 97 - 1 -----------UGCCGUCGCUGAAGUUGCUGCUGAUAGUGGUUUGCUAGCAACAAUGGCAUAAUCAGUGUGUACGGCUAACUGUGCCAACAGCAGCAAC-----AGCACAGGG -----------..((((.(((...(((((((((((((.((((((((((.......))))).))))).)))((((((....))))))...))))))))))-----))))).)). ( -38.80) >DroYak_CAF1 164125 97 - 1 -----------UGCCGUUGCUGAAGUUGCUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAGUGUUUACGGCUAACUGUGCCAACAGCAGCAAC-----AGCAUAGAG -----------(((.(((((((..((((((((.(((....))).)).))))))..(((((((.(.(((.......))).).)))))))....)))))))-----.)))..... ( -35.00) >DroAna_CAF1 159646 90 - 1 -----------CGAUGUUGCUGUAGUUGCUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAAUGUUUACAGCUAACUGUUCCAACAGCAGCAAC-----AU------- -----------..((((((((((.((((((((.(((....))).)).))))))..............((((.((((....))))...))))))))))))-----))------- ( -29.40) >DroPer_CAF1 209650 107 - 1 GCCGUUUCCCGUGCCGUUGCU------GUUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAGUUCUUACAGCUAACUGUGCCAACAGCAGCAACAGAACAGCAUAGAG ..........((((.(((.((------(((((((.((((.....)))).......(((((((.(.((((.....)))).).)))))))....)))))))))))).)))).... ( -34.00) >consensus ___________UGCCGUUGCUGAAGUUGCUGCUGAUAGUAGUUUGCUAGCAACAAUGGCAUAAUCAGUGUUUACGGCUAACUGUGCCAACAGCAGCAAC_____AGCAUAGAG ...............(((((((..((((((((.(((....))).)).))))))..(((((((.(.(((.......))).).))))))).)))))))................. (-26.59 = -27.62 + 1.03)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:16:12 2006