
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
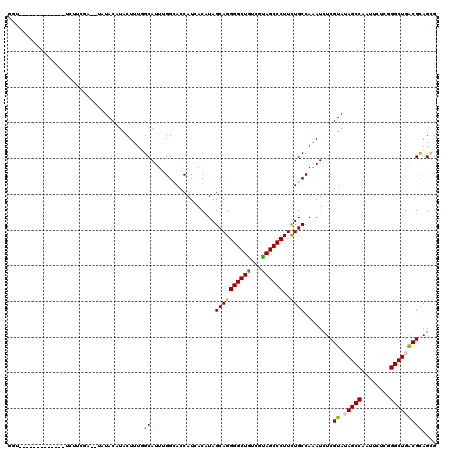
| Location | 12,460,567 – 12,460,707 |
| Length | 140 |
| Max. P | 0.942083 |

| Location | 12,460,567 – 12,460,674 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.34 |
| Mean single sequence MFE | -32.13 |
| Consensus MFE | -21.66 |
| Energy contribution | -21.13 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.590450 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12460567 107 + 22407834 GGU-------------UGUGCGAGGUAUACAUACUUUUGCAUUAGGCACCAUCACAUAGCAGGGGCUGUCGUAGCCCUUCUGCCAAAUCUCGUAUAGCCAAUUCUCUGGCUGGCGCAGCG .((-------------((((((((((...........(((.....)))..........((((((((((...))))))..))))...)))))...((((((......))))))))))))). ( -38.40) >DroVir_CAF1 38485 107 + 1 GGU-------------AACUCUUACGGUACAUACUUUGGCAUUUGGCACCAUAACAUAGCAGGGGCUAUCGUAGCCCUUCUGCCAGAUCUCGUAUAGCCAAUUCUCGGGCUGACGCAGCG .((-------------(.((.....)))))....((((((...((....))......((.((((((((...)))))))))))))))).(((((.(((((........)))))))).)).. ( -27.80) >DroGri_CAF1 34711 97 + 1 UGU-----------------------AUACGUACUUUGGCAUUGGGGACCAUUACAUAGCAGGGGCUGUCGUAGCCCUUCUGCCAAAUUUCGUAUAGCCAAUUUUCUGGCUGACGCAGCG (((-----------------------.((((...(((((((..((((....(((((((((....))))).))))))))..)))))))...))))((((((......))))))..)))... ( -34.50) >DroWil_CAF1 39317 102 + 1 ---------------AUUUUUGA---GUACAUACUUUUGCAUUUGGCACCAUGACGUAGCAGGGGCUAUCGUAGCCCUUUUGCCAGAUCUCGUAUAGCCAAUUCUCGGGCUGACGUAGUG ---------------......((---((....))))....((((((((...((......))(((((((...)))))))..))))))))(((((.(((((........)))))))).)).. ( -26.60) >DroMoj_CAF1 34584 105 + 1 CUU-------------UCUUCUU--UAUACAUACUUUGGCAUUUGGCACCAUCACAUAGCAGGGGCUGUCAUAGCCCUUCUGCCAAAUUUCGUAGAGCCAAUUUUCAGGCUGACGUAGAG ...-------------....(((--(((.....((.(((.((((((((..........(.((((((((...))))))))))))))))).))).))((((........))))...)))))) ( -27.80) >DroAna_CAF1 39448 120 + 1 GUCUCUCAGGGAUAUAUCUUUGAGAUAUACAUACUUUUGCAUUGGGAACCAUCACAUAGCAGGGGCUGUCGCAGCCCUUCUGCCAGAUCUCGUAGAGCCAGUUCUCGGGCUGACGCAGCG ...(((((((((....))))))))).........((((((..((.((....)).))..))))))((((.((((((((((((((........))))))...(....))))))).)))))). ( -37.70) >consensus GGU_____________UCUUCGA__UAUACAUACUUUGGCAUUUGGCACCAUCACAUAGCAGGGGCUGUCGUAGCCCUUCUGCCAAAUCUCGUAUAGCCAAUUCUCGGGCUGACGCAGCG ......................................((..................((((((((((...))))))..))))........((.(((((........))))))))).... (-21.66 = -21.13 + -0.52)


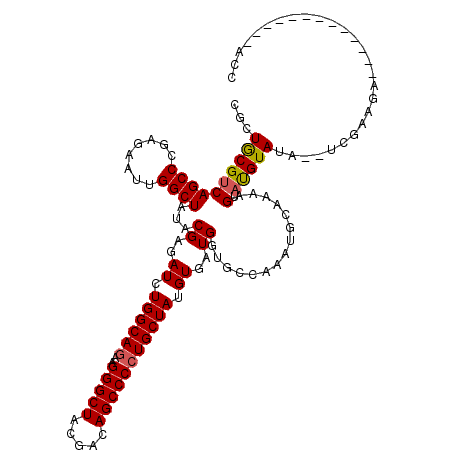
| Location | 12,460,567 – 12,460,674 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.34 |
| Mean single sequence MFE | -31.43 |
| Consensus MFE | -22.33 |
| Energy contribution | -22.30 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.752838 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12460567 107 - 22407834 CGCUGCGCCAGCCAGAGAAUUGGCUAUACGAGAUUUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUGAUGGUGCCUAAUGCAAAAGUAUGUAUACCUCGCACA-------------ACC .((.(((((((((((....))))))..........((((((..(((((.....)))))))))))......)))))...(((((......))))).....))...-------------... ( -34.10) >DroVir_CAF1 38485 107 - 1 CGCUGCGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAGAAGGGCUACGAUAGCCCCUGCUAUGUUAUGGUGCCAAAUGCCAAAGUAUGUACCGUAAGAGUU-------------ACC ..((.(((.((((........))))..)))))...((((((..((((((...))))))))))))(.(((((((((...((((....))))))))))))).)...-------------... ( -33.50) >DroGri_CAF1 34711 97 - 1 CGCUGCGUCAGCCAGAAAAUUGGCUAUACGAAAUUUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUAAUGGUCCCCAAUGCCAAAGUACGUAU-----------------------ACA .........((((((....))))))(((((...(((((((...((((((..(((((....))..)))..))))))....)))))))...)))))-----------------------... ( -30.90) >DroWil_CAF1 39317 102 - 1 CACUACGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAAAAGGGCUACGAUAGCCCCUGCUACGUCAUGGUGCCAAAUGCAAAAGUAUGUAC---UCAAAAAU--------------- ..............(((..(((((....((.(((.(((((...((((((...)))))).))))).))).))..)))))((((....))))...)---))......--------------- ( -28.40) >DroMoj_CAF1 34584 105 - 1 CUCUACGUCAGCCUGAAAAUUGGCUCUACGAAAUUUGGCAGAAGGGCUAUGACAGCCCCUGCUAUGUGAUGGUGCCAAAUGCCAAAGUAUGUAUA--AAGAAGA-------------AAG .(((.(((.((((........))))..)))...(((((((...(((((((.(((((....))..))).))))).))...))))))).........--.)))...-------------... ( -27.90) >DroAna_CAF1 39448 120 - 1 CGCUGCGUCAGCCCGAGAACUGGCUCUACGAGAUCUGGCAGAAGGGCUGCGACAGCCCCUGCUAUGUGAUGGUUCCCAAUGCAAAAGUAUGUAUAUCUCAAAGAUAUAUCCCUGAGAGAC .((((((.((((((.....(..(.((.....)).)..).....)))))))).))))...((((.((((.(((...))).))))..))))......(((((..((....))..)))))... ( -33.80) >consensus CGCUGCGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUGAUGGUGCCAAAUGCAAAAGUAUGUAUA__UCGAAGA_____________ACC ...((((((((((........))))...((..((.((((((..(((((.....))))))))))).))..))...............).)))))........................... (-22.33 = -22.30 + -0.03)

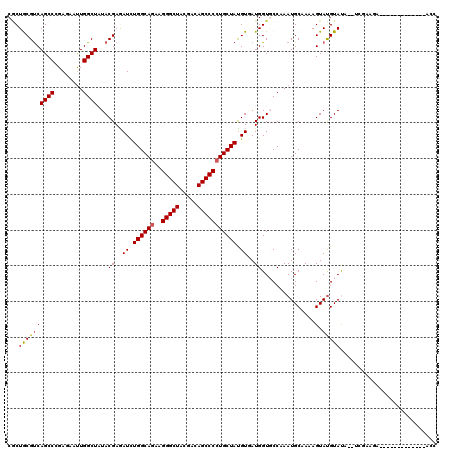
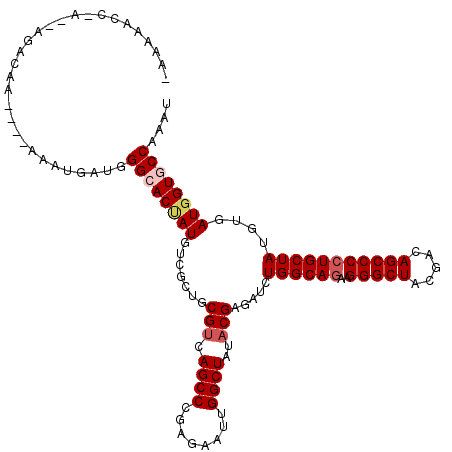
| Location | 12,460,594 – 12,460,707 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.14 |
| Mean single sequence MFE | -36.90 |
| Consensus MFE | -31.08 |
| Energy contribution | -31.75 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.64 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.942083 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12460594 113 - 22407834 -AAAACCCAA--AGCCAA----AUAUGAUGGGCACUAUGUCGCUGCGCCAGCCAGAGAAUUGGCUAUACGAGAUUUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUGAUGGUGCCUAAU -.........--......----......((((((((..(((((..((..((((((....))))))...)).....((((((..(((((.....))))))))))).))))))))))))).. ( -38.70) >DroVir_CAF1 38512 112 - 1 AAAAAACA----ACAUAA----AUAUGAUGGGCACCAUGUCGCUGCGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAGAAGGGCUACGAUAGCCCCUGCUAUGUUAUGGUGCCAAAU ......((----.(((..----..))).))(((((((((.((((.(((.((((........))))..)))))...((((((..((((((...)))))))))))))).))))))))).... ( -39.30) >DroGri_CAF1 34728 112 - 1 -CAAACAC-C--AGUGAA----AAAUGAUGGGCACAAUGUCGCUGCGUCAGCCAGAAAAUUGGCUAUACGAAAUUUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUAAUGGUCCCCAAU -.....((-(--((((..----....(((((((........))).))))((((((....)))))).)))......((((((..(((((.....))))))))))).....))))....... ( -31.80) >DroWil_CAF1 39339 119 - 1 -AAAAACCAACCACACAAAUCAUAAUGAUGGGCACUAUGUCACUACGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAAAAGGGCUACGAUAGCCCCUGCUACGUCAUGGUGCCAAAU -.................(((.....))).(((((((((.(.((.(((.((((........))))..)))))...(((((...((((((...)))))).))))).).))))))))).... ( -37.00) >DroMoj_CAF1 34609 111 - 1 -UAAAUAU----AUACAA----AAAUGAUGGGCACCAUGUCUCUACGUCAGCCUGAAAAUUGGCUCUACGAAAUUUGGCAGAAGGGCUAUGACAGCCCCUGCUAUGUGAUGGUGCCAAAU -.......----......----........((((((((.......(((.((((........))))..))).....((((((..(((((.....)))))))))))....)))))))).... ( -37.90) >DroAna_CAF1 39488 113 - 1 -AAAAGCCAA--AGCAUA----UAAUGAUGGGCACUAUGUCGCUGCGUCAGCCCGAGAACUGGCUCUACGAGAUCUGGCAGAAGGGCUGCGACAGCCCCUGCUAUGUGAUGGUUCCCAAU -....((...--.))...----......((((.(((((.(((..(.(((((........))))).)..))).((.((((((..((((((...)))))))))))).)).))))).)))).. ( -36.70) >consensus _AAAAACC_A__AGACAA____AAAUGAUGGGCACUAUGUCGCUGCGUCAGCCCGAGAAUUGGCUAUACGAGAUCUGGCAGAAGGGCUACGACAGCCCCUGCUAUGUGAUGGUGCCAAAU ..............................((((((((.......(((.((((........))))..))).....((((((..(((((.....)))))))))))....)))))))).... (-31.08 = -31.75 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:38 2006