
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,389,295 – 12,389,424 |
| Length | 129 |
| Max. P | 0.979108 |

| Location | 12,389,295 – 12,389,389 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 70.50 |
| Mean single sequence MFE | -39.48 |
| Consensus MFE | -19.07 |
| Energy contribution | -21.04 |
| Covariance contribution | 1.97 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.824317 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
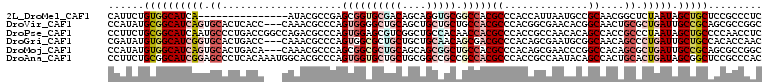
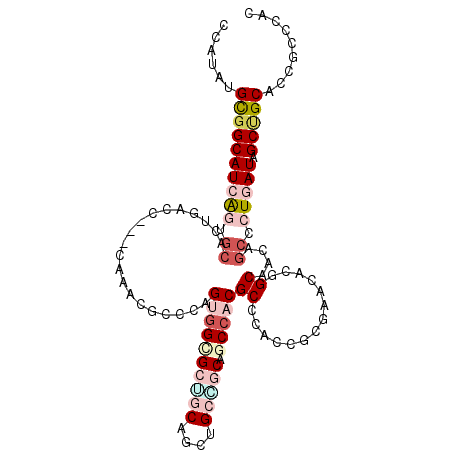
>2L_DroMel_CAF1 12389295 94 + 22407834 CAUUCUGUGGCAUCA---------------AUACGCCGAGCGGUGCGACAGCAGGUGCGGCCACGCCCACCAUUAAUGCCGCAACGGCUCUAAUAGCUGCUCCGCCCUC ........(((....---------------....)))(((.((((.((((((.((((.(((...)))))))((((..((((...))))..)))).)))).))))))))) ( -37.20) >DroVir_CAF1 270853 106 + 1 CCAUAUGCGGCAUCAGUGCACUCACC---CAAACGCCCAGUGGGGCUGCAGCUGCUGCUGCCACGCCCACGGCGAACACGGCAACUGCGCUGAUUGCCGCAGCGCCGGC ((...(((((((((((((((......---....((((..((((((..(((((....)))))..).))))))))).....(....))))))))).))))))).....)). ( -51.60) >DroPse_CAF1 295534 109 + 1 CCUUCUGCGGCAUCAAUGCCCUGACCGGCCAGACGCCCAGUGGAGCGUCGGCUGCCACAACCACGCCCACCGCCAACACAGCCACCGCCCUAAUAGCUGCCCCAACCUC ......(((((.(((......)))..(((..(((((((...)).)))))(((((........................)))))...)))......)))))......... ( -28.56) >DroGri_CAF1 275369 106 + 1 CGAUAUGUGGCAUCGGUGCACUGACC---CAAACGCCCAGUGGCGCUGCUGCUGCAACAGCGACGCCCACAGCGAAUGCGGCAACAGCCCUGAUUGCUGCCACACCAAC .....(((((((.((((.(((((...---........)))))..((((.(((((((...((..........))...))))))).))))....)))).)))))))..... ( -38.20) >DroMoj_CAF1 272497 106 + 1 CCAUAUGUGGCAUCAGUGCACUGACA---CAAACGCCCAGCGGCGCUGCAGCAGCGGCUGCCACGCCCACAGCGAACCCGGCCACAGCGCUGAUUGCCGCAGCGCCGGC ((...((((((.((((....))))..---....(((...(.((((..(((((....)))))..)))))...)))......))))))((((((.......)))))).)). ( -44.50) >DroAna_CAF1 200452 109 + 1 CCUUCUGCGGCAUCGGAGCCCUCACAAAUGGCACGCCCAGUGGUGCUGCUGCGGCCGCCGCCACGCCCACCGCCAAUACAGCCACUGCACUGAUAGCGGCUCCGCCCAC .....((.(((...((((((........((((.......((((((..((.(.(((....))).))).)))))).......))))..((.......))))))))))))). ( -36.84) >consensus CCAUAUGCGGCAUCAGUGCACUGACC___CAAACGCCCAGUGGCGCUGCAGCUGCCGCAGCCACGCCCACCGCGAACACGGCAACAGCCCUGAUAGCUGCACCGCCCAC ......((((((((((.((....................((((((((((....))))).)))))((..............))....)).))))).)))))......... (-19.07 = -21.04 + 1.97)
| Location | 12,389,295 – 12,389,389 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 70.50 |
| Mean single sequence MFE | -51.70 |
| Consensus MFE | -24.57 |
| Energy contribution | -25.41 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.52 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.48 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.979108 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12389295 94 - 22407834 GAGGGCGGAGCAGCUAUUAGAGCCGUUGCGGCAUUAAUGGUGGGCGUGGCCGCACCUGCUGUCGCACCGCUCGGCGUAU---------------UGAUGCCACAGAAUG ..((((((.((.((((((((.((((...)))).))))))))..))(((((.((....)).))))).))))))(((((..---------------..)))))........ ( -43.10) >DroVir_CAF1 270853 106 - 1 GCCGGCGCUGCGGCAAUCAGCGCAGUUGCCGUGUUCGCCGUGGGCGUGGCAGCAGCAGCUGCAGCCCCACUGGGCGUUUG---GGUGAGUGCACUGAUGCCGCAUAUGG .(((....(((((((.((((.(((.(..((.((..(((((((((.((.(((((....))))).)))))))..))))..))---))..).))).)))))))))))..))) ( -58.70) >DroPse_CAF1 295534 109 - 1 GAGGUUGGGGCAGCUAUUAGGGCGGUGGCUGUGUUGGCGGUGGGCGUGGUUGUGGCAGCCGACGCUCCACUGGGCGUCUGGCCGGUCAGGGCAUUGAUGCCGCAGAAGG ...((..(.(((((((((.....))))))))).)..))((((((((((((((...))))).)))).))))).((((((..(((......)))...))))))........ ( -52.10) >DroGri_CAF1 275369 106 - 1 GUUGGUGUGGCAGCAAUCAGGGCUGUUGCCGCAUUCGCUGUGGGCGUCGCUGUUGCAGCAGCAGCGCCACUGGGCGUUUG---GGUCAGUGCACCGAUGCCACAUAUCG ((..(((((((((((........)))))))))))..))(((((((((((((((((...)))))))((.(((((.(.....---).)))))))...)))))).))))... ( -50.20) >DroMoj_CAF1 272497 106 - 1 GCCGGCGCUGCGGCAAUCAGCGCUGUGGCCGGGUUCGCUGUGGGCGUGGCAGCCGCUGCUGCAGCGCCGCUGGGCGUUUG---UGUCAGUGCACUGAUGCCACAUAUGG ((.((((((((((((....(((..((.((((.((((.....)))).)))).)))))))))))))))))))((((((((.(---(((....)))).)))))).))..... ( -54.40) >DroAna_CAF1 200452 109 - 1 GUGGGCGGAGCCGCUAUCAGUGCAGUGGCUGUAUUGGCGGUGGGCGUGGCGGCGGCCGCAGCAGCACCACUGGGCGUGCCAUUUGUGAGGGCUCCGAUGCCGCAGAAGG ((((.(((((((((.......))(((((((((.(..(.((((.(((((((....))))).))..)))).)..)))).))))))......)))))))...))))...... ( -51.70) >consensus GAGGGCGGAGCAGCAAUCAGCGCAGUGGCCGUAUUCGCGGUGGGCGUGGCAGCAGCAGCUGCAGCACCACUGGGCGUUUG___GGUCAGUGCACUGAUGCCACAGAAGG ((.((((..((((((........))))))..)))).)).(((((((((((((...))))))).)).))))..((((((.................))))))........ (-24.57 = -25.41 + 0.84)

| Location | 12,389,320 – 12,389,424 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 71.54 |
| Mean single sequence MFE | -42.56 |
| Consensus MFE | -12.82 |
| Energy contribution | -14.52 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.30 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.627069 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
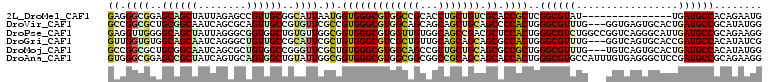
>2L_DroMel_CAF1 12389320 104 + 22407834 CGGUGCGACAGCAGGUGCGGCCACGCCCACCAUUAAUGCCGCAACGGCUCUAAUAGCUGCUCCGCCCUCGGCGGGACGUACCAAUCCGUCGAUGGCGGCCGCAC ..(((((.((((.((((.(((...)))))))((((..((((...))))..)))).))))..(((((.((((((((.........)))))))).))))).))))) ( -50.10) >DroVir_CAF1 270890 104 + 1 UGGGGCUGCAGCUGCUGCUGCCACGCCCACGGCGAACACGGCAACUGCGCUGAUUGCCGCAGCGCCGGCAGCGCAGCGCACGAAUCCAUCAAUGGCAGCCGCAU ((.((((((.(((((.((((((.((((...)))).....(((..((((((.....).))))).))))))))))))))....((.....))....)))))).)). ( -55.00) >DroPse_CAF1 295574 104 + 1 UGGAGCGUCGGCUGCCACAACCACGCCCACCGCCAACACAGCCACCGCCCUAAUAGCUGCCCCAACCUCGGCCCCGAGACCCAAUCCAUCGAUGGCCGCAGCAC .((.(((..(((((........................)))))..))))).....(((((......((((....))))..(((.((....)))))..))))).. ( -29.96) >DroGri_CAF1 275406 104 + 1 UGGCGCUGCUGCUGCAACAGCGACGCCCACAGCGAAUGCGGCAACAGCCCUGAUUGCUGCCACACCAACAGUGCAACGCACGAAUCCAUCGAUGGCAGCAGCCU .(((((((.(((((((...((..........))...))))))).))))......(((((((((.......((((...))))((.....))).))))))))))). ( -42.60) >DroMoj_CAF1 272534 104 + 1 CGGCGCUGCAGCAGCGGCUGCCACGCCCACAGCGAACCCGGCCACAGCGCUGAUUGCCGCAGCGCCGGCAGCUCAACGCCCGAAUCCUUCAAUGGCCGCAGCGU .((((..(((((....)))))..))))....(((....((((((..(((.(((((((((......)))))).))).)))..((.....))..))))))...))) ( -43.10) >DroAna_CAF1 200492 104 + 1 UGGUGCUGCUGCGGCCGCCGCCACGCCCACCGCCAAUACAGCCACUGCACUGAUAGCGGCUCCGCCCACGGCGACUCGCCCCAACCCCUCGAUGGCAGCCGCGU .(((((.((((.(((....)))..((.....)).....))))....)))))....((((((.((((...))))....(((.............))))))))).. ( -34.62) >consensus UGGCGCUGCAGCUGCCGCAGCCACGCCCACCGCGAACACGGCAACAGCCCUGAUAGCUGCACCGCCCACAGCGCAACGCACCAAUCCAUCGAUGGCAGCAGCAU ....((....)).((.((.((((......((((((.((..((....))..)).))))))...(((.....)))...................)))).)).)).. (-12.82 = -14.52 + 1.70)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:12 2006