
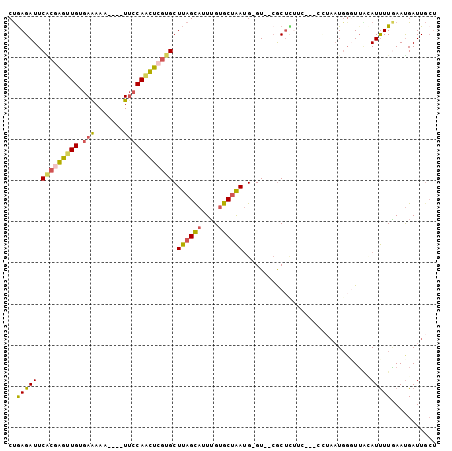
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,323,883 – 12,324,020 |
| Length | 137 |
| Max. P | 0.901734 |

| Location | 12,323,883 – 12,323,980 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 77.03 |
| Mean single sequence MFE | -24.75 |
| Consensus MFE | -13.42 |
| Energy contribution | -13.53 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.560056 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12323883 97 - 22407834 CUGAGAUUCACGAGUUGUGAAAAA----UUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGUGUCGCUUUGC---CUUAAUGGUUUAAAUUUUGAAUGAUUGCU (((((...(((((((((.((....----.)))))))))))))))).(((((.((((..(((((......)))---))...)))).))))).............. ( -24.70) >DroSec_CAF1 146762 95 - 1 CUGAGAUUCACGAGUUGUGAAAAA----UUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGU--CGCUCUUC---CCUAAUGGGUUACAUUUUGAAUGAUUGCU ....(((((((((((((.((....----.))))))))))..((((((....)))))).))))--)((....(---((....)))...(((.....)))...)). ( -25.90) >DroSim_CAF1 150946 92 - 1 CUGAGAUUCACGGGUUGUGAAAAA----UUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGU--CGCUCUUU---CCUAUU---UUCCAUUUUGAAUGAUUGCU (((((...(((((((((.((....----.))))))))))))))))((((((((..((((((.--........---))))))---...)))...)))))...... ( -23.00) >DroEre_CAF1 144191 85 - 1 CUGAGAUUCGCUAGUUGUGAGAAA----UUCCAGCUCGUGCUUAGCAUUUGUGCUAAUG-GU--CGC------------AUGGGUUGCAUUUUGAAUGAUUGCU .........((.(((..(..((((----(..((((((((((((((((....))))))..-..--.))------------)))))))).)))))..)..))))). ( -25.90) >DroYak_CAF1 151968 94 - 1 CUAAGAUUCGCGAGUUGUGAAAAA----UUCCAACUCGUGCUCAGUAUUUGUGCCGAAG-GU--CACUUUUG---GCUAAUGGGUUAAAUCUUAAACGAUUGCU ....((..(((((((((.((....----.))))))))))).))((((.(((((((((((-(.--..))))))---))...((((......)))).)))).)))) ( -27.70) >DroAna_CAF1 135239 98 - 1 CCGAGAUUCCAUAGUUGUAAAAAAUAAAUGGCACUUCGUGCUUAGCAUUGGCGCUAAUG-GU--GGCUCCUUGGUGGUAAUGGGUAGUAUUUUUAAUGAUU--- ............(((..(.(((((((...(((((...)))))...(((((.((((((.(-(.--....)))))))).))))).....))))))).)..)))--- ( -21.30) >consensus CUGAGAUUCACGAGUUGUGAAAAA____UUCCAACUCGUGCUUAGCAUUUGUGCUAAUG_GU__CGCUCUUC___CCUAAUGGGUUACAUUUUGAAUGAUUGCU ..(((((.(((((((((.(((.......)))))))))))).((((((....))))))...............................)))))........... (-13.42 = -13.53 + 0.12)


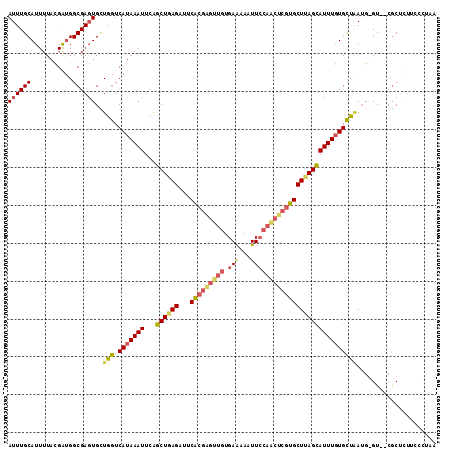
| Location | 12,323,907 – 12,324,020 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 81.34 |
| Mean single sequence MFE | -33.27 |
| Consensus MFE | -22.80 |
| Energy contribution | -23.53 |
| Covariance contribution | 0.73 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901734 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12323907 113 - 22407834 AUUUGCAUUUUACGAUGGCGAGUGCUGGUCAUAAAUUCAGCUGAGAUUCACGAGUUGUGAAAAAUUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGUGUCGCUUUGCCUUAA ....(((.........(((((.((((.(((((((((...((((((...(((((((((.((.....))))))))))))))))))))))))...)).))))))))).)))..... ( -34.50) >DroSec_CAF1 146786 111 - 1 AUUUGCAUUUUACGAUGGCGAGUGCUGGUCAUAAAUUCAGCUGAGAUUCACGAGUUGUGAAAAAUUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGU--CGCUCUUCCCUAA ....((((((.........))))))(((.(((((((...((((((...(((((((((.((.....)))))))))))))))))))))))))))..(((.--.......)))... ( -33.90) >DroSim_CAF1 150967 111 - 1 AUUUGCAUUUUACGAUGGCGAGUGCUGGUCAUAAAUUCAGCUGAGAUUCACGGGUUGUGAAAAAUUCCAACUCGUGCUUAGCAUUUGUGCUAAUGGGU--CGCUCUUUCCUAU .....((((....))))..(((((((.(((((((((...((((((...(((((((((.((.....))))))))))))))))))))))))...)).)).--)))))........ ( -32.70) >DroEre_CAF1 144215 101 - 1 AUUUGCAUUUUACGAUGGCGAGUGUUGGUCAUAAAUUCAGCUGAGAUUCGCUAGUUGUGAGAAAUUCCAGCUCGUGCUUAGCAUUUGUGCUAAUG-GU--CGC---------A .....((((....))))((((.((((((.(((((((...((((((...(((.(((((..(....)..))))).))))))))))))))))))))))-.)--)))---------. ( -34.40) >DroYak_CAF1 151992 110 - 1 AUUUGCAUUUUACGAUGGCGAGUGUUGGUCAUAAAUUCAGCUAAGAUUCGCGAGUUGUGAAAAAUUCCAACUCGUGCUCAGUAUUUGUGCCGAAG-GU--CACUUUUGGCUAA ....(((...(((...(((((((.(((((..........))))).))))((((((((.((.....)))))))))))))..)))..)))(((((((-(.--..))))))))... ( -34.10) >DroPer_CAF1 224858 96 - 1 AUUUGCAUUUUAUGGCGGCGACUGAUGAUCAUAAAUUUGGCUGAGAUUCGCG--------------CCAUCUCAUGCUCAGCAUUUCUGUUUAUG-GU--CGCAGUUUCGCAA ..............((((.(((((.(((((((((((...((((((.......--------------..........))))))......)))))))-))--))))))))))).. ( -30.03) >consensus AUUUGCAUUUUACGAUGGCGAGUGCUGGUCAUAAAUUCAGCUGAGAUUCACGAGUUGUGAAAAAUUCCAACUCGUGCUUAGCAUUUGUGCUAAUG_GU__CGCUCUUCCCUAA ((((((...........))))))..(((.(((((((...((((((...(((((((((.(((...))))))))))))))))))))))))))))..................... (-22.80 = -23.53 + 0.73)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:46 2006