
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,299,258 – 12,299,406 |
| Length | 148 |
| Max. P | 0.776177 |

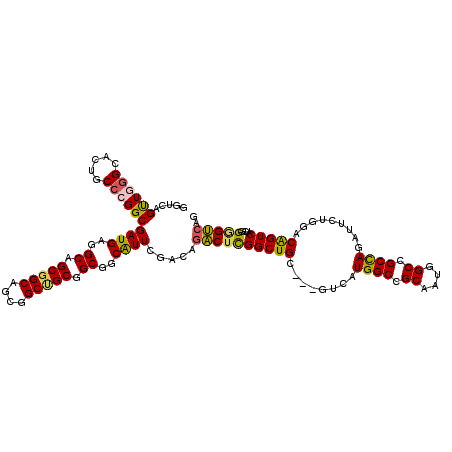
| Location | 12,299,258 – 12,299,375 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -57.05 |
| Consensus MFE | -38.38 |
| Energy contribution | -37.88 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.589335 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
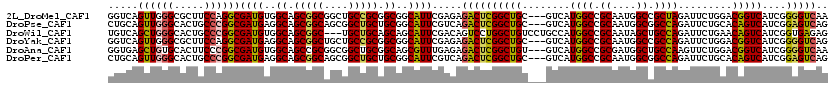
>2L_DroMel_CAF1 12299258 117 - 22407834 GGUCAGUUGGGCGCUUCCAGGCGAUGUGGCAGCGGCGGCUGCCGCGGCGGCAUUCGAGAGACUCGGCUGC---GUCAUGGCCGCAAUGGCCGCUAGAUUCUGGACGGUCAUCGGGGUCAA .........(((((..((.(.((.(((((((((....))))))))).)).)..((....))...))..))---))).(((((.(.(((((((((((...)))).))))))).).))))). ( -56.60) >DroPse_CAF1 184519 117 - 1 CUGCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCUGCGGCAUUCGUCAGACUCGGCUGC---GUCAUGGCCGCAAUGGCGGCCAGAUUCUGCACAGUCAUCGGAGUCAG ..((((((((((...))))(((((((..((((((((....))))))))..)).)))))......))))))---....(((((((....)))))))(((((((.........))))))).. ( -58.90) >DroWil_CAF1 143237 117 - 1 UGUCAGCUGGGCACUGCCCGGCGAUGUGGCAGCGGC---UGCUGCAGCAGCAUUCGACAGUCCUGGCUGUCCUGCCAUGGCCGCAAUAGCUGCCAGAUUCUGAACAGUCAUCGGUGAGAG (.((((((((((...)))))))((((((((((..((---(((....)))))....((((((....))))))))))))((((.((....)).)))).............))))..))).). ( -52.90) >DroYak_CAF1 126364 117 - 1 GGUCAGUUGGGCGCUUCCAGGCGAUGAGGCAGCGGCUGCUGCCGCGGCGGCAUUCGAGAGACUCGGCUGC---GUCAUGGCCGCAAUGGCCGCCAGAUUCUGGACGGUCAUCGGGGUCAG .(((.....)))(.((((.(((.((((.(((((...((((((....))))))..((((...)))))))))---.)))).)))...(((((((((((...)))).))))))).)))).).. ( -56.10) >DroAna_CAF1 110476 117 - 1 GGUGAGCUGUGCACUUCCCGGCGAUGUGGCAGCCGCGGCGGCUGCGGCAGCGUUUGAGAGACUCGGCUGU---GUCAUGGCCGCGAUGGCUGCCAAGUUCUGGACGGUCAUCGGGGUCAA (..((((((.(..((..(..(((.(((.(((((((...))))))).))).)))..)..)).).))))).)---..).(((((.((((((((((((.....))).))))))))).))))). ( -58.90) >DroPer_CAF1 190043 117 - 1 CUGCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCUGCGGCAUUCGUCAGACUCGGCUGC---GUCAUGGCCGCAAUGGCGGCCAGAUUCUGCACAGUCAUCGGAGUCAG ..((((((((((...))))(((((((..((((((((....))))))))..)).)))))......))))))---....(((((((....)))))))(((((((.........))))))).. ( -58.90) >consensus GGUCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCGGCGGCAUUCGACAGACUCGGCUGC___GUCAUGGCCGCAAUGGCCGCCAGAUUCUGGACAGUCAUCGGGGUCAG .....((((((.....))))))((((..((.(((((....))))).))..)))).....((((((((((........((((.((....)).)))).........)))))....))))).. (-38.38 = -37.88 + -0.50)

| Location | 12,299,298 – 12,299,406 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 78.43 |
| Mean single sequence MFE | -45.27 |
| Consensus MFE | -33.17 |
| Energy contribution | -32.87 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.776177 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
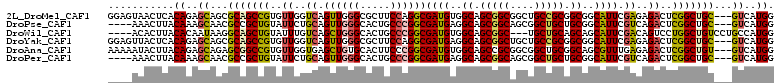
>2L_DroMel_CAF1 12299298 108 - 22407834 GGAGUAACUCACAGAGCAGCGCAGCCGUGUUGGUCAGUUGGGCGCUUCCAGGCGAUGUGGCAGCGGCGGCUGCCGCGGCGGCAUUCGAGAGACUCGGCUGC---GUCAUGG (((((..((.((.((.((((((....)))))).)).)).))..)))))(((((..(((((((((....)))))))))(((((..((....))....)))))---))).)). ( -48.00) >DroPse_CAF1 184559 104 - 1 ----AAACUUACAAAGCAACGCCGCUGUAUUCUGCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCUGCGGCAUUCGUCAGACUCGGCUGC---GUCAUGG ----..............((((.((((...(((((.(((((((...)))))))((((..((((((((....))))))))..)))).).))))..)))).))---))..... ( -44.60) >DroWil_CAF1 143277 104 - 1 ----ACACUUACACAAUAAGGCAGCUGUAUUUGUCAGCUGGGCACUGCCCGGCGAUGUGGCAGCGGC---UGCUGCAGCAGCAUUCGACAGUCCUGGCUGUCCUGCCAUGG ----...............(((((((((...((((.(((((((...)))))))))))..))))).((---(((....)))))....((((((....)))))).)))).... ( -46.80) >DroYak_CAF1 126404 108 - 1 GGAGUUACUCACAGAGCAGCGCAGCCGUGUUGGUCAGUUGGGCGCUUCCAGGCGAUGAGGCAGCGGCUGCUGCCGCGGCGGCAUUCGAGAGACUCGGCUGC---GUCAUGG (((((..((.((.((.((((((....)))))).)).)).))..)))))....(.((((.(((((...((((((....))))))..((((...)))))))))---.)))).) ( -45.40) >DroAna_CAF1 110516 108 - 1 AAAAAUACUUACAGAGCAGAGCGGCCGUGUUGGUGAGCUGUGCACUUCCCGGCGAUGUGGCAGCCGCGGCGGCUGCGGCAGCGUUUGAGAGACUCGGCUGU---GUCAUGG .........(((((.(((.(((.(((.....)))..))).))).((..(..(((.(((.(((((((...))))))).))).)))..)..))......))))---)...... ( -42.20) >DroPer_CAF1 190083 104 - 1 ----AAACUUACAAAGCAACGCCGCUGUAUUCUGCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCUGCGGCAUUCGUCAGACUCGGCUGC---GUCAUGG ----..............((((.((((...(((((.(((((((...)))))))((((..((((((((....))))))))..)))).).))))..)))).))---))..... ( -44.60) >consensus ____AAACUUACAGAGCAACGCAGCCGUAUUGGUCAGUUGGGCACUGCCCGGCGAUGAGGCAGCGGCAGCGGCUGCGGCGGCAUUCGACAGACUCGGCUGC___GUCAUGG ...........((..((...(((((((..((..((.((((((.....))))))((((..((.(((((....))))).))..)))).))..))..)))))))...))..)). (-33.17 = -32.87 + -0.30)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:34 2006