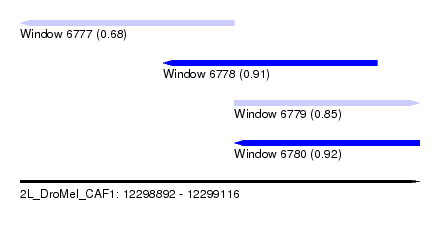
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,298,892 – 12,299,116 |
| Length | 224 |
| Max. P | 0.921559 |

| Location | 12,298,892 – 12,299,012 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.83 |
| Mean single sequence MFE | -42.18 |
| Consensus MFE | -26.88 |
| Energy contribution | -26.97 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.678651 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12298892 120 - 22407834 GCUGCGGCGGAUUGGGCAGGAUGGGCGUGGUCACGCUGGUGGCCGUUUGGCCCAGAGGUAUGUUGAUGUUGCUGGCCAGGUUCCAGAGAUUGGCCUGAAUUUGCUGAAUCUUCUUCUGCU ...((((.(((((.((((((.(((((.(((((((....)))))))....))))).((((..(((..((..(((.....)))..))..)))..))))...)))))).)))))....)))). ( -43.30) >DroVir_CAF1 147450 120 - 1 GUUGCGGUGGGUUUGGCAGUAUCGGUGUGGAUACAGUGGUCGGCGUCUGGCCUAGCGGUAUGUUAAUAUUGGUCGCCAGAUUCCAUAGAUUCGCCUGAAUUUGCUGUAUCUUCUUCUGUU ..(((((..((((((((.(((((......))))).......((.(((((((..(.((((((....)))))).).))))))).))........))).)))))..)))))............ ( -40.50) >DroGri_CAF1 154728 120 - 1 GUUGCGGUGGGUUUGGCAGUAUCGGUGUGGACACUGUGGUUGCCGUUUGGCCCAGUGGUAUAUUUAUGUUUGUGGCCAGAUUCCACAAAUUCGCCUGAAUUUGCUGGAUCUUCUUCUGUU ...((((.(((((..((((..((((.(((((...(((((.....(((((((((((..(((....)))..))).)))))))).)))))..)))))))))..))))..)))))....)))). ( -44.30) >DroWil_CAF1 142872 120 - 1 GUUGCGGUGGGUUUGGCAAUAUAGACGUGGAAACACUUGUCGGCGUUUGGCCCAAUGGUAUGUUUAUAUUAGUGGCCAAGUUCCAUAGAUUUGCCUGUAUCUGUUGAAUCUUCUUUUGCU ...((((.(((((..(((((((((..(((....))).....((..(((((((...((((((....))))))..)))))))..))..........)))))).)))..)))))....)))). ( -39.40) >DroMoj_CAF1 156027 120 - 1 GUUGCGGUGGGUUUGGCAGUAUCGGUGUGGAAACUGUGGUUGGCGUCUGGCCGAGUGGUAUAUUAAUAUUGGUCGCCAGAUUCCAUAGAUUCGCCUGUAUUUGCUGUAUCUUCUUCUGUU ...((((.(((....((((((.(((.(((((..((((((.....(((((((.((.((((((....)))))).))))))))).)))))).))))))))....))))))....))).)))). ( -41.40) >DroPer_CAF1 189684 120 - 1 GCUGCGGUGGGUUCGGCAGUAUCGGUGUGGACACACUCUGGGCCGUCUGUCCCAAGGGGAUGUUGAUAUUGCUGGCCAGGUUCCAGAGAUUCGCCUGGAUCUGCUGGAUCUUCUUUUGUU .........(((.(((((((((((((.((((.....)))).)))....(((((...)))))...)))))))))))))(((.(((((((((((....)))))).))))).)))........ ( -44.20) >consensus GUUGCGGUGGGUUUGGCAGUAUCGGUGUGGACACACUGGUCGCCGUCUGGCCCAGUGGUAUGUUAAUAUUGGUGGCCAGAUUCCAUAGAUUCGCCUGAAUUUGCUGAAUCUUCUUCUGUU ...((((.(((((((((((...(((.(((((....((((.....(((((((.((..(((((....)))))..))))))))).))))...))))))))...)))))))))))....)))). (-26.88 = -26.97 + 0.09)


| Location | 12,298,972 – 12,299,092 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.56 |
| Mean single sequence MFE | -54.88 |
| Consensus MFE | -36.52 |
| Energy contribution | -36.33 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.905551 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12298972 120 - 22407834 CAGUCCCACCGCCUGCAGCUGCUGCAGCAGUUGAAUGGAGGCCGGGGUAAUGGCAUCUGCGCCGAGAACCGGCGAAGGCUGCUGCGGCGGAUUGGGCAGGAUGGGCGUGGUCACGCUGGU .....(((((((((((..((((((((((((((....(((.((((......)))).))).(((((.....)))))..)))))))))))))).....))))).)))((((....))))))). ( -61.80) >DroPse_CAF1 184240 120 - 1 CAGGCCCACGGCCUGCAACUGUUGCAGCAAUUGUAUGGAGGCCGGUGUCAGUGCAUCGGCGCCCAGAACGGGCGACUGCUGCUGCGGUGGGUUCGGCAGUAUCGGUGUGGACACACUCUG ((((....((((((.((((.((((...)))).)).)).))))))(((((...((((((((((((.....)))))(((((((..((.....)).))))))).))))))).)))))..)))) ( -52.80) >DroGri_CAF1 154808 120 - 1 CAGUCCAACUGUUUGCAGCUGCUGCAGCAAUUGAAUGCUGGCUGGUGUCAGAGCAUCGGCUCCCAACACUGGCGACGGCUGUUGCGGUGGGUUUGGCAGUAUCGGUGUGGACACUGUGGU (((.((.(((((..((((((((..(((((......)))))((..((((..((((....))))...))))..))).))))))).))))).)).)))(((((.((......)).)))))... ( -49.70) >DroWil_CAF1 142952 120 - 1 CAAUCCCACUGCCUGCAACUGUUGCAGCAAUUGAAUGGAGGCGGGUGUUAAGGCAUCUGCUCCCAAAACUGGCGACGGUUGUUGCGGUGGGUUUGGCAAUAUAGACGUGGAAACACUUGU (((.((((((((..(((((((((((..((.((...(((..((((((((....))))))))..))).)).))))))))))))).)))))))).)))...........(((....))).... ( -56.00) >DroAna_CAF1 110201 120 - 1 CAACCCAACCGCCUGCAACUGCUGCAACAGUUGGAUGGAGGCCGGGGUUAUGGCAUCGGCUCCUAGGACUGGUGACGGCUGCUGCGGCGGAUUGGGCAGUAUCGGUGUGGCCACACUGGU ...((((((((((.(((.(.((((((.(((((...(((..((((..((....))..))))..))).))))).)).)))).).)))))))).)))))....((((((((....)))))))) ( -56.20) >DroPer_CAF1 189764 120 - 1 CAGGCCCACGGCCUGCAACUGUUGCAGCAAUUGUAUGGAGGCCGGUGUCAGUGCAUCGGCGCCCAGAACGGGCGACUGCUGCUGCGGUGGGUUCGGCAGUAUCGGUGUGGACACACUCUG ((((....((((((.((((.((((...)))).)).)).))))))(((((...((((((((((((.....)))))(((((((..((.....)).))))))).))))))).)))))..)))) ( -52.80) >consensus CAGUCCCACCGCCUGCAACUGCUGCAGCAAUUGAAUGGAGGCCGGUGUCAGGGCAUCGGCGCCCAGAACUGGCGACGGCUGCUGCGGUGGGUUCGGCAGUAUCGGUGUGGACACACUGGU ..........(((.....(((((((((((......(((..((((((((....))))))))..)))...(((....))).)))))))))))....)))......(((((....)))))... (-36.52 = -36.33 + -0.19)
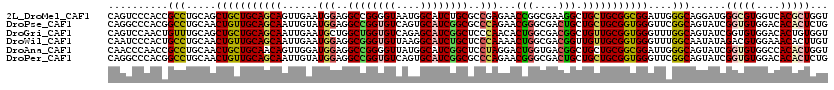
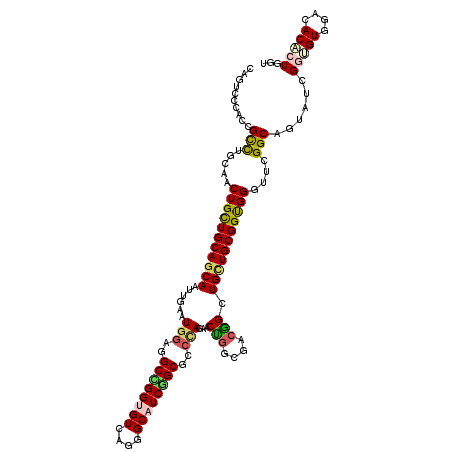
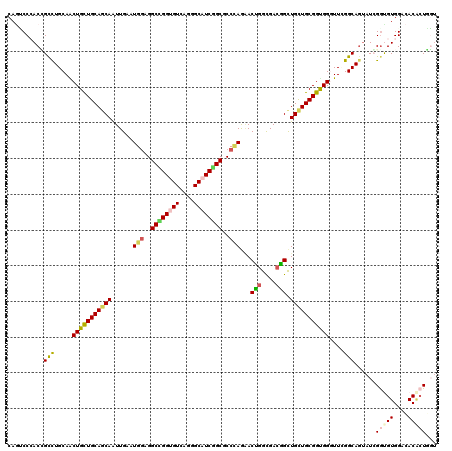
| Location | 12,299,012 – 12,299,116 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.23 |
| Mean single sequence MFE | -41.87 |
| Consensus MFE | -31.09 |
| Energy contribution | -29.32 |
| Covariance contribution | -1.77 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.33 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.79 |
| SVM RNA-class probability | 0.851948 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
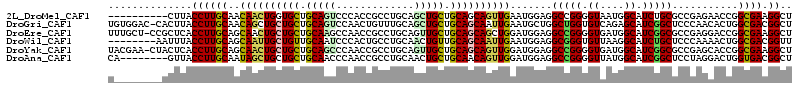
>2L_DroMel_CAF1 12299012 104 + 22407834 AGCCUUCGCCGGUUCUCGGCGCAGAUGCCAUUACCCCGGCCUCCAUUCAACUGCUGCAGCAGCUGCAGGCGGUGGGACUGCAGCACCAGUUGUUGCAAGGUAAG---------- .(((((.((((.....))))(((((.(((........))).))....(((((((((((((.((((....))))..).)))))))...))))).))))))))...---------- ( -40.30) >DroGri_CAF1 154848 113 + 1 AGCCGUCGCCAGUGUUGGGAGCCGAUGCUCUGACACCAGCCAGCAUUCAAUUGCUGCAGCAGCUGCAAACAGUUGGACUGCAGCAGCAGCUGUUGCAAGGUAAGUG-GUCCACA ....((.((((((((..(.(((....))))..))))..(((((((......))))(((((((((((...(((.....))).....)))))))))))..)))...))-).).)). ( -44.40) >DroEre_CAF1 119766 113 + 1 AGCCUUCGCCGGUCCUCGGCGCCGAUGCCAUCACCCCGGCCUCCAUCCAGCUGCUGCAGCAACUGCAGGCGGUUGGCUUGCAGCAGCAGUUGCUGCAAGGUGAGCGG-AGCAAA .......(((((.....((((....))))......)))))((((...((((((((((((...))))).)))))))(((..(.(((((....)))))...)..)))))-)).... ( -51.60) >DroWil_CAF1 142992 106 + 1 AACCGUCGCCAGUUUUGGGAGCAGAUGCCUUAACACCCGCCUCCAUUCAAUUGCUGCAACAGUUGCAGGCAGUGGGAUUGCAACAGCAAUUGCUGCAAGGUAAAUU-------- .(((.....(((((.(((..((.(.((......)).).))..)))...)))))(((((.....)))))(((((..((((((....)))))))))))..))).....-------- ( -30.40) >DroYak_CAF1 126103 113 + 1 AGCCUUCGCCGGUGCUCGGCGCCGAUGCCAUCACCCCGGCCUCCAUCCAACUGCUGCAGCAACUGCAGGCGGUUGGGCUGCAGCAGCAGUUGCUGCAAGGUGAGUAG-UUCGUA ....(((((((((((...)))))((.(((........))).))..((((((((((((((...))))).))))))))).((((((((...)))))))).))))))...-...... ( -48.30) >DroAna_CAF1 110241 106 + 1 AGCCGUCACCAGUCCUAGGAGCCGAUGCCAUAACCCCGGCCUCCAUCCAACUGUUGCAGCAGUUGCAGGCGGUUGGGUUGCAGCAGCAGCUAUUGCAAGGUAAC--------UG (((((((..........(((((((............))).))))...(((((((....)))))))..))))))).((((((.((((......))))...)))))--------). ( -36.20) >consensus AGCCGUCGCCAGUCCUCGGAGCCGAUGCCAUAACCCCGGCCUCCAUCCAACUGCUGCAGCAGCUGCAGGCGGUUGGACUGCAGCAGCAGUUGCUGCAAGGUAAGU________A .(((...((.(((....((....((.(((........))).))...)).)))))((((((((((((..((((.....))))....)))))))))))).)))............. (-31.09 = -29.32 + -1.77)


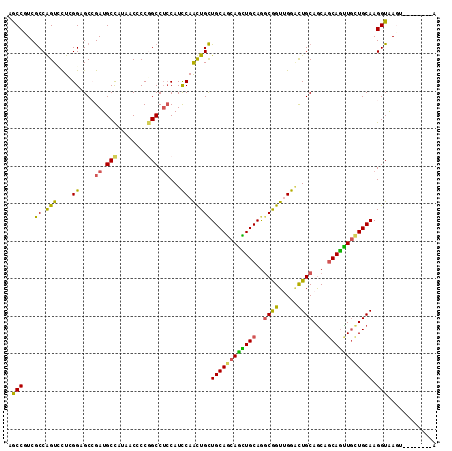
| Location | 12,299,012 – 12,299,116 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.23 |
| Mean single sequence MFE | -44.47 |
| Consensus MFE | -30.34 |
| Energy contribution | -30.24 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921559 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 12299012 104 - 22407834 ----------CUUACCUUGCAACAACUGGUGCUGCAGUCCCACCGCCUGCAGCUGCUGCAGCAGUUGAAUGGAGGCCGGGGUAAUGGCAUCUGCGCCGAGAACCGGCGAAGGCU ----------....((((((..((((((.(((.(((((..((.....))..))))).))).))))))...(((.((((......)))).)))))((((.....)))).)))).. ( -42.30) >DroGri_CAF1 154848 113 - 1 UGUGGAC-CACUUACCUUGCAACAGCUGCUGCUGCAGUCCAACUGUUUGCAGCUGCUGCAGCAAUUGAAUGCUGGCUGGUGUCAGAGCAUCGGCUCCCAACACUGGCGACGGCU .......-........((((..((((.(((((.((((.....))))..))))).))))..))))......((((((..((((..((((....))))...))))..))..)))). ( -44.80) >DroEre_CAF1 119766 113 - 1 UUUGCU-CCGCUCACCUUGCAGCAACUGCUGCUGCAAGCCAACCGCCUGCAGUUGCUGCAGCAGCUGGAUGGAGGCCGGGGUGAUGGCAUCGGCGCCGAGGACCGGCGAAGGCU .(..((-(((..(..(((((((((.....)))))))))(((.((((((((((...))))))..)).)).))).)..)))))..).(((.....(((((.....)))))...))) ( -50.80) >DroWil_CAF1 142992 106 - 1 --------AAUUUACCUUGCAGCAAUUGCUGUUGCAAUCCCACUGCCUGCAACUGUUGCAGCAAUUGAAUGGAGGCGGGUGUUAAGGCAUCUGCUCCCAAAACUGGCGACGGUU --------.....(((((((..((((((((((.(((....((.....))....))).))))))))))..(((..((((((((....))))))))..)))......)))).))). ( -38.90) >DroYak_CAF1 126103 113 - 1 UACGAA-CUACUCACCUUGCAGCAACUGCUGCUGCAGCCCAACCGCCUGCAGUUGCUGCAGCAGUUGGAUGGAGGCCGGGGUGAUGGCAUCGGCGCCGAGCACCGGCGAAGGCU ......-...(((...((((((((.....)))))))).(((((.((.(((((...))))))).)))))...)))(((((.((..((((......)))).)).)))))....... ( -47.30) >DroAna_CAF1 110241 106 - 1 CA--------GUUACCUUGCAAUAGCUGCUGCUGCAACCCAACCGCCUGCAACUGCUGCAACAGUUGGAUGGAGGCCGGGGUUAUGGCAUCGGCUCCUAGGACUGGUGACGGCU ..--------((((((...(...(((((.((((..(((((..((.(((.((((((......)))))).).)).))...)))))..)))).))))).....)...)))))).... ( -42.70) >consensus U________ACUUACCUUGCAACAACUGCUGCUGCAAUCCAACCGCCUGCAGCUGCUGCAGCAGUUGAAUGGAGGCCGGGGUGAUGGCAUCGGCGCCGAGAACCGGCGAAGGCU ..............((((((..((((((((((.(((((.............))))).)))))))))).......(((((.((....)).)))))...........)))).)).. (-30.34 = -30.24 + -0.11)

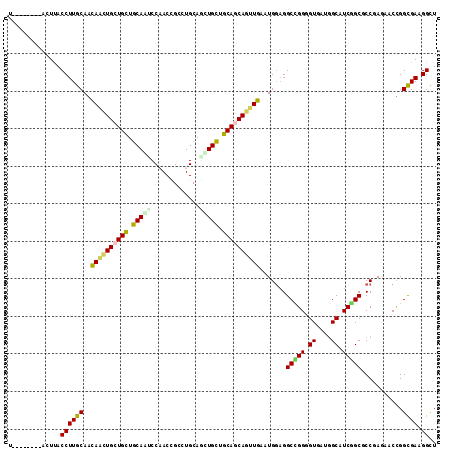
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:32 2006