
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,291,366 – 12,291,460 |
| Length | 94 |
| Max. P | 0.864835 |

| Location | 12,291,366 – 12,291,460 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 81.48 |
| Mean single sequence MFE | -35.12 |
| Consensus MFE | -19.95 |
| Energy contribution | -21.23 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.807998 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
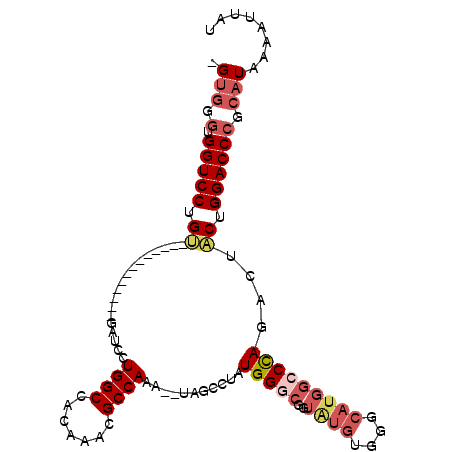
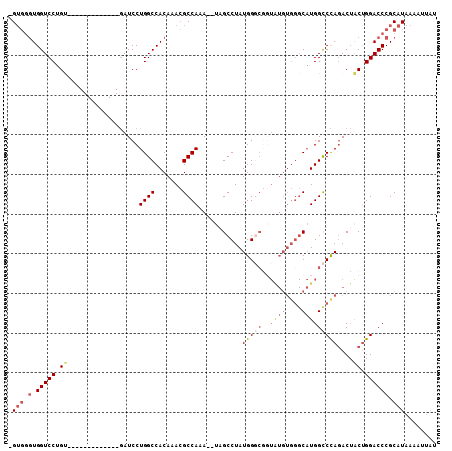
>2L_DroMel_CAF1 12291366 94 + 22407834 -GUGGGUGGUCCUGU-------------GAUCCUGGCCACAAACGCCAAAUAUAGCCUAUGGGCGUUAUGUGGGCAUGGCCCAGACUACAGGACCCGCAUAAAAUUAU -(((.(.((((((((-------------(.((..(((((.....(((..(((..(((....)))..)))...))).)))))..)).)))))))))).)))........ ( -39.80) >DroSec_CAF1 114128 93 + 1 GGUGGGUGGUCCUGU-------------GAUCCUGGCCACAAACGCCAAA--UAGCCUAUGGGCGGUAUGUGGGCAUGGCCCAGACUACUGGACCCGCAUAAAAUUAU .(((.(.(((((.((-------------(.((..(((((.....(((..(--(((((....)))..)))...))).)))))..)).))).)))))).)))........ ( -34.10) >DroSim_CAF1 117896 90 + 1 GGUGGGUGGUCCUGU-------------GAUCCUGGCCACAAACGCCAAA--UAGCCUAUGG---GUAUGUGGGCAUGGUCCAGACUACUGGACCCGCAUAAAAUUAU ((((.((((((..(.-------------...)..))))))...))))...--..((((((..---....))))))..(((((((....)))))))............. ( -34.60) >DroEre_CAF1 112420 92 + 1 -GUGGGUGGUCCUGC-------------GAUCCUGGCCACAGACGCCACA--UCGCCUAUGGGCGGUGUGUGGGCAUGGCCUAGACUGCUGGACCCGCAUAAAAUUAU -(((.(.(((((.((-------------(.((..(((((....(((.(((--(((((....)))))))))))....)))))..)).))).)))))).)))........ ( -45.80) >DroYak_CAF1 118468 92 + 1 -GUGGGUGGUCCUGU-------------GAUCCUGGCCACAAACGCCAAA--UAGCCUAUGGGCGGUAUGUGGGCAUGGCCCAGACUACUGGACCCGCAUAAAAUGAU -(((.(.(((((.((-------------(.((..(((((.....(((..(--(((((....)))..)))...))).)))))..)).))).)))))).)))........ ( -33.70) >DroPer_CAF1 179905 94 + 1 -GGUCCUGGUCCUGCAUUCCGCCUCCUGAAUCCUGGCCACAAAAGCCACA--CUGCCUAUGGCC----------CCUGGCCUACAC-ACUGGACCCGCAUAAAAUUCU -((((((((((..(.((((........)))).).))))).....((((..--..(((...))).----------..))))......-...)))))............. ( -22.70) >consensus _GUGGGUGGUCCUGU_____________GAUCCUGGCCACAAACGCCAAA__UAGCCUAUGGGCGGUAUGUGGGCAUGGCCCAGACUACUGGACCCGCAUAAAAUUAU .(((.(.(((((.((..................((((.......))))...........(((((..((((....)))))))))....)).)))))).)))........ (-19.95 = -21.23 + 1.28)

| Location | 12,291,366 – 12,291,460 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 81.48 |
| Mean single sequence MFE | -34.83 |
| Consensus MFE | -20.97 |
| Energy contribution | -21.50 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.864835 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
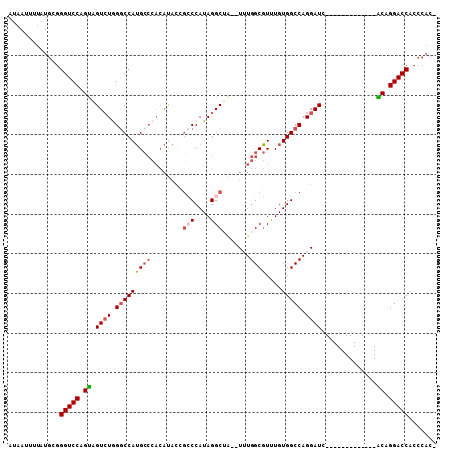
>2L_DroMel_CAF1 12291366 94 - 22407834 AUAAUUUUAUGCGGGUCCUGUAGUCUGGGCCAUGCCCACAUAACGCCCAUAGGCUAUAUUUGGCGUUUGUGGCCAGGAUC-------------ACAGGACCACCCAC- .............((((((((.((((.(((((((((((.(((..(((....)))..))).))).))..)))))).)))).-------------))))))))......- ( -39.60) >DroSec_CAF1 114128 93 - 1 AUAAUUUUAUGCGGGUCCAGUAGUCUGGGCCAUGCCCACAUACCGCCCAUAGGCUA--UUUGGCGUUUGUGGCCAGGAUC-------------ACAGGACCACCCACC .............(((((.((.((((.(((((((((((.(((..(((....)))))--).))).))..)))))).)))).-------------)).)))))....... ( -33.70) >DroSim_CAF1 117896 90 - 1 AUAAUUUUAUGCGGGUCCAGUAGUCUGGACCAUGCCCACAUAC---CCAUAGGCUA--UUUGGCGUUUGUGGCCAGGAUC-------------ACAGGACCACCCACC .............(((((.((..(((((.(((((((((.((((---(....)).))--).))).))..)))))))))...-------------)).)))))....... ( -28.20) >DroEre_CAF1 112420 92 - 1 AUAAUUUUAUGCGGGUCCAGCAGUCUAGGCCAUGCCCACACACCGCCCAUAGGCGA--UGUGGCGUCUGUGGCCAGGAUC-------------GCAGGACCACCCAC- .............(((((.((.((((.(((((((((((((...((((....)))).--))))).))..)))))).)))).-------------)).)))))......- ( -41.40) >DroYak_CAF1 118468 92 - 1 AUCAUUUUAUGCGGGUCCAGUAGUCUGGGCCAUGCCCACAUACCGCCCAUAGGCUA--UUUGGCGUUUGUGGCCAGGAUC-------------ACAGGACCACCCAC- .............(((((.((.((((.(((((((((((.(((..(((....)))))--).))).))..)))))).)))).-------------)).)))))......- ( -33.70) >DroPer_CAF1 179905 94 - 1 AGAAUUUUAUGCGGGUCCAGU-GUGUAGGCCAGG----------GGCCAUAGGCAG--UGUGGCUUUUGUGGCCAGGAUUCAGGAGGCGGAAUGCAGGACCAGGACC- .............(((((...-.....(((((((----------(((((((.....--)))))))))..))))).(.((((........)))).).)))))......- ( -32.40) >consensus AUAAUUUUAUGCGGGUCCAGUAGUCUGGGCCAUGCCCACAUACCGCCCAUAGGCUA__UUUGGCGUUUGUGGCCAGGAUC_____________ACAGGACCACCCAC_ .............(((((.((.((((.(((((((((........(((....))).......))))....))))).))))..............)).)))))....... (-20.97 = -21.50 + 0.53)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:21 2006