
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 12,052,580 – 12,052,695 |
| Length | 115 |
| Max. P | 0.577150 |

| Location | 12,052,580 – 12,052,695 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.66 |
| Mean single sequence MFE | -33.45 |
| Consensus MFE | -24.22 |
| Energy contribution | -24.83 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.12 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.577150 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
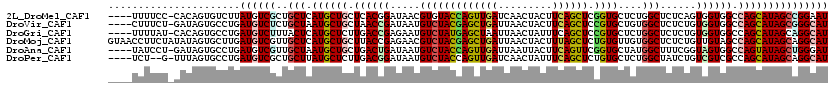
>2L_DroMel_CAF1 12052580 115 - 22407834 ----UUUUCC-CACAGUGUCUUAUGUCGCUGCUCAUGCUGCUCACGGAUAACGUGUACCAGUUGAUCAACUACUUCAGCUCGGUGCUCUGGCUCUCAGUGGUGGCCAGCAUAGCCGGAAU ----..((((-...((((.(....).))))(((.((((((..(((.........(((((((((((.........)))))).))))).(((.....)))..)))..))))))))).)))). ( -36.90) >DroVir_CAF1 9819 115 - 1 ----CUUUCU-GAUAGUGCCUGAUGUCUCUGCUAAUGCUGCUAACCGAUAAUGUCUACGAGCUGAUUAACUACUUCAGCUCCGUGCUGUGGCUCUCUGUGGUGGCCAGCAUAGCGGGCAU ----......-....((((((((....)).((((.((((((((.(((.....(((((((((((((.........)))))).))))....)))......)))))).))))))))))))))) ( -36.50) >DroGri_CAF1 10190 115 - 1 ----UUUUAU-CACAGUGCCUGAUGUCUUUACUCAUGCUCUUGACCGAGAAUGUCUAUGAGCUAAUUAACUAUUUCAGCUCCGUGCUCUGGCUCUCUGUGGUGGCCAGCAUAGCAGGCAU ----...(((-(((((.(((.((.............((((..(((.......)))...))))...............((.....)))).)))...))))))))(((.((...)).))).. ( -29.80) >DroMoj_CAF1 9385 120 - 1 GUAACCUUCUAUAUAGUGCUUGAUGUCGUUGCUCAUGCUGCUUACCGAGAACGUCUACGAGCUGAUUAACUACUUUAGCUCUGUGUUGUGGCUCUCUGUUGUAGCCAGCAUAGCAGGCAU ...............(((((((.(((.((((.....(((((..((.((((..(((((((((((((.........))))))).)))....))))))).)).)))))))))))).))))))) ( -33.40) >DroAna_CAF1 7011 115 - 1 ----UAUCCU-GAUAGUGCCUGAUGUCGUUGCUAAUGCUGCUGACUGAUAAUGUCUACCAGUUGAUUAAUUACUUCAGUUCGGUGCUAUGGCUUUCGGUAGUGGCCAGUAUAGCUGGGAU ----.((((.-......(((...(..((..((((..((.((((((((((((((((........)))..))))..)))).)))))))..))))...))..)..)))(((.....))))))) ( -30.10) >DroPer_CAF1 6512 113 - 1 ----UCU--G-UUUAGUGCCUGAUGUCGCUGCUUAUGCUCUUGACGGAUAAUGUCUACCAGUUGAUCAACUAUUUCAGCUCUGUGCUCUGGCUAUCUGUCGUCGCCAGCAUAGCAGGCAU ----...--.-....(((((((.(((.((((.....((...(((((((((..((((((.((((((.........))))))..)))....))))))))))))..))))))))).))))))) ( -34.00) >consensus ____UUUUCU_CAUAGUGCCUGAUGUCGCUGCUCAUGCUGCUGACCGAUAAUGUCUACCAGCUGAUUAACUACUUCAGCUCCGUGCUCUGGCUCUCUGUGGUGGCCAGCAUAGCAGGCAU ......................((((((..(((.((((((.((((((.....(((((((((((((.........)))))).))))....)))......)))))).))))))))))))))) (-24.22 = -24.83 + 0.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:12:28 2006