
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 11,797,817 – 11,798,016 |
| Length | 199 |
| Max. P | 0.999951 |

| Location | 11,797,817 – 11,797,936 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.54 |
| Mean single sequence MFE | -44.97 |
| Consensus MFE | -41.41 |
| Energy contribution | -41.72 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972780 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 11797817 119 + 22407834 -UGCCGACUCACUUAUGAUCGGUUUCACUUCCUGCUUGACUUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCU -.(((((.(((....)))))))).((((((..(((((((..(((....((.(((((.(((((((((((....)))))))).))).))))))).)))))).))))...))))))....... ( -46.80) >DroGri_CAF1 6011 120 + 1 UGUCCGACUCACUUAUGAUCGGUUUCACUUCCUGCUUGACUUGGGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCU ...((((.(((....)))))))..((((((..(((((((.....(((.((.(((((.(((((((((((....)))))))).))).)))))))))).))).))))...))))))....... ( -44.70) >DroSec_CAF1 5222 119 + 1 -UGCCGACUCACUUAUGAUCGGUUUCACUUCCUGCUUGACUUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGGCCGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUUAUCU -.(((((.(((....)))))))).((((((..(((((((..(((....((.(((((.(((((((((((....)))))).))))).))))))).)))))).))))...))))))....... ( -46.80) >DroWil_CAF1 8927 119 + 1 -UGCCGACUCACUUAUAAUCGGUUUCACUUCCUGCUUGACUUGUGUCAGCACACCGGCUGGUAUUUGCGGAUGCGGAUGCACUGGUGGUGGCGGCAUCAUGGCAUCCGGAUGGUUCAUCU -(((((.(.(((........((........))((((.(((....)))))))((((((...((((((((....)))))))).)))))))))))))))...........(((((...))))) ( -38.40) >DroMoj_CAF1 6120 120 + 1 UGUCCGACUCACUUAUGAUCGGUUUCACUUCCUGCUUGACUUGGGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCU ...((((.(((....)))))))..((((((..(((((((.....(((.((.(((((.(((((((((((....)))))))).))).)))))))))).))).))))...))))))....... ( -44.70) >DroAna_CAF1 9764 119 + 1 -CGCCGACUCACUUAUGAUCGGUUUCACCUCCUGCUUGACUUGCGUCAGCACACCAGCCGGUAUUUGUGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCU -.(((((.(((....)))))))).((((((..(((((((..(((....((.(((((.(((((((((((....)))))))).))).))))))).)))))).))))...))))))....... ( -48.40) >consensus _UGCCGACUCACUUAUGAUCGGUUUCACUUCCUGCUUGACUUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCU ..(((((.(((....)))))))).((((((..((((((((....))))((.(((((.(((((((((((....)))))))).))).)))))))........))))...))))))....... (-41.41 = -41.72 + 0.31)

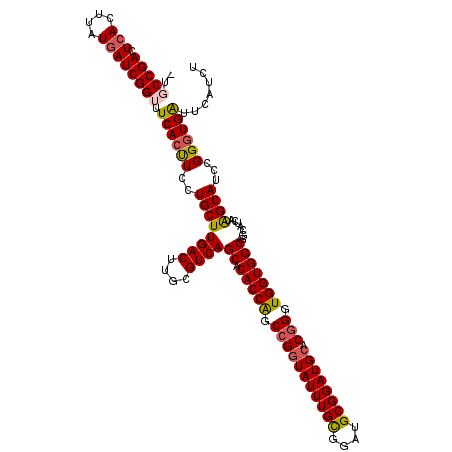

| Location | 11,797,817 – 11,797,936 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.54 |
| Mean single sequence MFE | -39.90 |
| Consensus MFE | -36.96 |
| Energy contribution | -37.18 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979417 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 11797817 119 - 22407834 AGAUGAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGUGCAUCCGCAUCCGCAAAUACCGGCUGGUGUGCUGACGCAAGUCAAGCAGGAAGUGAAACCGAUCAUAAGUGAGUCGGCA- .......((((((((((((...(((((((........)).))))).))))))((..(((((....)))))..((((....)))).)).))..))))..(((((((....))).))))..- ( -41.80) >DroGri_CAF1 6011 120 - 1 AGAUGAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGUGCAUCCGCAUCCGCAAAUACCGGCUGGUGUGCUGACCCAAGUCAAGCAGGAAGUGAAACCGAUCAUAAGUGAGUCGGACA .......((((((((((((...(((((((........)).))))).))))))((..(((((....)))))..((((....)))).)).))..))))..(((((((....))).))))... ( -38.30) >DroSec_CAF1 5222 119 - 1 AGAUAAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGGCCAUCCGCAUCCGCAAAUACCGGCUGGUGUGCUGACGCAAGUCAAGCAGGAAGUGAAACCGAUCAUAAGUGAGUCGGCA- .......((((((((((((...(((((((........))).)))).))))))((..(((((....)))))..((((....)))).)).))..))))..(((((((....))).))))..- ( -41.80) >DroWil_CAF1 8927 119 - 1 AGAUGAACCAUCCGGAUGCCAUGAUGCCGCCACCACCAGUGCAUCCGCAUCCGCAAAUACCAGCCGGUGUGCUGACACAAGUCAAGCAGGAAGUGAAACCGAUUAUAAGUGAGUCGGCA- ..........(((((((((...(((((.((........))))))).))))))((..(((((....)))))..((((....)))).)).))).......((((((.......))))))..- ( -35.40) >DroMoj_CAF1 6120 120 - 1 AGAUGAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGUGCAUCCGCAUCCGCAAAUACCGGCUGGUGUGCUGACCCAAGUCAAGCAGGAAGUGAAACCGAUCAUAAGUGAGUCGGACA .......((((((((((((...(((((((........)).))))).))))))((..(((((....)))))..((((....)))).)).))..))))..(((((((....))).))))... ( -38.30) >DroAna_CAF1 9764 119 - 1 AGAUGAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGUGCAUCCGCAUCCACAAAUACCGGCUGGUGUGCUGACGCAAGUCAAGCAGGAGGUGAAACCGAUCAUAAGUGAGUCGGCG- .......((((((((((((...(((((((........)).))))).))))))....(((((....)))))((((((....))).)))..).)))))..(((((((....))).))))..- ( -43.80) >consensus AGAUGAAUCACCCGGAUGCUAUGAUGCCGCCACCACCCGUGCAUCCGCAUCCGCAAAUACCGGCUGGUGUGCUGACGCAAGUCAAGCAGGAAGUGAAACCGAUCAUAAGUGAGUCGGCA_ .......((((((((((((...(((((((........)).))))).))))))....(((((....)))))((((((....))).))).))..))))..(((((((....))).))))... (-36.96 = -37.18 + 0.22)


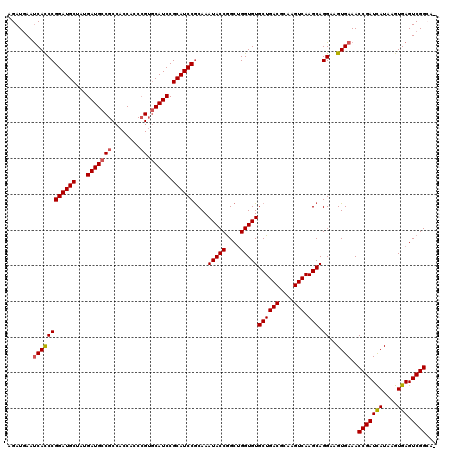
| Location | 11,797,856 – 11,797,976 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.61 |
| Mean single sequence MFE | -53.72 |
| Consensus MFE | -51.04 |
| Energy contribution | -50.85 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.95 |
| Structure conservation index | 0.95 |
| SVM decision value | 4.80 |
| SVM RNA-class probability | 0.999951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 11797856 120 + 22407834 UUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUU ...(((((((.(((((.(((((((((((....)))))))).))).)))))((((((....(((..(((.((((.......)))).))).)))......))))))))).))))........ ( -56.20) >DroVir_CAF1 5460 120 + 1 UUGGGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAAAGCCUU ....((((((.(((((.(((((((((((....)))))))).))).)))))((((((....(((..(((.((((.......)))).))).)))......))))))))).)))......... ( -54.20) >DroSec_CAF1 5261 120 + 1 UUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGGCCGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUUAUCUUCAUGUGGGGCUUCAAAUUGCCGCGCUCGACGAACGCCUU ...(((((((.(((((.(((((((((((....)))))).))))).)))))((((((....(((.((((.((((.......)))).)))))))......))))))))).))))........ ( -58.20) >DroWil_CAF1 8966 120 + 1 UUGUGUCAGCACACCGGCUGGUAUUUGCGGAUGCGGAUGCACUGGUGGUGGCGGCAUCAUGGCAUCCGGAUGGUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCAACGAACGCCUU ((((...(((.((((((...((((((((....)))))))).))))))...((((((....(((..(((.((((.......)))).))).)))......)))))))))..))))....... ( -44.60) >DroMoj_CAF1 6160 120 + 1 UUGGGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAAAGCCUU ....((((((.(((((.(((((((((((....)))))))).))).)))))((((((....(((..(((.((((.......)))).))).)))......))))))))).)))......... ( -54.20) >DroAna_CAF1 9803 120 + 1 UUGCGUCAGCACACCAGCCGGUAUUUGUGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUU ...(((((((.(((((.(((((((((((....)))))))).))).)))))((((((....(((..(((.((((.......)))).))).)))......))))))))).))))........ ( -54.90) >consensus UUGCGUCAGCACACCAGCCGGUAUUUGCGGAUGCGGAUGCACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUU ....((((((.(((((.(((((((((((....)))))))).))).)))))((((((....(((..(((.((((.......)))).))).)))......))))))))).)))......... (-51.04 = -50.85 + -0.19)

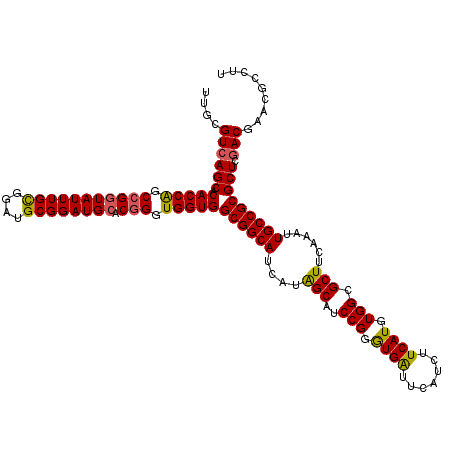
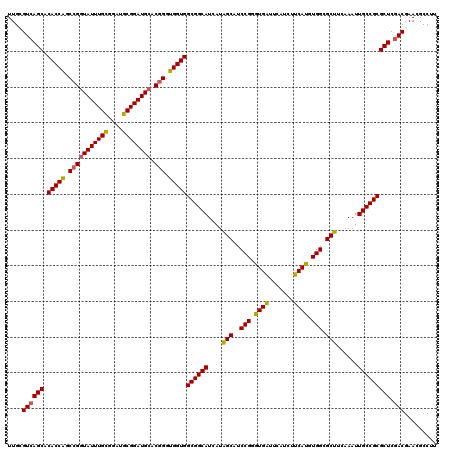
| Location | 11,797,896 – 11,798,016 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.39 |
| Mean single sequence MFE | -45.87 |
| Consensus MFE | -45.64 |
| Energy contribution | -45.03 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.14 |
| Mean z-score | -0.73 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.732374 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 11797896 120 + 22407834 ACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUUGCUGCACAGUGUGCACGCGUGUGGUUUCUCACCCGAAUGG .(((((((..((((((....(((..(((.((((.......)))).))).)))......)))))).......(((.(((.((((((((...))))).)))...)))))))))))))..... ( -46.90) >DroPse_CAF1 9524 120 + 1 ACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAAAGCCUUGCUGCACAGAGUGCACGCGUGUGGUUUCUCGCCCGAAUGG .(((((((..((((((....(((..(((.((((.......)))).))).)))......)))))).......((((.((.((((((((...))))).)))...)))))))))))))..... ( -46.50) >DroSec_CAF1 5301 120 + 1 CCGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUUAUCUUCAUGUGGGGCUUCAAAUUGCCGCGCUCGACGAACGCCUUGCUGCACUGUGUGCACGCGUGUGGUUUCUCACCCGAAUGG .(((((((..((((((....(((.((((.((((.......)))).)))))))......)))))).......(((.(((.((((((((...))))).)))...)))))))))))))..... ( -48.70) >DroWil_CAF1 9006 120 + 1 ACUGGUGGUGGCGGCAUCAUGGCAUCCGGAUGGUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCAACGAACGCCUUGCUGCACAGUGUGCACGCAUGCGGUUUCUCGCCCGAAUGG .(.(((((..((((((....(((..(((.((((.......)))).))).)))......))))))..........(((..((((((((...))))).))).))).....))))).)..... ( -40.00) >DroAna_CAF1 9843 120 + 1 ACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUUGCUGCACAGAGUGCACGCGUGCGGUUUCUCGCCCGAAUGG .(((((((..((((((....(((..(((.((((.......)))).))).)))......))))))..........(((..((((((((...))))).))).))).....)))))))..... ( -46.60) >DroPer_CAF1 9624 120 + 1 ACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAAAGCCUUGCUGCACAGAGUGCACGCGUGUGGUUUCUCGCCCGAAUGG .(((((((..((((((....(((..(((.((((.......)))).))).)))......)))))).......((((.((.((((((((...))))).)))...)))))))))))))..... ( -46.50) >consensus ACGGGUGGUGGCGGCAUCAUAGCAUCCGGGUGAUUCAUCUUCAUGUGGCGCUUCAAAUUGCCGCGCUCGACGAACGCCUUGCUGCACAGAGUGCACGCGUGUGGUUUCUCGCCCGAAUGG .(((((((..((((((....(((..(((.((((.......)))).))).)))......)))))).......(((.(((.((((((((...))))).)))...)))))))))))))..... (-45.64 = -45.03 + -0.61)

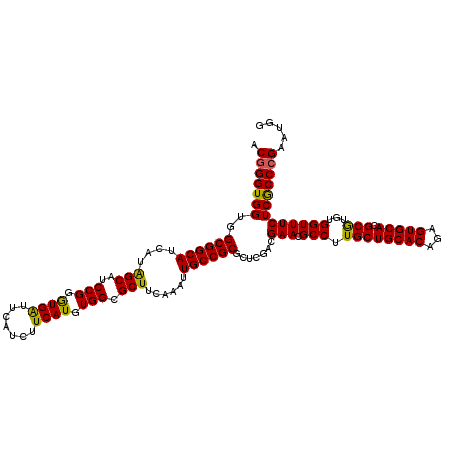
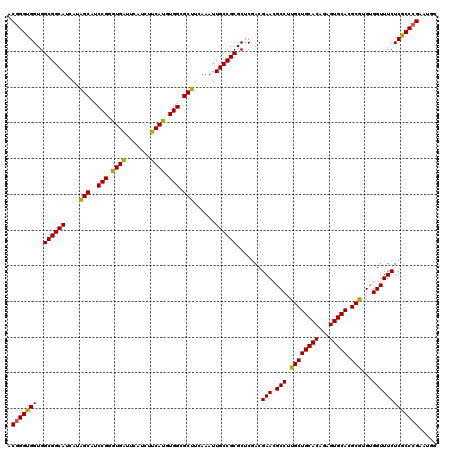
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:09:35 2006