
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 11,386,742 – 11,386,873 |
| Length | 131 |
| Max. P | 0.798861 |

| Location | 11,386,742 – 11,386,834 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.76 |
| Mean single sequence MFE | -26.30 |
| Consensus MFE | -21.10 |
| Energy contribution | -21.13 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.753413 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
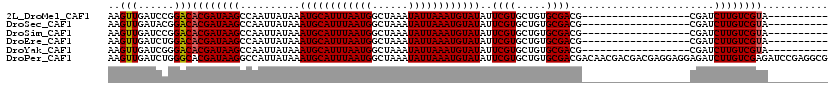
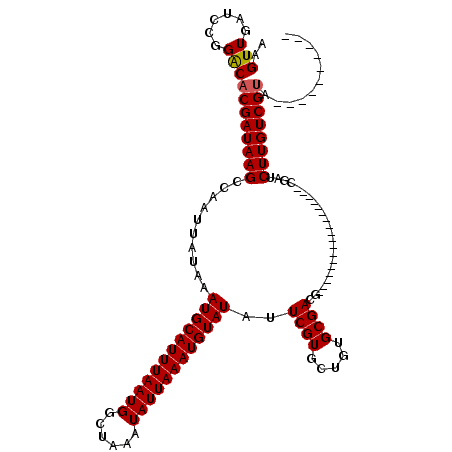
>2L_DroMel_CAF1 11386742 92 - 22407834 AAGUUGAUCCGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG------------------CGAUCUUGUCGUA---------- ..((((...(((.((((((((.....)))...((((((((((((......))))))))))))..))))))))..))))(------------------((((...))))).---------- ( -24.50) >DroSec_CAF1 103733 92 - 1 AAGUUGAUACGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG------------------CGAUCUUGUCGUA---------- ..((((.(((((.((((((((.....)))...((((((((((((......))))))))))))..))))))))))))))(------------------((((...))))).---------- ( -27.60) >DroSim_CAF1 110677 92 - 1 AAGUUGAUCCGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG------------------CGAUCUUGUCGUA---------- ..((((...(((.((((((((.....)))...((((((((((((......))))))))))))..))))))))..))))(------------------((((...))))).---------- ( -24.50) >DroEre_CAF1 107251 92 - 1 AAGUUGAUCUGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG------------------CGAUCUUGUCGUA---------- ..(((......)))((((((((..........((((((((((((......))))))))))))..(((((........))------------------))).)))))))).---------- ( -23.50) >DroYak_CAF1 107095 92 - 1 AAGUUGAUCGGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG------------------CGAUCUUGUCGUA---------- ..(((......)))((((((((..........((((((((((((......))))))))))))..(((((........))------------------))).)))))))).---------- ( -23.50) >DroPer_CAF1 127241 120 - 1 AAGUUGAUCUGGGCACGAUAAGGCCAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACGACAACGACGACGAGGAGGAGAUCUUGUCGAGAUCCGAGGCG ..((((((((.(((((((...((((((((..........))))))))..((((.....))))..)))))))...............((((((((......)))))))))))))...))). ( -34.20) >consensus AAGUUGAUCCGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG__________________CGAUCUUGUCGUA__________ ..(((......)))((((((((..........((((((((((((......))))))))))))..((((.....))))........................))))))))........... (-21.10 = -21.13 + 0.03)
| Location | 11,386,755 – 11,386,873 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 91.74 |
| Mean single sequence MFE | -30.08 |
| Consensus MFE | -24.16 |
| Energy contribution | -25.35 |
| Covariance contribution | 1.19 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.64 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.798861 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
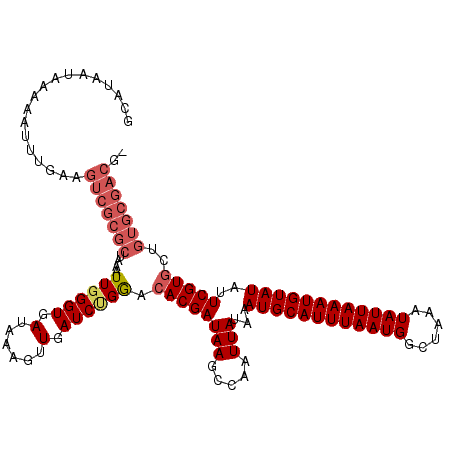
>2L_DroMel_CAF1 11386755 118 - 22407834 GCAUAAUAAUAAUUCGAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCCGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG- ..................(((((((......((((.(......).))))((.((((((((.....)))...((((((((((((......))))))))))))..)))))))))))))).- ( -31.80) >DroPse_CAF1 128186 119 - 1 GCAUAAUAAAAAUUUGAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCUGGGCACGAUAAGGCCAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACGA ..................(((((((....(..(((.(......).)))..)((((((...((((((((..........))))))))..((((.....))))..)))))).))))))).. ( -34.10) >DroEre_CAF1 107264 118 - 1 ACAUAAUAGGAAUUUGAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCUGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG- ..................(((((((....(..(((.(......).)))..).((((((((.....)))...((((((((((((......))))))))))))..)))))..))))))).- ( -29.60) >DroYak_CAF1 107108 118 - 1 ACAUAAUAAAAAUUUAAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCGGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG- ..................(((((((..((((((.(..........((((.....)))).).))))))....((((((((((((......)))))))))))).........))))))).- ( -30.40) >DroMoj_CAF1 139369 109 - 1 GCAUAAUAAAAGUUUGAAGUUGCGCUAAAUUGGGUGAUAAAGUUGAUCCGAGCACGAUAAGCCCAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUCUUCGUUC---------- ...............(((((((.(((....(((((.(......).)))))))).)))..............((((((((((((......))))))))))))))))....---------- ( -20.50) >DroPer_CAF1 127281 119 - 1 GCAUAAUAAAAAUUUGAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCUGGGCACGAUAAGGCCAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACGA ..................(((((((....(..(((.(......).)))..)((((((...((((((((..........))))))))..((((.....))))..)))))).))))))).. ( -34.10) >consensus GCAUAAUAAAAAUUUGAAGUCGCGCUAAAUUGGGUGAUAAAGUUGAUCUGGACACGAUAAGCCAAUUAUAAAUGCAUUUAAUGGCUAAAUAUUAAAUGUAUAUUCGUGCUGUGCGACG_ ..................(((((((....((((((.(......).)))))).((((((((.....)))...((((((((((((......))))))))))))..)))))..))))))).. (-24.16 = -25.35 + 1.19)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:03:53 2006