
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 11,256,321 – 11,256,434 |
| Length | 113 |
| Max. P | 0.586312 |

| Location | 11,256,321 – 11,256,434 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.36 |
| Mean single sequence MFE | -49.48 |
| Consensus MFE | -32.03 |
| Energy contribution | -31.98 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.586312 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
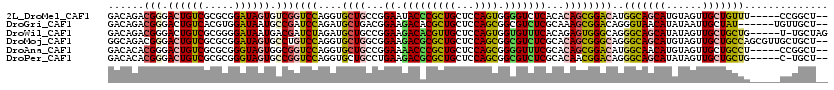
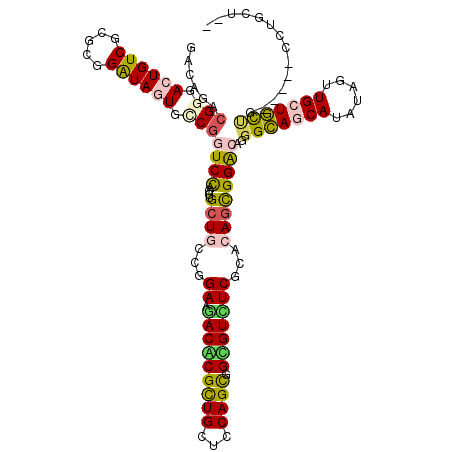
>2L_DroMel_CAF1 11256321 113 - 22407834 GACAGACGGGACUGUCGCGCGGAUAGUGUCGGUCCAGGUGCUGCCGGAAUACCCGCUGCUCCAGUGGGGUCUCACACAGCGGACAUGGCAGCAUGUAGUUGCUGUUU-----CCGGCU-- ...((((.((((((.(((.......))).))))))..)).))(((((((..(((((((...))))))).......((((((.(((((....)))))...))))))))-----))))).-- ( -49.70) >DroGri_CAF1 118469 112 - 1 GACAGACGGGACUGUCACGUGGAUAAUGCCGAUCCAGAUGCUGACGGAAGACACGCUGCUCCAGCGGCGUCUCGCAAAGCGGACAGGGUAACAUAUAAUUGCUAU------UGUUGCU-- ...........(((((.(((((((.......))))...(((...(....)..(((((((....)))))))...)))..))))))))(((((((............------)))))))-- ( -34.30) >DroWil_CAF1 122298 114 - 1 GACAGACGGGACUGUCGCGGGGAUAAUGACGAUCUAGAUGCUGCCGGAAGACACGUUGCUCCAGUGGUGUUUCACAGAGUGGGCAGGGCAGCAUAUAGUUGCUGCUG-----U-UGCUAG (((((......)))))..((.((((.(..(((.(((.(((((((((.....)...((((.(((.(.(((...)))..).))))))))))))))).))))))..).))-----)-).)).. ( -36.60) >DroMoj_CAF1 109932 118 - 1 GGCAGACGGGACUGUCGCGCGGAUAGUGCCUGUCCAGGUGCUGGCGGAAGACGCGCUGCUCCAGCGGCGUCUCGCACAGCGGGCAGGGCAGCAUGUAGUUGCUGCCAGCGUUGCUGCU-- ((((((((...(((((.(((...(((..(((....)))..)))(((..((((((((((...)))).)))))))))...))))))))(((((((......)))))))..)))..)))))-- ( -60.60) >DroAna_CAF1 82472 113 - 1 GACACACGGGACUGUCGCGCGGGUAGUGGCGGUCCAGGUGCUGCCGGAAAACCCGCUGCUCCAGCGGGGUUUCGCACAGCGGACAUGGCAACAUGUAGUUGCUGCCU-----CCGGCU-- ..(((...((((((((((.......))))))))))..)))..((((((...(((((((...))))))).....(((((((..(((((....))))).)))).))).)-----))))).-- ( -61.00) >DroPer_CAF1 113136 112 - 1 GACACACGGGACUGUCGCGCGGGUAGUGCCGGUCCAGGUGCUGCCUGAAGACGCGCUGCUCCAGCGGCGUCUCGCACAACGGACAGGGCAGCAUAUAGUUGCUGCUG-----C-UGCU-- ......((((((.(((...(((((((..((......))..)))))))..)))..(((((....)))))))))))......((.((((((((((......))))))).-----)-))))-- ( -54.70) >consensus GACAGACGGGACUGUCGCGCGGAUAGUGCCGGUCCAGGUGCUGCCGGAAGACACGCUGCUCCAGCGGCGUCUCGCACAGCGGACAGGGCAGCAUAUAGUUGCUGCUG_____CCUGCU__ ......(((.((((((.....)))))).)))((((....((((...((.(((((((((...)))).)))))))...))))))))..(((((((......))))))).............. (-32.03 = -31.98 + -0.05)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:02:49 2006