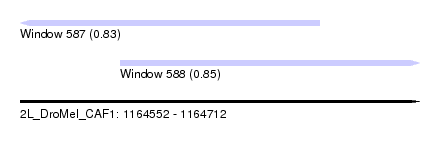
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 1,164,552 – 1,164,712 |
| Length | 160 |
| Max. P | 0.849974 |

| Location | 1,164,552 – 1,164,672 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.61 |
| Mean single sequence MFE | -54.38 |
| Consensus MFE | -44.28 |
| Energy contribution | -44.37 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.828776 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1164552 120 - 22407834 UGCCGGCCGCCGUGAGAUUGUCCAGCAGAUGCUGGAUGCCCUUGGUGGCCAGCGACUGGUAGGUGUGCUCAUUUCUUCCCGGCACCGGGCGCAGCAUCGAUAGCGAUGCAUCCAGCGCUA ....(((((((..(((..((((((((....))))))))..))))))))))((((.((((....(((((..........(((....))))))))((((((....))))))..)))))))). ( -57.00) >DroSec_CAF1 58146 120 - 1 UGCCGGCCGCCAUGAGAUUGUCCAGCAGAUGCUGGAUGCCCUUGGUGGCCAGUGACUGGUAGGUGUGCUCAUUUCUUCCCGGCACCGGGCGCAGCAUCGAUAGCGAUGCAUCCAGCGCUA ....((((((((..((..((((((((....))))))))..))))))))))((((.((((....(((((..........(((....))))))))((((((....))))))..)))))))). ( -55.40) >DroSim_CAF1 58819 120 - 1 UGCCGGCCGCCGUGAGAUUGUCCAGCAGAUGCUGGAUGCCCUUGCUGGCCAGCGACUGGUAGGUGUGCUCAUUUCUUCCCGGCACCGGGCGCAGCAUCGAUAGCGAUGCAUCCAGCGCUA ....(((((..(..((..((((((((....))))))))..))..))))))((((.((((....(((((..........(((....))))))))((((((....))))))..)))))))). ( -53.80) >DroEre_CAF1 51997 120 - 1 UGCCGGCCGCCGUGAAAUUGUCCAGCAGAUGCUGGAUGCCCUUGGUGGCCAGCGACUGGUAGGUGUGCUCGUUUCUUCCCGGAACGGGACGCAGCAUUGAUAGCGAUGCAUCCAGCGCUA ....((((((((.......(((((((....))))))).....))))))))((((.((((...((((.((((((((.....)))))))))))).((((((....))))))..)))))))). ( -59.70) >DroYak_CAF1 53477 120 - 1 UGCCGGCCGCCGUGAGAUUGUCCAGCAGAUGCUGGAUGCCCUUGGUGGCCAACGACUGGUAGGUGUGCUCAUUCCUUCCCGGAACGGGACGCAGCAUCGAUAGCGAUGCAUCCAGCGCUA ....(((((((..(((..((((((((....))))))))..))))))))))..((.((((...((((.(((.((((.....)))).))))))).((((((....))))))..))))))... ( -55.20) >DroAna_CAF1 58269 120 - 1 UUCCGGCGGCUGUGAGAUUGUCCAGCAAGUGCUGGAUUCCCUUGAUGGCCAGCGAUAGGUAGGUGUGAUCAUUUCUUCCUGGAACUGGACGCAGCAUCGACAGCGAUGCAUCCAGGGCUA ((((.(((((((((((...(((((((....)))))))...))).)))))).))...(((.(((.((....)).))).)))))))(((((....((((((....)))))).)))))..... ( -45.20) >consensus UGCCGGCCGCCGUGAGAUUGUCCAGCAGAUGCUGGAUGCCCUUGGUGGCCAGCGACUGGUAGGUGUGCUCAUUUCUUCCCGGAACCGGACGCAGCAUCGAUAGCGAUGCAUCCAGCGCUA ....((((((((.......(((((((....))))))).....))))))))((((.((((...((((.(.....((.....))....).)))).((((((....))))))..)))))))). (-44.28 = -44.37 + 0.09)

| Location | 1,164,592 – 1,164,712 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.94 |
| Mean single sequence MFE | -43.27 |
| Consensus MFE | -36.71 |
| Energy contribution | -37.10 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.849974 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1164592 120 + 22407834 GGGAAGAAAUGAGCACACCUACCAGUCGCUGGCCACCAAGGGCAUCCAGCAUCUGCUGGACAAUCUCACGGCGGCCGGCAAAUUGGAGCACGUUGCUCUGCUCUCCUAUUCGACGACGGC ((((.((...((((((..((.(((((.(((((((.((..(((..((((((....))))))....)))..)).)))))))..)))))))...).)))))...))))))............. ( -46.20) >DroSec_CAF1 58186 120 + 1 GGGAAGAAAUGAGCACACCUACCAGUCACUGGCCACCAAGGGCAUCCAGCAUCUGCUGGACAAUCUCAUGGCGGCCGGCAAAAUGGAGCACGUUGCUCUGCUCUCCUACUCGACGACUGC (((.((....((((..............((((((.(((.(((..((((((....))))))....))).))).))))))......(((((.....)))))))))..)).)))......... ( -44.10) >DroSim_CAF1 58859 120 + 1 GGGAAGAAAUGAGCACACCUACCAGUCGCUGGCCAGCAAGGGCAUCCAGCAUCUGCUGGACAAUCUCACGGCGGCCGGCAAAAUGGAGCACGUUGCUCUGCUCUCCUACUCCACGACGGC .(((......((((.............(((((((.((..(((..((((((....))))))....)))...))))))))).....(((((.....)))))))))......)))........ ( -45.40) >DroEre_CAF1 52037 120 + 1 GGGAAGAAACGAGCACACCUACCAGUCGCUGGCCACCAAGGGCAUCCAGCAUCUGCUGGACAAUUUCACGGCGGCCGGCAAAAUGGAGCACGUUGCUCUGCUUUCCUACUCCACAACGGC ((((((....((((((..((.(((...(((((((.((..(((..((((((....))))))....)))..)).)))))))....)))))...).)))))..)))))).............. ( -44.40) >DroYak_CAF1 53517 120 + 1 GGGAAGGAAUGAGCACACCUACCAGUCGUUGGCCACCAAGGGCAUCCAGCAUCUGCUGGACAAUCUCACGGCGGCCGGCAAAAUGGAGCAUGUUGCUCUGCUUUCCUACUCCACAACGGC (((((((...(((((((.((.(((...(((((((.((..(((..((((((....))))))....)))..)).)))))))....)))))..)).)))))..)))))))............. ( -48.30) >DroAna_CAF1 58309 120 + 1 AGGAAGAAAUGAUCACACCUACCUAUCGCUGGCCAUCAAGGGAAUCCAGCACUUGCUGGACAAUCUCACAGCCGCCGGAAAACUGGAGCACCUUGCUCUGCUUUCGUACACAACCACAGC .(((((......................(((((......((((.((((((....))))))...))))......)))))......(((((.....))))).)))))............... ( -31.20) >consensus GGGAAGAAAUGAGCACACCUACCAGUCGCUGGCCACCAAGGGCAUCCAGCAUCUGCUGGACAAUCUCACGGCGGCCGGCAAAAUGGAGCACGUUGCUCUGCUCUCCUACUCCACGACGGC ((((......((((.............(((((((.((..(((..((((((....))))))....)))..)).))))))).....(((((.....)))))))))))))............. (-36.71 = -37.10 + 0.39)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:34:14 2006